Discussió
En aquest projecte s’han desenvolupat mètodes per la recerca de tres selenoproteïnes en 14 genomes de protists. S’ha iniciat el procés mitjançant el programa BLAST, concretament tblastn, per determinar els genomes en els quals molt probablement es podrien trobar selenorpoteïnes o homòlegs d’aquestes.
S’ha procedit mitjançant l’exonerate i el Genewise a la predicció dels gens que en aquests genomes transcriurien una selenoproteïna i, finalment, s’han alineat les proteïnes resultants amb les queries inicials mitjançant el programa Tcoffee per determinar la qualitat de l'alineament.
Abans de començar a discutir els resultats s'ha de dir que ens ha passat quelcom molt curiós durant el treball i això ha exigit un nivell d'esforç molt superior per part del grup. Inicialment es va usar el software del programa TCoffee amb el que treballa el shell, després de fer l'export, i la gran majoria dels resultats obtinguts per les dues selenoproteïnes que presentaven hits molt significatius varen donar negatius. El grup però no es va conformar amb aquests resultats i amb la intranquil·litat que això implica es varen provar varis alineaments manualment usant el programa TCoffee directament de la plana web. Sorprenentment, es van obtenir resultats significativament diferents i el pànic va corrompre el grup. Gràcies els bons coneixements de l'assignatura es va concloure que calia repetir tots els TCoffee d'aquells organismes pels que s'havia obtingut hits molt significatius i tornar a interpretar els resultats. Aquests, seran els que es comenten seguidament.
1.1 Alineaments significatius
SelO
Els alineaments que resulten del tBLASTn de la query selO humana amb el genomes tant de P.capsici com de S.arctica són molt significatius, tal com es pot veure a la taula dels resultats.
A causa de l'ús inicial del software més antic, de bon principi, tot i tenir hits molt significatius no es va obtenir cap alineament amb cap dels 14 genomes per selO. Degut a això es van buscar dues selO homòlogues de dos protists filogenèticament més propers, amb l'esperança d'obtenir algun resultat. Es van realitzar de nou tots els passos per la recerca de proteïnes homòlogues utilitzant selO de E.siliculosus i selO de P. infestans T30-4.
Abans d'interpretar els resultats, cal tenir en compte una de les dificultats que presenta l'alineament d'aquesta selenoproteïna, i és que tant la selenocisteïna com la cisteïna en les proteïnes homòlogues de les queries es troben al final de la proteïna, concretament en l’antepenúltima posició d’aquesta. Això dificulta la detecció de regions conservades rere el codó TGA (en el cas de Cys) o UGA (per Sec) i, per tant, la identificació de la selenoproteïna i els seus homòlegs.
1.1 SelO de E. siliculosusEls resultats obtinguts per E.siliculosus tampoc varen ser gaire esperançadors. No es va obtenir cap proteïna tot i obtenir:
- dos hits molt significatius en el genoma d'A.laibachii Nc14 (scaffold gi|325192005|emb|FR824421.1|, E-value 1-100 i més d'un 40% d'identitat).
- quatre hits en el de F.cylindrus (dos per l'scaffold_15 i dos per l'scaffold_3, amb valors de E-value compresos entre 9-21 i 4-08).
- un en I.multifiliis strain G5 (contig gi|340500597|gb|GL984358.1|, E-value 1e-74 i més d'un 45% d'identitat).
- un per P.capsici (contig PHYCAscaffold_29, E-value 2-109).
- deu en el genoma de P.polycephalum (el primer hit, el més significatiu, correspon al Contig6376, E-value 2-11 i 38,1% d'identitat, la resta comprenen E-values entre 1-05 i 5-10 i identitats entre 55.56% i 71.11%).
- nou per S.arctica (on el primer, el més significatiu, es correspon al scaffold supercont1.1171, amb un E-value de 3-24 i un 62,64% d'identitat, i la resta van resultar en E-values compresos entre 2-08 i 9-20; i identitats entre el 39% i el 78%).
Els resultats obtinguts del tblastn per P.infestans van mostrar els resultats següents, pels quals no es va obtenir cap alineament:
- quatre hits en el genoma del protist C.fasciculata, dos corresponents al scaffold_15 (el primer amb una identitat de 30.56% i el segon de 33.67%) i dos corresponents al scaffold_3 (el primer amb una identitat de 30.56% i el segon de 23.26%), amb uns E-values compresos entre 4-09 i 2-16.
- un hit en el genoma del protist I.multifiliis_strain_G5, corresponent al scaffold gi|340500597|gb|GL984358.1| amb un E-value de 7-77 i una identitat de 42.31%.
- deu hits en el genoma del protist P.polycephalum. Els hits més significatius van ser el segon (Contig192370), el tercer (Contig184282) i el quart (Contig33891), tots amb un E-value de 7-06 i una identitat de 60.53%. La resta van donar uns E-value compresos entre 1-04 i 5-06 i uns percentatges d’identitat d'entre 28.57% i 73.08%.
- nou hits en el genoma de protist S.arctica. El primer hit, que es correspon al scaffold supercont1.1171, va ser el més significatiu amb un E-value de 2-20 i una identitat de 56.52%. Els altres hits van resultar en E-value compresos entre 3-05 i 8-17 i en percentatges d'identitat d'entre 33.68% i 73.21%.
D’altra banda, pels següents hits obtinguts al realitzar el tblastn, sí que es varen obtenir alineaments: es van observar dos hits molt significatius en el genoma del protist A.laibachii Nc14. Tant en el cas del primer hit (amb una identitat de 51.88%) com en el del segon hit (amb una identitat de 62.18%), que es corresponen amb el scaffold gi|325192005|emb|FR824421.1|, es va obtenir un E-value de 1e-178. Seguidament, es va realitzar un anàlisi més exhaustiu mitjançant el programa exonerate i el programa Genewise. Al comparar els resultats obtinguts en ambdós programes, va ser seleccionat el resultat procedent del programa Genewise, ja que proporcionava un alineament més òptim (la regió conservada era més extensa). Llavors es va procedir a realitzar un alineament múltiple de les proteïnes predites pel programa Genewise per comprovar si els dos hits es corresponien a la mateixa seqüència o eren seqüències diferents. L’alineament va mostrar que els dos hits del scaffold gi|325192005|emb|FR824421.1| tenien la mateixa seqüència. Això es deu a que el segon hit (10705,12310) inclou dins de la seva seqüència el primer hit (11174,11841).
Amb tot això es conclou que A.laibachii Nc14 presenta al seu genoma una proteïna homòloga de selO i per comprovar-ho fem un alineament múltiple de selO de P.infestans, de selO humana i de la selenoproteïna predita en A.laibachii Nc 14:
1.3 SelO humanaPel protist P.capsici es va obtenir un hit molt significatiu, que es correspon amb l'scaffold PHYCAscaffold_29, amb un E-value de 1-123. Comparant els resultats de l'exonerate amb els de Genewise es va veure que la millor predicció era la que feia el programa exonerate, ja que alinea una seqüència més extensa i n'obtenim un resultat més òptim. Tot i així, la selenocisteïna no s'alinea amb una X sinó amb una asparagina (N). Sabem que poc probablement es pot trobar un homòleg de selO amb N, per tant, creiem que és cosa del programa. Per acabar de confirmar-ho, es van buscar tant elements SECIS com presència de tRNA. Mentre els resultats pel tRNA varen ser negatius, es va predir un element SECIS, usant el mateix patró que per selTryp, i es va obtenir un COVE score de 13,24. Això ens fa pensar que segurament sí que hi és present aquesta selenoproteïna al genoma de P.capsici, encara que no ho podem afirmar amb tota seguretat. Tot i així, al presentar eEFsec, pstk, secp43, SLAPL, sps1 i sps2, és a dir, gairebé tota la maquinària necessària per poder traduir selenoproteïnes tenim grans esperances en aquest genoma i creiem que en una futura prediccióó més precisa serà identificada.
Pel que fa als resultats obtinguts pel protist S.arctica, amb el programa tBLASTn es van obtenir onze hits, dels quals, el primer, que va ser el més significatiu, es correspon al scaffold supercont1.1171 amb un E-value de 2-22. Només es van alinear els hits del 4 al 7, que comprenen valors de E-values entre 1e-06 i 1e-19, mitjançant el programa exonerate. Tots es corresponen al scaffold supercont1.415 i per comprovar si es tractava en tots els casos de la mateixa seqüència i, o bé eren còpies dins el genoma o bé unes incloïen les altres, es va procedir a fer un alineament múltiple d'aquests hits amb selO humana:
S'observa que tots els hits s'alineen per igual. Es defineixen tres regions ben conservades però en la regió prèvia a la X veiem una gran deleció. Creiem que això pot ser degut al que ja hem esmentat al principi: selO al tenir la selenocisteïna al final fa més difícil poder realitzar un bon alineament.
Observant els resultats de l'exonerate i del Genewise, només amb l'exonerate es prediu l'alineament entre X i lisina (K). Com que tampoc esperem homòlegs de selO amb K, pensem que es deu a la dificultat de de detectar-ho a la regió final, afegida a la pròpia dificultat de detectar selenoproteïnes. Respecte els elements SECIS es van predir varis elements i al numero 2 n'observem el millor COVE score: 3,41. Amb aquest valor no podem concloure amb seguretat gaire cosa, ja que s'ha fet amb el mateix patró que totes les anteriors que ja sabem que ens pot donar falsos positius. Els resultats en la predicció de tRNA varen ser negatius, però en aquest genoma es van predir altres elements de la maquinària com eEFsec, pstk, sbp2, secp43, SLAPL, sps1 i sps2. Això ens fa pensar que segurament espècies anteriors a S. arctica varen tenir o tenen selenoproteïnes i, per tant, en aquest genoma s'ha conservat la maquinària i l'estructura de selO.
Pel protist A.laibachii Nc14 els resultats de l'alineament van ser molt significatius. Tant en el cas del primer hit com en el del segon hit, que es corresponen amb el scaffold gi|325192005|emb|FR824421.1|, i a uns valors de E-value de 5-113. Tot i que els resultats de l'alineament mitjançant Tcoffee, de selO amb la proteïna predita amb l'exonerate no van mostrar els resultats desitjats, és a dir, no es va veure la X alineada amb res, s'observa un alineament bo que no ens permet descartar aquest genoma. Per això es va procedir a la recerca d'elements SECIS i se'n van predir varis, entre els quals el numero 2 va mostrar el valor COVE score més alt: 6,92. En la recerca de la maquinària es va detectar la presència de tRNAsec en aquest genoma, de eEFsec, i de secp43.
Prèviament hem conclòs que A.laibachii Nc14 presenta al seu genoma un homòleg amb cisteïna de selO provinent del mateix contig. Amb aquestes dades podríem pensar que enlloc de tractar-se d'un homòleg es tracta d'una selenoproteïna mal anotada. Però si s'agafa la protïena predita a Genewise contra selO humana i la predita contra selO de P.infestans , es pot veure que en la descrita amb selO humana s'estava perdent la informació de l'última part de la proteïna on hi ha la C.
Fent un alineament múltiple es conclou que al genoma d'A.laibachii Nc 14 existeix un homòleg de la selO amb cisteïna.
Altres alineaments obtinguts mitjançant tBLASTn de selO humana amb els genomes dels protist, van donar resultats significatius que després d'un anàlisi més exhaustiu van ser descartats. Principalment, per dues raons: o bé, perquè l'alineament de la selenoproteïna no es donava, o bé perquè els hits eren molt curts i poc significatius.
Pel protist F.cylindrus es van obtenir quatre hits, dos corresponents al scaffold_15 i altres dos corresponents al scaffold_3, amb uns E-values de 7-05 i 3-16, respectivament. En aquest cas els resultats varen ser descartats perquè en cap dels casos s'obtenia un alineament òptim i tampoc es va detectar la presència d'elements SECIS. Tot i així es van predir tRNAsec en aquest genoma igual que eEFsec, sbp2, secp43, SLAPL, sps1 i sps2. Això ens fa pensar que aquestes prediccions no s'han d'ignorar tot i que tampoc esperaríem que en anàlisis més precisos trobéssim la selO. Segurament en aquest genoma s'han obtingut aquests resultats ja que conté estructures molt conservades pròpies de les selenoproteïnes obrint portes a la possibilitat de trobar-ne d'altres.
Pel protist I.multifiliis strain G5 es va obtenir un hit, corresponent al scaffold gi|340500597|gb|GL984358.1| amb un E-value de 5-81. En els resultats de Tcoffee amb la proteïna predita a partir de l'exonerate s'observa una regió molt conservada a l'inici però no s'alinea la selenocisteïna de selO. Respecte els resultats de Genewise, en la proteïna predita alineada s'observa una altre regió conservada diferent i un alineament de X amb asparagina (N). Com ja s'ha esmentat anteriorment no esperem trobar homòlegs amb N de selO, i com en la recerca dels elements SECIS no s'han obtingut resultats no creiem que en aquest genoma existeixi la selO com a tal.
Per P.polycephalum es van obtenir catorze hits. Aquest genoma va ser descartat pel segon motiu: perquè la majoria dels hits tenien una allargada molt curta i molt poc significativa. El primer hit, corresponent al scaffold Contig6376 i va donar un E-value de 2-14. Els altres hits van ser menys significatius, amb E-values compresos entre 1-04 i 9-09. Els hits 3, 4 i 5; 7, 8 i 9; i el 13 es van alinear en els resultats predits amb el programa Genewise i s'ha fet una alineament múltiple per mostrar els resultats. S'observa que en general els alineaments són molt dolents, tot i que s'alinei la part del final, on hi ha la selenocisteïna. Per això, i al no trobar elements SECIS tampoc creiem que aquesta selenoproteïna hi sigui present.
SelP
Es va trobar un hit significatiu quan es va realitzar un tblastn agafant la query d'un invertebrat més proper als nostres protists: D. discoideum AX4. Tot i així, el valor és molt proper al límit que hem decidit en la realització del tblastn automàtic (E-value superior a 0,001).
El programa d'exonerate automàtic no va ser capaç d'obtenir resultats, així que es va realitzar un genewise on es va obtenir una proteïna curta i no contenia cap U ni C, d'aquesta manera el tcoffee genewise no donarà un bon alineament.
Al descartar l'únic hit significatiu possible, podem afirmar que no hem obtingut cap selenoproteïna ni cap homòleg amb cisteïna. Això és possible, ja que la SelP és una proteïna que de moment s'ha trobat en vertebrats i només en dos invertebrats. Malgrat utilitzar la query d'un homòleg conegut, S. kowalevskii que conserva el primer domini de la SelP humana com es pot apreciar en el següent Tcoffee, aquesta regió és un domini enzimàtic que pot no tenir rellevància en protists. També eés el cas de la funció del segon domini, transportar el seleni reactiu pel cos, la qual els protistes no la necessiten.
SelTryp
Els alineaments que resulten del tBLASTn de la query selTryp humana i el genomes tant de T.congolense com de C.fasciculata són molt significatius, tal com es pot veure a la taula dels resultats.
Pel protist C.fasciculata es van obtenir quatre hits: el primer i el segon, que es corresponen amb el contig1035, i va donar uns valors d'E-value de 1-24 i de 8-13 respectivament; i el tercer i quart hit, corresponents al contig896, que van donar uns E-values de 2-24 i 8-13, respectivament. Davant aquests resultats podríem pensar que existeixen quatre còpies de selTryp en el genoma de C.fasciculata.
Tots quatre hits varen ser analitzats i es van obtenir els següents resultats: la selenocisteïna de la proteïna selTryp s’alinea amb les proteïnes predites en el genoma d’aquest. Mentre pel contig1035 usant Genewise no s'obtenia cap alineament, amb els resultats de l'exonerate es va predir que la selenocisteïna s'alinea amb una leucina (L). No esperem trobar homòlegs amb lisina de selTryp, per tant, es creu que segurament al programa li costa reconèixer la selenocisteïna com a tal i ens ho està alineant amb l’aminoàcid que més convé. Respecte el contig896 cal dir que amb el programa Genewise és amb el que s'ha obtingut l'alineament més òptim i significatiu, ja que el programa alinea la selenocisteïna amb una altre suposada selenocisteïna (X-X).
Per poder afirmar que el trobat era una selenoproteïna es van buscar tant els elements SECIS com els tRNA de les seqüències genòmiques extretes i els resultat no van ser gaire concloents. Es van predir varis elements SECIS usant l’opció “loose (canonical and non-canonical)” obtenint el número 3 amb un valor COVE score de 2.63, el més alt. Tenint en compte que usant aquest patró s’estan detectant elements SECIS amb desviacions significatives de les consens, segurament els elements predits es corresponen a falsos positius. Tot i això, i tenint en compte que els resultats pel tRNA van ser negatius, el que fa pensar que potser es tracta d’una selenoproteïna és que al resultat del tblastn veiem un 40% d’identitat en ambdós contigs i els valors E-value no són gens despreciables. A més, en aquest genoma, s'han identificat els següents elements de la maquinària eEFsec, secp43, SLAP, sps1 i sps2. Tanmateix, només ho afirmaríem en el cas del contig896, que és en el que s'ha predit l'alineament correctament.
Pel protist T.congolense es va obtenir un únic hit, però molt significatiu: amb més d’un 70% d’identitat i amb un E-value de 2-86. Es correspon al scaffold T.congo.pschr.5 i, igual que amb l’anterior, es va procedir a la recerca tant dels elements SECIS com dels tRNA. Val a dir però, que els resultat tampoc van ser gaire concloents. Es van predir 9 possibles elements SECIS usant també l’opció “loose (canonical and non-canonical)” però tots amb un COVE score de 0. Tenint en compte l'anterior, deduïm que segurament els elements predits es corresponen a falsos positius. Tot i això i que els resultats pel tRNA van ser negatius, el que fa pensar que potser es tracta d’una selenoproteïna és el resultat del tblastn i l'alineament resultant de Genewise.i, igual que abans, la identificació de la maquinària eEFsec, secp43, SLAP, sps1 i sps2. Diem el de Genewise perquè amb l'exonerate s'ha alineat la selenocisteïna de la query amb una lisina (K) i no molt poc probablement esperem trobar homòlegs amb K per selTryp.
Es pensa que al tractar-se de la recerca de selenoproteïnes tot encara està en desenvolupament i que cal de prediccions més exactes i exhaustives per determinar si selTryp hi és o no amb seguretat tant a T.congolense com en C.fasciculata.
També es van obtenir resultats significatius en els genomes de les dues espècies de Leishmania.
Per L.donovani BPK282A1 es van obtenir tres hits, els tres corresponents al contig_35, amb uns E-values de 4-24 el primer hit, 7-16 el segon i 1-05 el tercer. Tot i això, no es va trobar la selenocisteïna de la query alineada correctament, és a dir, amb una X o una U sinó que es va alinear amb asparagina (N). Sabem, però, que és molt poc probable que existeixin homòlegs en asparagina de selTryp, per tant, creiem que possiblement es tracta d’un error del programa al reconèixer la selenocisteïna com a tal i ens ho està alineant, altre vegada, amb l’aminoàcid que més convé. També es van buscar elements SECIS per veure si realment es podia descartar la presència de selTryp al genoma. Es van obtenir varies prediccions i la número 3, amb el valor COVE score més alt: 1.61, indica que ni podem negar ni afirmar, ja que es va fer amb el mateix patró que els anteriors. Creiem que o bé, d’aquesta selenoproteïna se n’ha conservat el gen al genoma però ja no es tradueix, o bé, el programa no l’anota correctament.
Finalment, per L.tarentolae es van obtenir dos hits, que es corresponen al scaffold Lt_contig1348, amb E-values de 3-24 en el cas del primer hit, i 4-14 en el segon. En els resultats de l’alineament ens succeeix el mateix que en l’anterior, no s’alinea correctament sinó que ho fa amb una glicina (G). Tampoc esperem homòlegs en glicina de selTryp, per tant, concloem el mateix. En aquest cas, però, el resultat de la recerca de SECIS pot ser més concloent: ens dona un únic hit amb COVE score de 0. Amb això interpretem que gairebé segur estem parlant d’un fals positiu, per tant, segurament la selenoproteïna com a tal no es tradueix en aquesta espècie.
En ambdues espècies també es van predir els següents elements de la maquinària eEFsec, secp43, SLAP, sps1 i sps2.
Partint de la premissa anterior, pensem que en Leishmania hi ha una regió molt conservada de selTryp i per confirmar-ho es realitza un BLASTp recíproc amb la seqüència de T.congolense. Efectivament, veiem que altres espècies d’aquesta família presenten alineaments significatius amb la selenoproteïna, per tant, afirmem que, al menys, comparteixen dominis conservats entre elles.
Similaritat de SelTryp i SelTCom es pot veure, en els resultats del BLASTp va aparèixer una altre selenoproteïna: la selT. Això ens va fer pensar que selTryp i selT podrien compartir regions o fins i tot tenir el fragment on es troba la selenocisteïna molt semblants. Per tal de comprovar–ho, es va realitzar un TCoffee amb les seqüències de selTryp i selT (obtinguda a SelenoDB).
La seqüència de selT és:
> SPP00000025_1.0 # Protein # Selenoprotein T (SelT) # Homo sapiens # Complete MRLLLLLLVAASAMVRSEASANLGGVPSKRLKMQYATGPLLKFQICVSUGYRRVFEEYMRVISQRYPDIRIEGENYLPQPIYRHIASFLSVFKLVLIGLIIVGKDPFAFFGMQAPSIWQWGQENKVYACMMVFFLSNMIENQCMSTGAFEITLNDVPVWSKLESGHLPSMQQLVQILDNEMKLNVHMDSIPHHRS
Es va realitzar un TCoffee per veure la similaritat de les seves estructures on es veu que la regió més conservada és on es troba la selenocisteïna:
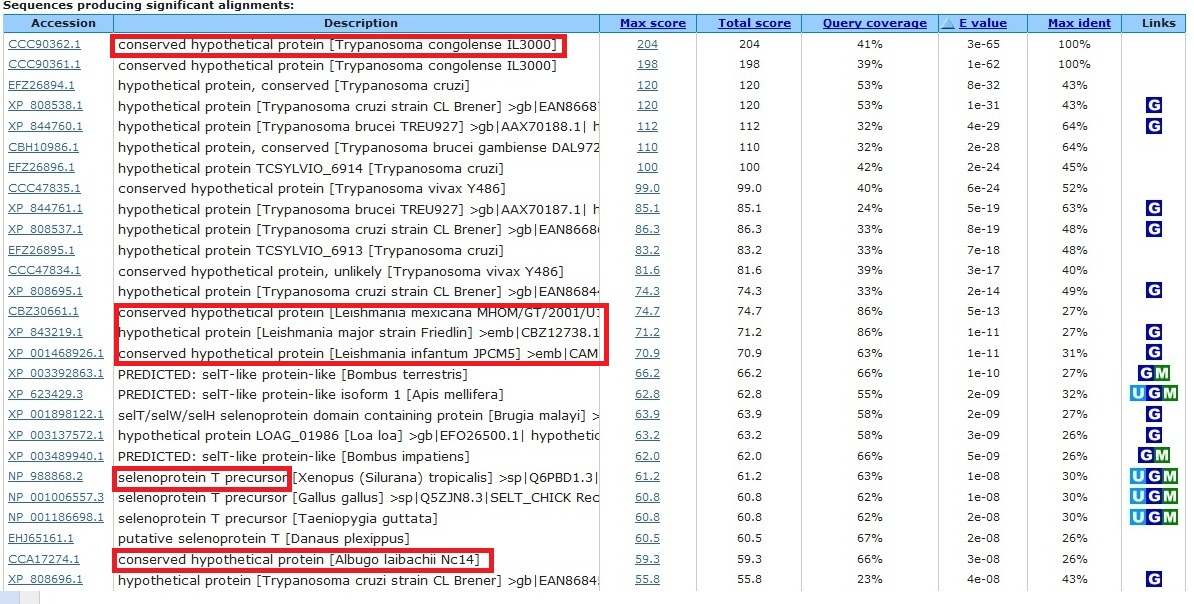
Sorprenentment, en els resultats del BLASTp també observem que un dels alineaments significatius es correspon a una proteïna hipotètica al genoma de Albugo laibachii Nc14, protist pel qual no s’havia obtingut cap hit al tblastn i, per tant, s’havia descartat. Per comprovar que aquestes proteïnes s’alineen es fa un TCoffee i efectivament obtenim un resultat positiu.
Però l'alineament no es dona com a selenoproteïna sinó com a cisteïna, és a dir, es podria pensar que al genoma d’aquest protist es va trobar una selTryp homòloga amb cisteïna.
Però tenint en compte que les estructures de selT i selTryp no són molt diferents, es va realitzar un TCoffee múltiple amb la selenoproteïna T, la selenoproteïna Tryp i la homòloga de A.laibachii Nc14 per comprovar si es podia o no tractar d'un homòleg de cisteïna de selTryp.
Eus aquí la gran sorpresa: l'alineament resultant és més òptim. Per tant, es va pensar que pot ser aquesta proteïna de A.laibachii Nc14 que crèiem homòloga per selTryp ho era per SelT. Es va tornar a fer el Tcoffe, però només de selT amb la homòloga.
Es va veure que, efectivament, era homòloga amb cisteïna per selT i no per selTryp com inicialment s'hagués pogut creure, ja que l'alineament era clarament més bo en selT que en selTryp.
El fet de que les dues selenoproteïnes tinguin homologia en les regions on hi ha la selenocisteïna ens ha produït fals positius de SelTryp, com el cas de A.laibachii Nc14.
Per acabar de confirmar que les selenoproteïnes obtingudes a T.congolense i C.fasciculata són realment de SelTryp, es realitzen múltiples alineaments amb el tcoffee.
Primer realitzem alineament múltiple de SelT, SelTryp i proteïna obtinguda del genewise de T.congolense.
Observem que hi ha alineament molt bo en les dues regions que comparteixen selTryp i selT, però fora d’aquestes regions l’alineament no ho és tant. Si comparem aquest alineament múltiple realitzat (selT, selTryp i selTryp de T.congolense), amb el tcoffee genewise de selTryp i la selenoproteïna de T.congolense, per tal de veure si la presència de selT millora o empitjora l’alineament:
S'observa que el resultat d'ambdues soles és molt millor que en l’alineament múltiple. Així podem afirmar que la selenoproteïna trobada a T.congolense és selTryp i no selT.
Per últim, realitzem el mateix procediment per la proteïna obtinguda amb els resultats del Genewise de C.fasciculata.
Comparem l’alineament múltiple de selT, selTryp i C.fasciculata amb el de selTryp i C.fasciculata sols:
Igual que abans, s'ha observat que és millor l’alineament amb selTryp que amb selT. Així, també podem afirmar que el resultat obtingut és una selenoproteïna Tryp.
1.2 Alineaments no significatius
SelO
A partir dels resultats obtinguts al realitzar tBLASTn, es van descartar els següents protists ja que no se’n va obtenir cap resultat per selO humana:
- A. rara, C. fasciculata, D. Discoideum AX4, D. fasciculatum, G. niphandrodes, L. donovani BPK282A1, L. tarentolae i T. congolense.
Després de l’exonerate, els resultats van permetre descartar els següents hits, pels quals no es va predir res:
- Hit 13 de P. Polycephalum.
Per selO de E.siliculosus, el tBLASTn ja va descartar:
- G. niphandrodes, D. fasciculatum, D. discoideum AX4, L. donovani BPK282A1, L. tarentolae, T.congolense, C. fasciculata i A. rara
Per selO de P.infestans, el tBLASTn va descartar:
- A. rara, C. fasciculata, D. Discoideum AX4, D. fasciculatum, G. niphandrodes, L. donovani BPK282A1, L. Tarentolae, P.capsici i T.congolense
En cap de les dues anteriors l'exonerate va descartar altres hits.
SelP
El primer que es va fer tblastn de forma manual per cada protist contra la seqüència de SelP humana, obtenint, així, els seus E-values respectius. D’aquests no hi havia cap hit significatiu tot i que quatre d’ells podien fer dubtar (P. capsici, G. niphandrodes, L. tarentolae i T. congolense).
Per tal de comprovar la fiabilitat dels nostres resultats obtinguts de forma manual es va repetir el procés de forma automatitzada i els resultats obtinguts van ser els mateixos.
Com que els humans estan molt lluny evolutivament dels protists, es va realitzar el mateix procediment però comparant amb un organisme més proper, com és S. kowalevskii . S’observa que els E-values en general són més petits, tot i així s'obté un hit significatiu.
SelTryp
A partir dels resultats obtinguts al realitzar tBLASTn, es van descartar els següents protists ja que no se’n va obtenir cap resultat:
- A. laibachii Nc14, A. rara, D. discoideum AX4, D. fasciculatum, F. cylindrus, G. niphandrodes, P. capsici, P. Polycephalum i S. Arctica.
Després de l’exonerate, els resultats van permetre descartar els següents hits, pels quals no es va predir res:
- És el cas del hit 0 de I. multifiliis strain G5 i el hit 0 de T. Congolense.