Introducció
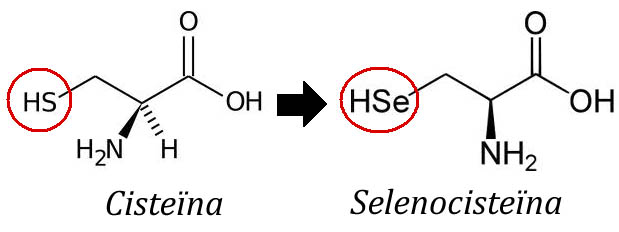
Les selenoproteïnes són una classe de proteïnes que contenen una selenocisteïna en la posició número 21 de la seva seqüència. La selenocisteïna (Sec) és un aminoàcid poc freqüent, anàleg de la Cisteina (Cys) però que enlloc d'un àtom de sofre(S) en conté un de Seleni (Se) i ve codificat pel codó UGA, el qual, per norma general, és un codó de terminació de la traducció.
El seleni és un micronutrient essencial pels eucariotes, especialment per la implicació que té en la formació de les selenoproteïnes. Un dèficit pot portar a malalties com la de Keshan, una cardiomiopatia que afecta les dones joves i els nens en certes regions de Xina, però un excés és tòxic. No obstant en la seva mesura adequada s’ha vist que té efecte protector contra les malalties inflamatòries com artritis, asma i aterosclerosis. (24 de selenoprot synthesis).[2][5]
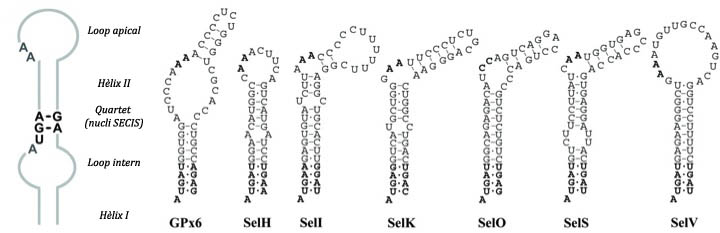
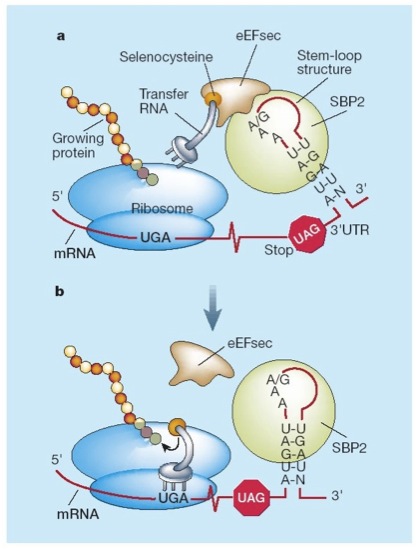
La principal senyal per a la recodificació de UGA com a codó per a la selenocisteina és una estructura de RNA secundaria anomenada SECIS (SElenoCysteineInsertion Sequence) que és una estructura tridimensional en forma de loop formada per 60 nucleòtids, 4 dels quals estàn conservats en el nucli. Es troba en l'extrem 3' no traduït (3'UTR) dels transcripts gènics eucariòtics o bé downstream després del codó UGA en posició 3’, en el cas de procariotes.[4]
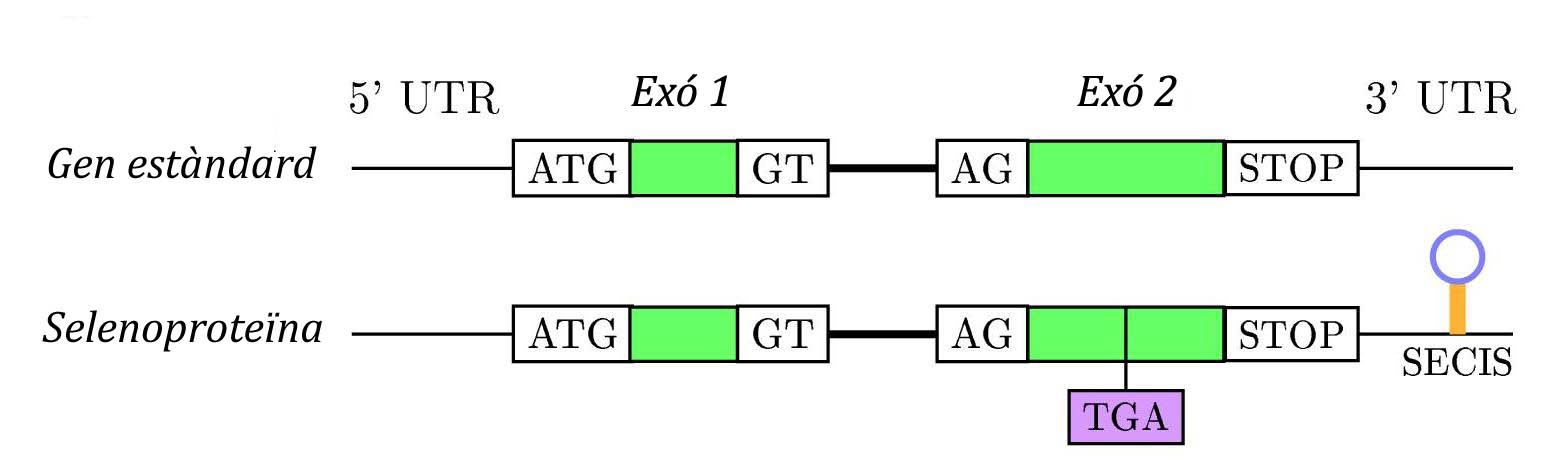
Aquest element SECIS apropa el tRNA de la selenocisteïna al codó UGA mitjançant la unió de SBP2 amb EfSec. Això permet seguir amb la traducció fins al pròxim codó STOP, i d’aquesta manera sintetitzar una selenoproteïna. Durant aquest procés intervenen varies proteïnes: [1][3]
- Selenofosfat sintetasa 1 i 2 (SPS1 i SPS2) que transformen el seleni de la dieta a monoselenofosfat per tal que es pugui transferir a la seril-tRNASec. Es creu que la SPS1 recicla el seleni d’altres selenocisteïnes mentre que la SPS2 és més important perquè genera el monoselenofosfat.
- SLA/LP és una selenocisteïna sintasa que participa en el pas de Ser-tRNASec a Sec-tRNASec (selenocistenil-tRNASec) mitjançant la defosforilació de la Serina prèviament fosforilada per la PTSK per transferirli el monoselenifosfat al tRNA per formar el Sec-tRNASec.
- PSTK (Phosphoseryl tRNA kinase): kinasa que fosforil·la el Ser-tRNASec.
- Secp43 (selenocysteine associated protein), necessària per la metilació de tRNASec. Forma part del complex format per SCP2/tRNASec/eEFSec.
- SBP2 (SECIS binding protein): s’uneix a l’element SECIS i recluta el complex eEFsec-selenocisteil-tRNASec en el ribosoma.
- eEFsec : proporciona Sec-tRNASec al lloc A del ribosoma per la incorporació de Sec a la proteïna que s’està sintetitzant.
- tRNAsec : s’uneix al codó UGA i transporta la Sec fins al ribosoma.
Actualment es coneixen 21 famílies de selenoproteïnes en eucariotes superiors: Glutathione Peroxidases (GPx), Iodothyronine Deiodinase (DI), Selenoprotein 15 (Sel15 or 15kDa), Fish selenoprotein 15 (Fep15), SelM, SelH, SelI, SelJ, SelK, SelL, SelN, SelO, SelP, SelR, SelS, SelT, SelU, SelV, SelW, Thioredoxin Reductases (TR), SelenoPhosphate Synthetase (SPS) i algunes d'elles contenen més d'un membre en un genoma determinat. Fins fa poc, els mètodes computacionals per a la seva identificació estaven basats en la predicció d'elements SECIS, però això no funcionava en tots els casos ja que, tot i que sí que es manté l'estructura secundària, la seqüència es conserva molt poc, donant lloc a falsos positius i negatius. Un altre mètode consistia en escanejar la seqüència diana buscant marcs de lectura (ORFs) amb un UGA conservat, donant lloc a molts possibles candidats però pocs de verdaders. Així doncs, aquest fet ha portat a que en moltes anotacions de genomes, les selenoproteïnes s’ometin, a causa que un codó UGA sigui assumit com a codó STOP de la regió codificant, a què el primer AUG downstream del codó UGA sigui l'inici d'una regió codificant, o bé a causa d'exclusió de l'exó o de la regió que conté el codó UGA/Sec.[4] Per això actualment s’han descobert nous mètodes com el selenoprofiles que no elimina per complet la intervenció manual però la redueix molt. Aquest no es basa en la predicció de SECIS ni tampoc en les seqüències individuals de selenoproteïnes com a queries inicials, sinó en perfils de seqüències característics de cada família de seleproteïnes eucariòtica. És un mètode basat en la homologia que permet produir prediccions acurades de famílies conegudes de selenoproteïnes i pot ser usat juntament amb programes d'anotació automàtica.[12]
SelO és una selenoproteïna àmpliament distribuïda que té homòlegs en animals, bacteris, llevats i plantes. Tot i així la funció roman desconeguda. Està formada per 669 aminoàcids i en humans es troba en el cromosoma 22. Només els vertebrats homòlegs de SelO tenen selenocisteïna que es troba en la penúltima posició de l'extrem C-terminal. En els homòlegs de SelO en altres organismes, la selenocisteïna és substituïda per una cisteïna.[14]
També podeu consultar l'entrada que hem modificat a viquipèdia.
>SPP00000019_1.0 # Protein # Selenoprotein O (SelO) # Homo sapiens # Complete MAVYRAALGASLAAARLLPLGRCSPSPAPRSTLSGAAMEPAPRWLAGLRFDNRALRALPV EAPPPGPEGAPSAPRPVPGACFTRVQPTPLRQPRLVALSEPALALLGLGAPPAREAEAEA ALFFSGNALLPGAEPAAHCYCGHQFGQFAGQLGDGAAMYLGEVCTATGERWELQLKGAGP TPFSRQADGRKVLRSSIREFLCSEAMFHLGVPTTRAGACVTSESTVVRDVFYDGNPKYEQ CTVVLRVASTFIRFGSFEIFKSADEHTGRAGPSVGRNDIRVQLLDYVISSFYPEIQAAHA SDSVQRNAAFFREVTRRTARMVAEWQCVGFCHGVLNTDNMSILGLTIDYGPFGFLDRYDP DHVCNASDNTGRYAYSKQPEVCRWNLRKLAEALQPELPLELGEAILAEEFDAEFQRHYLQ KMRRKLGLVQVELEEDGALVSKLLETMHLTGADFTNTFYLLSSFPVELESPGLAEFLARL MEQCASLEELRLAFRPQMDPRQLSMMLMLAQSNPQLFALMGTRAGIARELERVEQQSRLE QLSAAELQSRNQGHWADWLQAYRARLDKDLEGAGDAAAWQAEHVRVMHANNPKYVLRNYI AQNAIEAAERGDFSEVRRVLKLLETPYHCEAGAATDAEATEADGADGRQRSYSSKPPLWA AELCVTUSS#
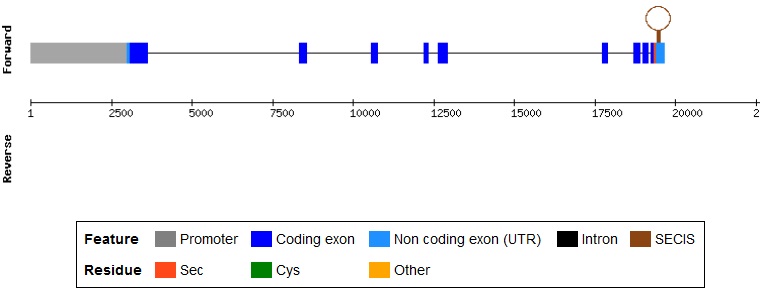
La selenoproteïna P va ser identificada al 1977. Es caracteritza per ser una glicoproteïna extracel·lular que es troba en abundància en el plasma i unida a les cèl·lules endotelials. En humans es troba al cromosoma 5, consta de 381 aminoàcids i conté 10 residus de selenocisteïna, els quals es troben distribuïts en dos dominis.[6]
El domini Nt és semblant a un domini enzimàtic ja que la selenocisteïna es troba en un motif redox UxxC, en canvi el domini Ct, és un mitjà de transport segur del seleni reactiu pel cos. El seu gen consta de cinc exons, dels quals la seqüència codificant es troba en el 2 i el 5. En l’exó 2 es troba el primer UGA i set residus de cisteïna i en el 5 hi ha 9 selenocisteïnes, 10 cisteïnes i dos SECIS.
Existeix l’evidència que la proteïna s’encarrega de l’homeòstasi del seleni i de la defensa oxidant en l’espai extracel·lular. Es va veure que tenia capacitat d’actuar com una phospholipid hydroperoxide glutathione peroxidase.
S’ha vist que és una proteïna que s’uneix a heparina, ja que té dues regiones riques en histidines que són candidates a ser lloc d’unió. És capaç d’unir-se a diferents metalls pesants com el mercuri. Es creu que la selenoproteïna P sols s’expressa en vertebrats. El fetge és la font del 75% de la secreció de selenoproteina P al plasma, encara que tots els teixits poden expressar el seu mRNA. Als ronyons i al cor també trobem altes concentracions. S’ha vist que els testis i el cervell tenen receptors per selenoproteïna P i que up-regulen el seu mecanisme per obtenir seleni de la selP quan hi ha deficiència de seleni. S’han realitzat estudis que demostren que la presència de SelP en el cervell confereix una neuroprotecció per malalties com l’Alzheimer, també és el cas de que s’ha observat que una disminució de SelP en les cèl·lules prostàtiques produeix un augment de radicals lliures que promouran el desenvolupament del tumor.[7][8]
També es va estudiar l’opció que la sobreproducció hepàtica de selenoproteïna P produís insulina resistència a nivell hepàtic i muscular, d’aquesta manera pot aparèixer una diabetis tipus II. La falta de SelP des d’inicis produeix una degeneració del tronc cerebral i el tàlam, i pot comportar problemes en l’aprenentatge i alteració de la transmissió sinàptica basal i de la plasticitat de la regió CA1 del hipocamp, produint manca de memòria a curt termini.[9][10]
També podeu consultar l'entrada que hem modificat a viquipèdia.
>SPP00000020_1.0 # Protein # Selenoprotein P (SelP) # Homo sapiens # Complete MWRSLGLALALCLLPSGGTESQDQSSLCKQPPAWSIRDQDPMLNSNGSVTVVALLQASUY LCILQASKLEDLRVKLKKEGYSNISYIVVNHQGISSRLKYTHLKNKVSEHIPVYQQEENQ TDVWTLLNGSKDDFLIYDRCGRLVYHLGLPFSFLTFPYVEEAIKIAYCEKKCGNCSLTTL KDEDFCKRVSLATVDKTVETPSPHYHHEHHHNHGHQHLGSSELSENQQPGAPNAPTHPAP PGLHHHHKHKGQHRQGHPENRDMPASEDLQDLQKKLCRKRCINQLLCKLPTDSELAPRSU CCHCRHLIFEKTGSAITUQCKENLPSLCSUQGLRAEENITESCQURLPPAAUQISQQLIP TEASASURUKNQAKKUEUPSN#
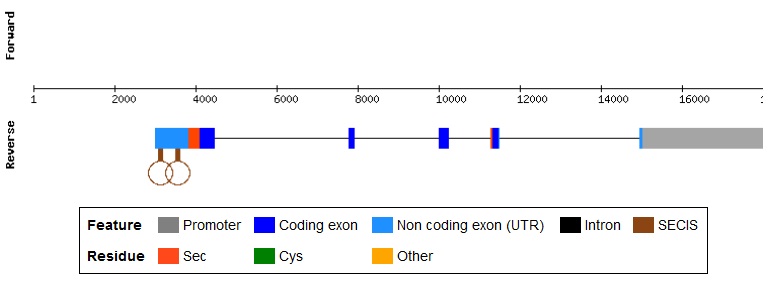
La selenoproteïna Tryp es troba localitzada en el cromosoma 4 (889449..891788) de Trypanosoma brucei i té una mida de 2340 parells de bases. Els tripanosomàtids són un grup exclusiu de protozous parasítics quinetoplàstids responsables de diferents tipus de tripanosomiasis causades per Trypanosoma spp., com la malaltia de la son o la malaltia de Chagas, i diferents tipus de leishmaniosis causades per Leishmania spp. Actualment, s'han destinat molts esforços a la seqüenciació completa d'ambdós genomes, que ja estan a la nostra disposició. L'anotació genòmica correcta i la comprensió de les funcions protèiques d'aquests organismes es consideren crucials pel desenvolupament de nous fàrmacs i per la profilaxis de les malalties que causen. El problema és que les eines de anotació actuals no són capaces d'identificar gens que codifiquen per selenocisteïnes (Sec), ja que es codifiquen pel codó UGA, un codó de terminació. Tant en Leishmania major, com Trypanosoma cruzi i Trypanosoma brucei s'han identificat gens codificants per homòlegs de la selenofosfatasa sintasa, un enzim que genera selenofosfat, el donador de seleni necessari per la biosíntesi de Sec. [11]
També podeu consultar l'entrada que hem modificat a viquipèdia.
> SelTryp; hypothetical protein [Trypanosoma brucei TREU927] PARTIALLY FROM XP_844760.1 MRNRFVRISFSLALLIAAFLAANAGSATNADNEATDYSSHRYKYLEMLKEADLKQMLHEKGEAFHHLRSK RELILAVLKLEKREEALRKARVTDTVQHEVRVEYCSGUGYRRHYEEVAESLLRSLPPELREQQKGKKPFI KFVGVVYSVGAFREFIGNILSTGFLASIAISFFAPFLRGALPPHIAEWIEQHRGMVVGAGFMMNMVASSL LQSGAFEVYLNGSLIYSKLETGAVPTAETLADHILRQIISGTAAGTRTA
Des del 2005, segons The International Society of Protistologists, existeixen sis regnes. Dintre aquests trobem el gran i extens antic regne dels protists o també anomenat protoctista que engloba a diferents espècies dels sis regnes actuals. Es caracteritza per ser un taxó parafilèctic, és a dir, que inclou diferents fílums d’organismes eucariotes unicel·lulars amb trets tan d’animals com de plantes i algunes algues unicel·lulars.
Un protist es defineix per les següents característiques:
- Té especialització intracel·lular, és a dir, divisió funcional dins la cèl·lula i per tant existència d’orgànuls funcionals
- Poden ser sèssils o de vida lliure
- Tenen reproducció asexual per divisió mitòtica
- Alguns presenten reproducció sexual verdadera on es forma un zigot
- Presenten respostes enfront la presència d’estímuls
- Alguns tenen exoesquelet simple
- La seva nutrició és autòtrofa, sapròfita o holozòica.
- Presenten uns sistemes de locomoció en el medi aquàtic més perfeccionats.
Els protists es troben allà on existeix la vida. Són molt adaptables a canvis deguts a factors físics. Necessiten humitat, tant si viuen al mar, a l’aigua dolça, a terra, en matèria en descomposició, en plantes o animals.
Tot i la seva àmplia distribució, molts protists poden viure amb èxit només en uns estrets marges ambientals. Tenen un paper enorme en l’economia natural, ja que al voltant de 10000 espècies de protists són simbionts que viuen a sobre o en plantes, animals i fins i tot altres protozous.
Un cop hem introduït els protists, descriurem els onze protists en què es basa el nostre estudi: