Discussió
Família SelR
Aquesta família està formada per tres proteïnes: SelR1, SelR2 i SelR3. La primera d'elles ens ha donat dos possibles hits, un dels quals l'hem descartat perquè era un alineament d'una regió molt petita de la proteïna. Les altres dues ens han donat els 2 mateixos hits en el BLAST i utilitzant els valors d'e-value i la informació dels bits del BLAST, i els diferents alineaments del t-coffee, hem pogut assignar un hit a cada una de les proteïnes.
SelR1
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna SelR1 es troba a l'scaffold GL841555.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
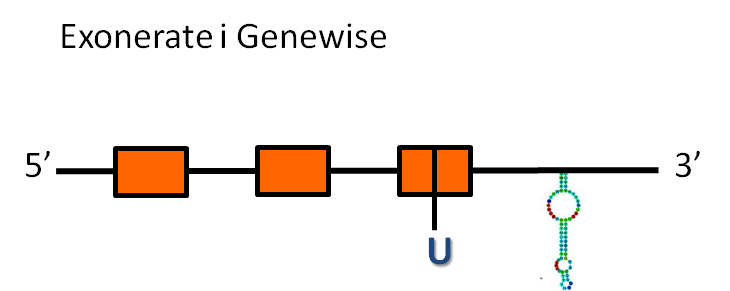
- La predicció d'exonerate té 9525 nucleòtids, que inclouen tres exons i dos introns, i una raw score de 482.
- La predicció de genewise té 9525 nucleòtids, que inclouen tres exons i dos introns, i té una puntuació de 181.42 bits.
Les dues prediccions comencen i acaben al mateix nucleòtid de l'scaffold, és a dir, els dos prediuen la mateixa proteïna.
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el tercer exó de les proteïnes predites tan per exonerate com per genewise.
El programa secisSearch ha trobat un possible elements secis a l'extrem 3' del gen, la presència d'el qual dóna suport a la predicció que es tracta una selenoproteïna conservada.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna SelR1:
SelR2
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna SelR2 es troba a l'scaffold GL861683.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
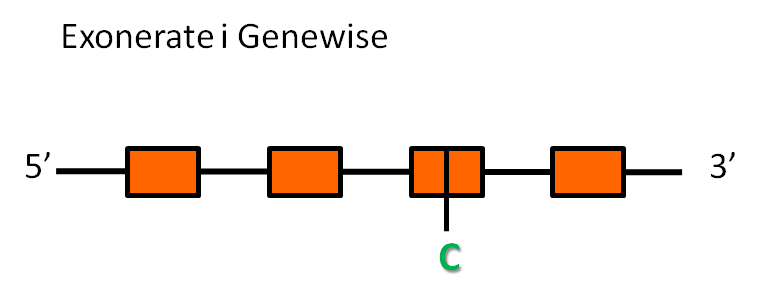
- La predicció d'exonerate té 24688 nucleòtids, que inclouen quatre exons i tres introns, i una raw score de 559.
- La predicció de genewise té 24688 nucleòtids, que inclouen quatre exons i tres introns, i té una puntuació de 207.68 bits.
Les dues prediccions comencen i acaben al mateix nucleòtid de l'scaffold, és a dir, els dos prediuen la mateixa proteïna.
La proteïna d' Homo sapiens no té una selenocisteïna, sinó que té una cisteïna, que es troba conservada en la nostra proteïna en el quart exó predit tan per exonerate com per genewise.
El programa secisSearch no ha trobat cap element secis a l'extrem 3' de la proteïna predita, cosa que tampoc és contradictòria perquè la proteïna no té selenocisteïna i, per tant, no és necessari.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna SelR2:
SelR3
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna SelR3 es troba a l'scaffold GL861565.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
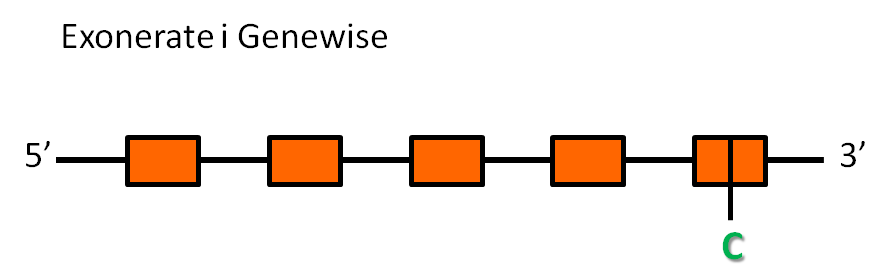
- La predicció d'exonerate té 119153 nucleòtids, que inclouen cinc exons i quatre introns, i una raw score de 704.
- La predicció de genewise té 119153 nucleòtids, que inclouen cinc exons i quatre introns, i té una puntuació de 275.56 bits.
Les dues prediccions comencen i acaben al mateix nucleòtid de l'scaffold, és a dir, els dos prediuen la mateixa proteïna.
La proteïna d' Homo sapiens no té una selenocisteïna, sinó que té una cisteïna, que es troba conservada en la nostra proteïna en la nostra proteïna en el cinquè exó predit tan per exonerate com per genewise.
El programa secisSearch no ha trobat cap element secis a l'extrem 3' de la proteïna predita, cosa que tampoc és contradictòria perquè la proteïna no té selenocisteïna i, per tant, no és necessari.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna SelR3:
Selenoprofiles
Per aquesta família el programa Selenoprofiles ha trobat tres hits. Cada un d'aquests hits coïncideix amb la posició de cada una de les tres proteïnes que formen la família SelR.
Cal dir, però, que quan afirmem que concorden en posició, no volem dir que es situïn al mateix nucleòtid exacte en els dos resultats, sinó que aproximadament coincideixen a i alineen la mateixa regió.
Cal dir que el programa Selenoprofiles no ens troba cap element secis per cap dels hits.
La proteïna que nosaltres anomenem SelR1 ens coincideix amb l'scaffold del hit SelR.1 del Selenoprofiles. Aquest hit consta de 3 exons, que s'estenen a 9474 nucleòtids, no es troba element secis i té una cïsteina alineada, tal com havíem trobat nosaltres. Aquest hit té la selenocisteïna alineada. Cal dir que les posicions no són exactament les mateixes entre aquest hit i el trobat per nosaltres, però sí que solapen a la mateixa regió.
La proteïna que nosaltres anomenem SelR2 ens coincideix amb l'scaffold del hit SelR.10 del Selenoprofiles. S'exté 24623 nucelòtids i consta de 4 exons, no s'han trobat elements secis . A més es troba una cïsteina alineada, tal com haviem fet nosaltres. Cal dir que les posicions no són exactament les mateixes entre aquest hit i el trobat per nosaltres, però sí que solapen a la mateixa regió.
La proteïna que nosaltres anomenem SelR3 ens coincideix amb la posició del hit SelR.6 del Selenoprofiles. Tot i així, en aquest hit veiem que el resultat obtingut per nosaltres difereix considerablement del resultat de Selenoprofiles. Veiem que els dos inicis cauen més o menys a la mateixa regió, però en canvi, els dos finals disten molt un de l’altre, fent que els dos alineaments tinguin una diferència de 122.000 nucleòtids aproximadament, sent molt més llarg l’obtingut per Selenoprofiles.