Introducció
SELENOPROTEÏNES
Què són?
Les selenoproteïnes són proteïnes que incorporen a la seva seqüència, com a mínim, un aminoàcid anomenat selenocisteïna (SeC), el qual correspon a l’aminoàcid 21, i s’anomena amb una lletra U.
El nombre de selenoproteïnes existents en les diferents espècies varia molt segons el tàxon. Per exemple, es coneixen unes 30 selenoproteïnes en peixos, 25 en humans, 3 en Drosophila melanogaster i 1 en Caenorhabditis elegans. Tanmateix, no tots els organismes tenen selenoproteïnes, per tant deduïm que no són essencials per a la vida. En el cas de les plantes encara no se n’ha trobat cap. De fet, entre els eucariotes és bastant comú, mentre que en altres fílums, no tant.
Funció de les selenoproteïnes
Les selenoproteïnes estan involucrades directament en les reaccions redox (d’oxidació-reducció). Presenten propietats antioxidants, de manera que protegeixen dels possibles danys cel·lulars causat per radicals lliures. Aquests radicals són productes derivats del metabolisme i s’ha vist que en humans contribueixen al desenvolupament de patologies com el càncer o malalties coronàries. Així doncs, participen en la homeostasis redox i en la defensa davant l’estrès oxidatiu.
A part de la funció antioxidant, les selenoproteines també participen en la regulació de la funció tiroidea i tenen un paper important en el sistema immunitari.
L'aminoàcid selenocisteïna
La selenocisteïna és igual que la cisteïna, però ha substituït el radical de sofre (SH) per un de seleni (SeH). El seleni i el sofre ocupen la mateixa columna a la taula periòdica dels elements, fet que els confereix propietats químiques similars, tot i que el seleni resulta ser un element més reactiu que el sofre, cosa que confereix a les selenoproteines importants capacitats oxido-reductores.
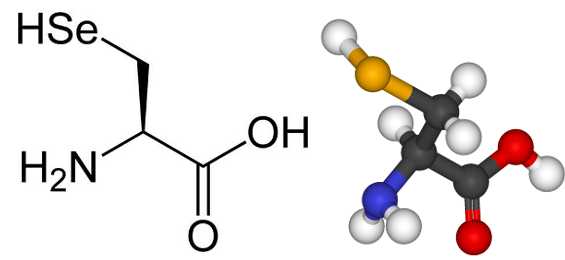
Aquest aminoàcid està codificat pel codó UGA, un codó que normalment indica un STOP en la transcripció. Això dificulta molt la cerca i predicció d’aquestes proteïnes. La recodificació d’aquest codó UGA com a aminoàcid selenocisteina és regulada o induida per una estructura d’ARN tridimensional que es troba en l'extrem 3' no traduït (3’-UTR) dels gens de les selenoproteïnes d’eucariotas, o downstream del codó UGA en procariotes, i que s’anomena SElenoCystein Insertion Sequence o element SECIS.
El paper del seleni en la natura
El seleni és un element traça de nombre atòmic 34, que tot i ser poc comú a la Terra es considera essencial per al desenvolupament de determinades funcions fisiològiques dels organismes. Tot i que la seva funció concreta i els processos exactes en que intervé encara no s’han definit amb exactitud, es coneixen i s’han descrit alguns dels efectes que provoca sobre l’organisme.
El seleni el trobem com a element químic en el nostre cos sobretot en forma de selenoproteïnes. Apareixen de manera molt àmplia al llarg de l’evolució i les podem trobar en els tres dominis de la vida: eucariota, bacterià i archaea. De fet, les selenoproteïnes són el principal reservori de seleni en els metazous.
El seleni (Se) és un micronutrient essencial que resulta de vital importància per a la fisiologia d'una gran varietat d'espècies, incloent-hi als humans. La falta de seleni pot donar lloc diverses malalties, com per exemple la malaltia de Keshan (nom que prové de la província de Kesh (Xina) on tenien nivells baixos de seleni), la qual es caracteritza per una debilitació muscular causant problemes cardíacs. Diversos estudis han demostrat que el subministrament de suplements de seleni pot ser de gran utilitat pels pacients amb aquesta malaltia. Per altra banda, l’excés de seleni pot conduir a una intoxicació greu denominada selenosi. Aquest síndrome es deriva del fet que el nostre organisme no és capaç de metabolitzar quantitats elevades d’aquest element que s’acumularà en extremitats i articulacions. A més a més, hi ha estudis demostren que el seleni actua en la prevenció de certes malalties, com és el cas del càncer de la pròstata. Tot i així tots aquests estudis encara no son prou concloents.
Recodificació del codó UGA
Com ja hem dit, el codó que codifica per la selenocisteïna és l’UGA, el qual normalment és un codó STOP, però la seva recodificació induïda per l’element SECIS farà que no sigui reconeguda com a parada de la traducció, i passi a codificar per una selenocisteïna.
L’element SECIS es tracta d’una estructura secundària d’ARN en forma de loop, situada en l’extrem 3’-UTR del gen de la selenoproteïna en eucariotes, o downstream del codó UGA en procariotes, que recluta la maquinaria de traducció evitant la terminació. Dels 60 nucleòtids que la formen, només 4 estan conservats en el nucli o core. Tot i aquesta baixa conservació a nivell deseqüència de ADN, la estructura secundària sí que es troba molt conservada.
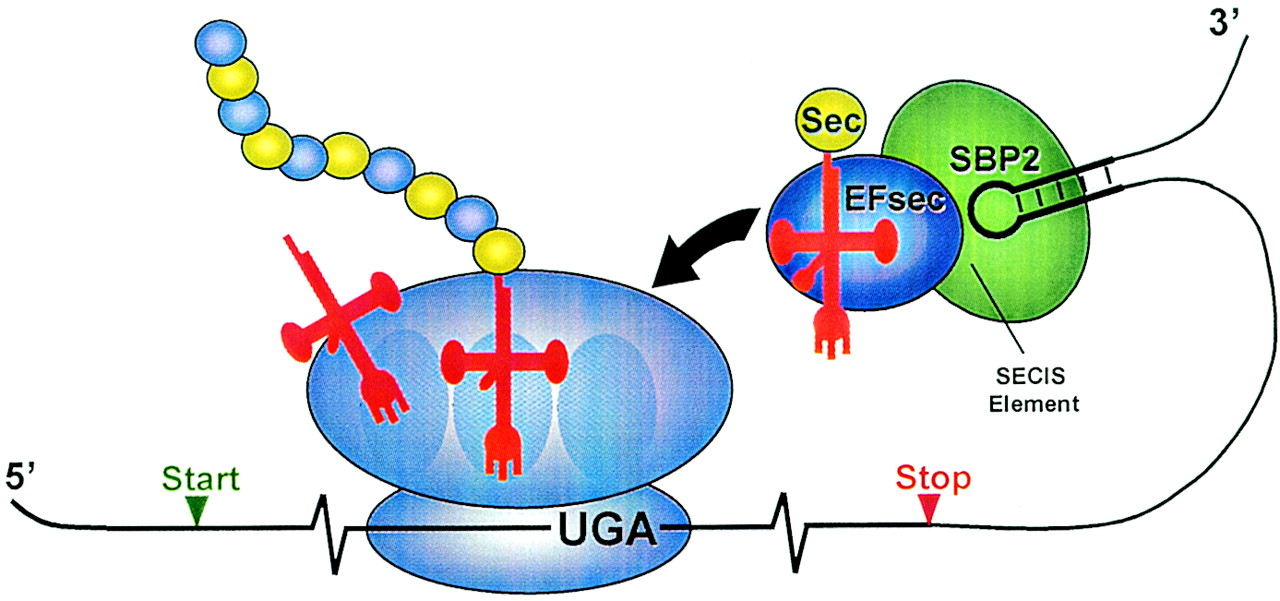
Aquest element recluta una proteïna anomenada SECIS Binding Protein (SBP2), la qual uneix un factor d’elongació específic de selenoproteïnes (eEFsec). eEFsec aproxima el tRNA específic de la selenocisteïna (tRNAsec) al codó UGA del mRNA de la selenoproteïna i en determina la seva traducció a selenocisteïna. La traducció de mRNA continua fins que la maquinaria de traducció reconeix un nou codó de terminació.
Sintesis de selenoproteïnes
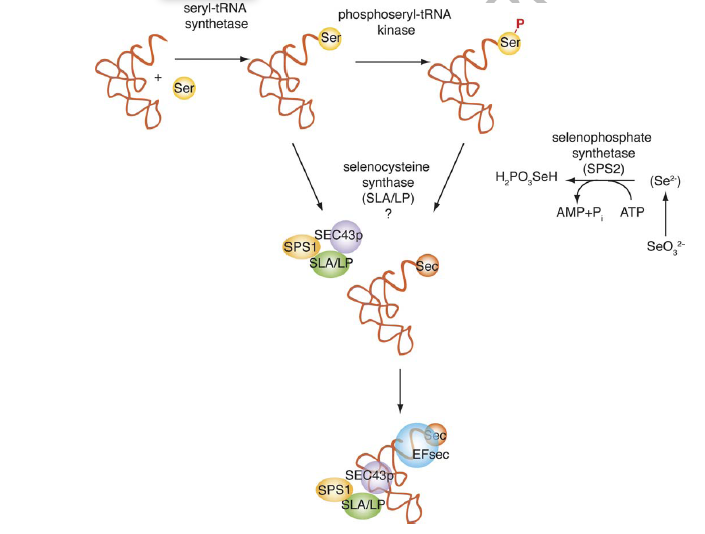
Cal una maquinaria específica tant per sintetitzar la selenocisteïna a partir de la serina (es sintetitza selenocistenil-tRNAsec a partir de seril-tRNAser), com per incorporar aquesta selenocisteïna en la selenoproteïna que s’està codificant.
En la sintesi de la selenocisteïna hi intervenen:
- Selenofosfat sintetasa 1 i 2 (SPS1 i SPS2): que transformen el seleni de la dieta a monoselenofosfat per tal que es pugui transferir al seril-tRNAser per acció de PSTK. Es creu que la SPS1 recicla el seleni d’altres selenocisteïnes mentre que la SPS2 és més important perquè genera el monoselenofosfat.
- PSTK (Phosphoseryl tRNA kinase): kinasa que fosforil·la la serina del seril-tRNAser per transferir-li el monoselenofosfat. Ara tenim doncs fosfoseril-tRNAser.
- SLA/LP: és una selenocisteïna sintasa que participa en el pas de fosfoseril-tRNAser a selenocistenil-tRNAsec mitjançant la defosforilació de la serina prèviament fosforilada per la PTSK.
- Secp43 (selenocysteine associated protein): es una proteïna necessària per estabilitzar el complex SECIS-SBP2/eEFsec/selenocistenil-tRNAsec i participa en la metilació de tRNAsec.
En la incorporació de la selenocïsteina en la selenoproteïna que s’està codificant hi intervenen:
- SBP2 (SECIS binding protein): s’uneix a l’element SECIS i recluta el complex eEFsec/selenocistenil-tRNAsec en el ribosoma.
- eEFsec (eukaryotic elongation factor, selenocysteine-tRNA-specific): és un factor d’elongació específic per selenocisteïna-tRNAsec ja que proporciona Sec-tRNASec al lloc A del ribosoma per la incorporació de Sec a la proteïna que s’està sintetitzant.
- tRNAsec: s’uneix al codó UGA i transporta la Sec fins al ribosoma.
Història natural de les selenoteïnes
Els selenoproteomes coneguts ens ofereixen una visió de l’evolució de les selenoproteïnes una mica confosa i desconcertant. Cada una d’elles sembla tenir la seva pròpia història evolutiva: poden ser presents en una determinada espècie o grup taxonòmic, absents en un altre grup filogenèticament proper, i en canvi, tornar a ser presents (com a selenoproteïnes o homòlegs en Cys) en un grup taxonòmic filogenèticament allunyat.
Cada nou genoma analitzat aporta noves sorpreses i fa que calgui replantejar fets que es donaven gairebé per establerts. Per exemple, fins fa poc es pensava que les selenoproteïnes eren essencials per la vida animal, donat que tots els animals coneguts tenien selenoproteïnes i C. elegans mantenia tota la maquinària molecular necessària només per sintetitzar una selenoproteïna. Tanmateix, després de l’obtenció de la seqüència del genoma de dotze espècies de Drosophila, es va descobrir, mitjançant anàlisis computacionals, que D.willistoni no té selenoproteïnes i que ha perdut la capacitat de codificar-ne (perquè ha perdut alguns gens que formen part de la maquinaria necessària per sintetitzar-les).
Aquest esdeveniment va fer plantejar a la comunitat científica dos grans preguntes:
1. La pèrdua de selenoproteïnes en D.willistoni va comportar alguna conseqüè ncia funcional, o resulta que les selenoproteïnes són realment innecessàries per als insectes? Per tal de respondre a aquesta pregunta, sabent que les selenoproteïnes estan implicades en la resposta al estrés oxidatiu, es va dur a terme un estudi comparatiu en el que posaven diferents espècies de Drosophila en medi oxidatiu i en medi neutre. Es va veure que D.willistoni sobrevivia menys temps que les altres espècies en el medi oxidatiu, i van arribar a la conclusió de que la pèrdua de les selenoproteïnes havia fet que D.willistoni fos més sensible al estrés oxidatiu.
2. Que es perd primer, les pròpies selenoproteïnes o la capacitat per codificar-les? Tot i ser cert que D.willistoni havia perdut alguns gens de la maquinària per sintetitzar selenoproteïnes, es va veure que alguns gens els quals s’assumia que la seva funció era la síntesi de selenoproteïnes estaven tant conservats com en altres espè cies que sí tenien selenoproteïnes (Secp43 i SPS1). Així doncs, van concloure que primer va ser la pèrdua de les selenoproteïnes, moltes per mutagènesis de la selenocisteina a cisteïna, i després la pèrdua dels gens de la maquinaria que van deixar de ser necessaris. El fet de que encara n’hi hagin alguns de conservats s’explicaria bé perquè aquests estan implicats en altres funcions importants, o bé perquè encara no ha passat prou temps com per a que desapareguin.
L’anàlisi recent de múltiples genomes d’insectes seqüenciats ha permès comprovar que la pèrdua de selenoproteïnes no es una cosa única i específica de D.willistoni, ja que s’ha vist que les selenoproteïnes s’han extingit en moltes altres espècies d’insectes, tot i que n’hi ha d’altres que encara les mantenen. Així doncs estàvem equivocats al pensar que les selenoproteïnes eren essencials per la vida animal. És molt complicat, des de un punt de vista evolutiu, trobar un patró d’extinció d’aquestes selenoproteïnes, però ens ha de quedar clar que s’han hagut de perdre de manera independent en les diferents espècies.
Un altre punt clau en la evolució de les selenoproteïnes, es la conversió d’aquestes en el seu homòleg amb cisteïna, que s’ha donat en múltiples casos en la natura. Si bé es molt freqüent trobar gens que han mutat de selenoproteïna al homòleg amb cisteïna, mai s’ha trobat el procés de mutagènesis invers, de cisteïna a selenocisteïna, per la qual cosa no esperem l’aparició de noves selenoproteïnes.
Dificultats en la predicció i anotació de selenoptoteïnes
La dificultat en la cerca de selenoproteïnes rau en el fet que contenen selenocisteïa, la qual és codificada pel codó UGA que generalment constitueix un codó d’STOP. Qualsevol programa automàtic d’identificació de gens s’equivocarà en la identificació de selenoproteïnes, ja que sempre que trobi un codó UGA el considerarà sistemàticament com un codó d'stop. Per aquest motiu, l’anotació de les selenoproteïnes sol ser incorrecte i ha de ser corregida un cop trobades.
Per poder-les identificar a nivell de seqüència nucleotídica és necessari trobar alguna manera d'identificar els elements SECIS amb l'objectiu de saber quins codons UGAs no codifiquen per codons d'stop sinó per Selenocisteïnes. El problema, és que no s’ha trobat cap patró de seqüència SECIS, és a dir, no hi ha cap conservació en la seqüència d’aquests elements que ens permeti identificar-los.
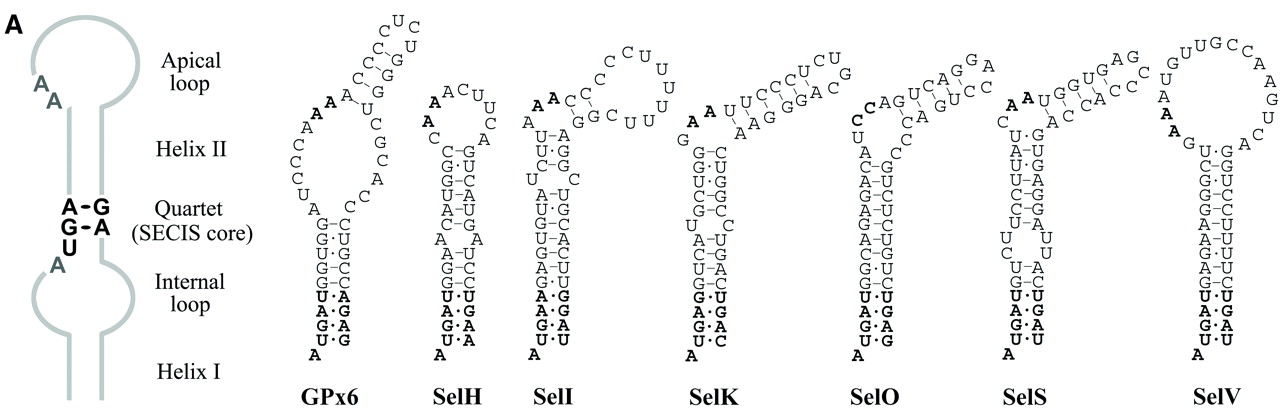
Inicialment es creia que entre tots ells només tenen conservats un quartet de nucleòtids (AUGA), però posteriorment s'han identificat quartets diferents. Per aquest motiu, els SECIS que tenen el quartet AUGA s'anomenen canònics i la resta no canònics.
Tot i que la seqüència nucleotídica d’aquests elements no es conserva, l’estructura tridimensional sí. Per tant, una estratègia per trobar aquests SECIS pot ser buscar les seqüències que es puguin plegar i formar aquesta estructura 3D coneguda (loop-helix-loop). PatScan és un dels diversos programes que poden fer aquesta cerca mitjançant la predicció aproximada de l'estructura d'un RNA petit. Se li dóna el patró estructural i un conjunt de seqüències i el programa mostra quines d’aquestes seqüències poden adoptar aquesta estructura.
Per fer una predicció eficient de la localització de selenoproteïnes es sobreposen la informació dels elements SECIS amb la localització d'exons del gen que estem estudiant. Combinant aquests dos tipus de informacions podem reduir notablement el nombre de possibles selenoproteïnes.
Podem aplicar altres estratègies, com ara la cerca de patrons de conservació upstream i downstream del codó UGA. Trobar un patró conservat a partir del codó UGA és indicatiu de que aquell codó no es un codó de STOP, perquè si ho fos significaria que tot el que segueix seria no codificant i per tant esperaríem una baixa conservació de la seqüència nucleotídica.
Actualment s’han descobert nous mètodes com el selenoprofiles que no elimina per complet la intervenció manual però la redueix molt. Aquest no es basa en la predicció de SECIS ni tampoc en les seqüè ncies individuals de selenoproteïnes com a querys inicials, sinó en perfils de seqüències característics de cada família de seleproteïnes eucariòtica. És un mètode basat en la homologia que permet fer prediccions acurades de famílies conegudes de selenoproteïnes i pot ser usat juntament amb programes d'anotació automàtica. La limitació principal d’aquest mètode es que només serà aplicable per aquelles selenoproteïnes ja conegudes i anotades en altres genomes.
Selenoproteïnes conegudes
Actualment es coneixen 21 famílies de selenoproteïnes en eucariotes superiors: Glutathione Peroxidases (GPx), Iodothyronine Deiodinase (DI), Selenoprotein 15 (Sel15 or 15kDa), Fish selenoprotein 15 (Fep15), SelM, SelH, SelI, SelJ, SelK, SelL, SelN, SelO, SelP, SelR, SelS, SelT, SelU, SelV, SelW, Thioredoxin Reductases (TR), SelenoPhosphate Synthetase (SPS) i algunes d'elles contenen més d'un membre en un genoma determinat.
FILOGÈNIA: SELENOPROTEÏNES EN ANIMALS MAMÍFERS
L'estudi de selenoproteïnes permet caracteritzar els genomes i s'utilitza, entre d'altres, per veure la proximitat filogenètica entre diferents organismes. Anomenem selenoproteoma al conjunt de proteïnes que són o que estan relacionades amb les selenoproteïnes.
Els tres dominis reconeguts en la natura viva són Eucariota, Arquea i Eubacteria (els dos últims també anomenats Arqueabacteria i Bacteria, respectivament). En els últims anys, els eucariotes s’han classificat en 4 regnes: animals, plantes, fongs i protists.
L’objectiu del nostre treball és el de estudiar i analitzar el selenoproteoma del Dimoni de Tasmània (Sarcophilus harrisii), que és una espècie de marsupial de la família Dasyuridae. Com podem veure a la imatge inferior, l'espècie més propera al Dimoni de Tasmània ès el Wallaby (Macropus rufogriseus).
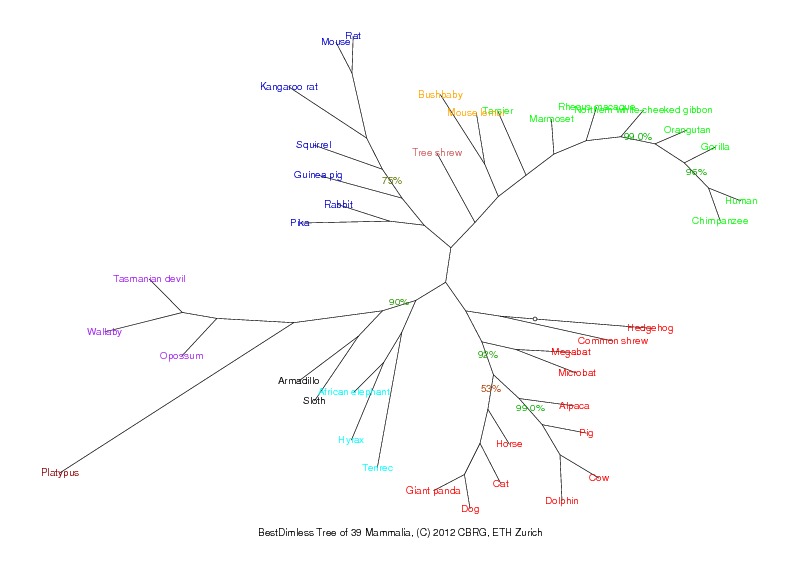
Així doncs, per tal de saber quines proteïnes cercar, un cop dins el regne animal, haurem de resseguir l' evolució de les selenoproteïnes començant pels vertebrats, passant pels mamífers i arribant finalment al Wallaby.
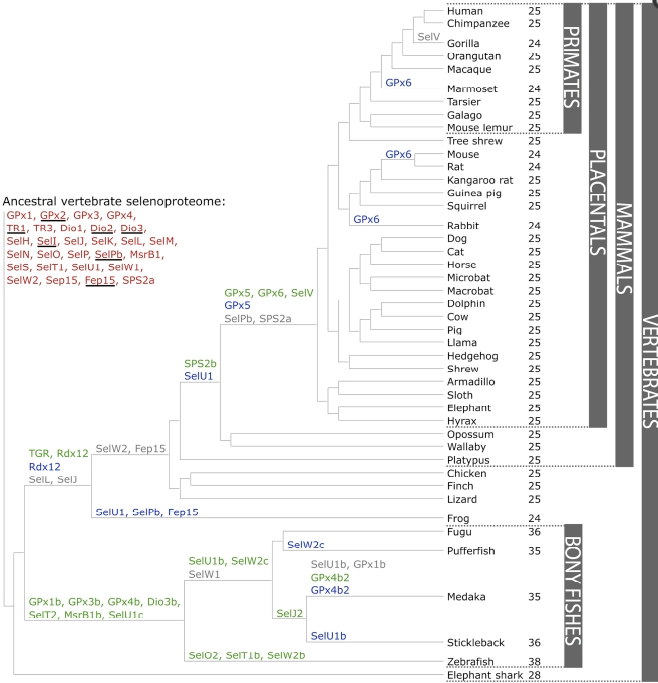
Com podem veure, al llarg de la evolució de les selenoproteïnes en els vertebrats, han tingut lloc 20 duplicacions (verd), 9 deleccions (gris) i 13 mutacions als homòlegs amb cisteïna(blau).
EL DIMONI DE TASMÀNIA
El dimoni de Tasmània
El diable o dimoni de Tasmània (Sarcophilus harrisii) és un marsupial carnívor que actualment només es troba en estat salvatge a l'estat insular australià de Tasmània. El diable de Tasmània és l'únic membre vivent del gènere Sarcophilus. Amb la mida d'un gos petit, però robust i musculat, el diable de Tasmània és actualment el marsupial carnívor més gran del món, després de l'extinció del llop marsupial el 1936. Es caracteritza pel seu pelatge negre, la pudor que emet quan es posa nerviós, un xiscle extremament fort i pertorbador i la seva ferocitat quan s'alimenta. Se sap que caça preses i s'alimenta també de carronya. Tot i que sol ser solitari, a vegades s'alimenta en grups. Tot i el seu aspecte corpulent, pot desenvolupar una velocitat sorprenent i pot pujar als arbres i nedar pels rius.
El diable de Tasmània fou extirpat del continent australià uns 400 anys abans de la colonització europea el 1788. Com que eren vistos com a amenaça pel bestiar a Tasmània, els diables foren caçats fins al 1941, quan foren protegits oficialment. Des de finals de la dè cada del 1990, la malaltia de tumors facials del diable de Tasmània ha reduït la població de diables significativament i actualment amenaça la supervivè ncia de l'espè cie.
El diable de Tasmània té una constitució com ajupida i gruixuda, amb un cap gran i una cua que fa aproximadament la meitat de la seva allargada corporal. El diable emmagatzema greix corporal a la cua, de manera que els exemplars malalts solen tenir una cua prima. S'estima en sis anys la longevitat mitjana d'un diable de Tasmània salvatge, tot i que en captivitat poden viure més temps. Té uns llargs bigotis a la cara que li permeten detectar preses quan busca en la foscor i l'ajuden a saber quan hi ha altres diables a prop durant l'alimentació. L'oïda és el seu sentit dominant i també té un excel·lent sentit de l'olfacte. Com que els diables cacen de nit, sembla que la seva visió és més aguda en blanc i negre. En aquestes condicions, poden detectar fàcilment objectes en moviment, però tenen dificultats a l'hora de veure objectes estacionaris. Té la mossegada més potent de qualsevol mamífer vivent. La potència de la mandíbula es deu en part al seu cap relativament gran. Té un conjunt de dents que creix lentament al llarg de la seva vida
és un símbol icònic de Tasmania i moltes organitzacions, grups i productes associats amb aquest estat l’utilitzen en els seus logotips.

Les femelles comencen a criar quan assoleixen la maduresa sexual, sovint al seu segon any de vida. En aquest punt, esdevenen fèrtils una vegada per any i produeixen diversos òvuls mentre els dura el zel. L'aparellament té lloc al març, en llocs protegits, tant durant el dia com la nit. Els mascles es barallen per les femelles durant l'època d'aparellament i les femelles s'aparellen amb el mascle dominant. Els diables no són monògams i les femelles s'aparellen amb diversos mascles si el mascle dominant no les vigila després d'aparellar-s'hi. La gestació dura 21 dies, al final dels quals neixen 20-30 cries. Quan neixen les cries, hi ha una competència ferotge quan es mouen de la vagina al marsupi. Un cop dins el marsupi, cadascuna d'elles roman enganxada a un mugró durant els següents cent dies. Malgrat la mida de la ventrada al naixement, la femella només té quatre mugrons, de manera que no hi ha mai més de quatre nadons nodrint-se al marsupi; com més vella esdevé una femella, més petites seran les seves ventrades. Les femelles de diable de Tasmània estan ocupades amb la cria dels seus fills durant aproximadament sis mesos de l'any.
Els diables de Tasmània s'estenen i són bastant comuns arreu de Tasmània. Viuen en tots els hàbitats de l'illa, incloent-hi els afores de les zones urbanes, i els agraden en particular els boscos escleròfils i les zones boscoses costaneres. El diable de Tasmània és un caçador nocturn i crepuscular i roman durant el dia entre arbustos frondosos o dins un cau.
Classificació taxonòmica del dimoni de Tasmània
- Regne: Animal
- Filum: cordats
- Classe: mamifers
- Infraclasse: marsupials
- Ordre: Dasiuromofia
- Família: Dasiuridae
- Gènere: Sarcophilus
- Espècie: Sarcophilus harrisii
Selenoproteoma del dimoni de Tasmània
Tenint en compte la classificació taxonòmica del nostre animal, i la filogènia dels vertebrats, en el genoma del Dimoni de Tasmània esperaríem trobar les següents proteïnes: DI1, DI2, DI3, GPx1, GPx2, GPx3, GPx4, TR1, TR3, SelH, SelI, SelK, SelM, SelN, SelO, SelP, SelPb, MsrB, SelT1, SelW1, Sep15 SPS2a, SPS2b, TGR.
Tot i així, tal com veiem en aquesta imatge, els noms de les selenoproteïnes poden variar una mica.
Així doncs, de les selenoproteïnes anteriors podem canviar els noms: MsrB també té el nom de SelR; TGR la podem anomenar TR2 i Sep15 també es pot dir Sel15. Per tant, el llistat final de selenoproteïnes que esperem trobar a priori és el següent: DI1, DI2, DI3, GPx1, GPx2, GPx3, GPx4, Sel15, SelH, SelI, SelK, SelM. SelN, SelO, SelP, SelPb, SelR1, SelS, SelT, SelW1, SPSa, SPS2b, TR1, TR2, TR3.