Discussió
Família GPx
Es tracta d'una família de proteïnes que actualment consta de 8 membres seqüenciats (GPx1 a GPx8). El BLAST de cada una d'elles contra el nostra genoma ens ha donat entre 5 i 7 hits, la majoria dels quals eren comuns en les diferents proteïnes. Això pot ser degut a una gran similitud entre aquestes proteïnes, però mitjançant l'anàlisi manual dels alineaments obtinguts i les seves puntuacions, hem acabat associant cada un dels hits a una proteïna de la família. Tot i això, no hem pogut associar cap hit a les proteïnes GPx5 i GPx6, ja que els seus millors hits eren presents en altres membres de la família i amb alineaments millors, cosa que coincideix amb la filogènia de l'evolució de les selenoproteïnes en vertebrats.
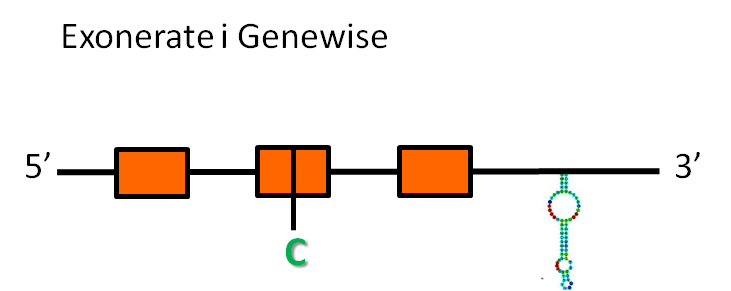
GPx1
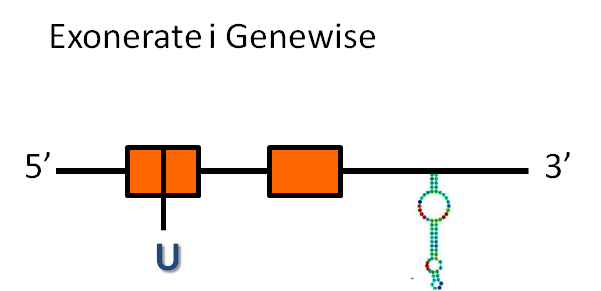
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna GPx1 es troba a l'scaffold GL841583.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
- La predicció d'exonerate té 2597 nucleòtids, que inclouen dos exons i un intró, i una raw score de 826.
- La predicció de genewise té 2597 nucleòtids, que inclouen dos exons i un intró, i té una puntuació de 353.93 bits.
Les dues prediccions comencen i acaben al mateix nucleòtid de l'scaffold, és a dir, els dos prediuen la mateix proteïna.
La proteïna d'Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el primer exó de les proteïnes predites tan per exonerate com per genewise.
El programa secisSearch ha trobat un possible element secis a l'extrem 3' del gen, cosa que dóna suport a la predicció que es tracta una selenoproteïna conservada.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna GPx1:
GPx2
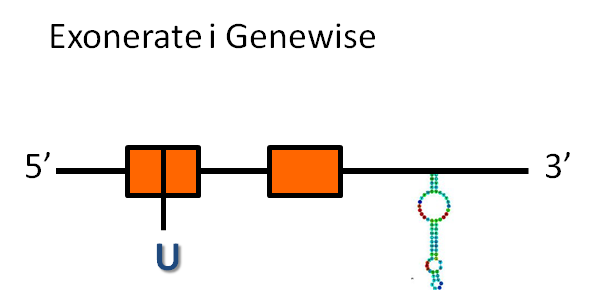
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna GPx2 es troba a l'scaffold GL834546.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
- La predicció d'exonerate té 2658 nucleòtids, que inclouen dos exons i un intró, i una raw score de 933.
- La predicció de genewise té 2658 nucleòtids, que inclouen dos exons i un intró, i té una puntuació de 404.40 bits.
Les dues prediccions comencen i acaben al mateix nucleòtid de l'scaffold, és a dir, els dos prediuen la mateix proteïna.
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el primer exó de les proteïnes predites tan per exonerate com per genewise.
El programa secisSearch ha trobat dos possibles elements secis a l'extrem 3' del gen, a diferents distàncies del final del gen, però no tenim el coneixement per discriminar quin dels dos és el real. Tot i això, la presència d'aquests elements secis dóna suport a la predicció que es tracta una selenoproteïna conservada.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna GPx2:
GPx3
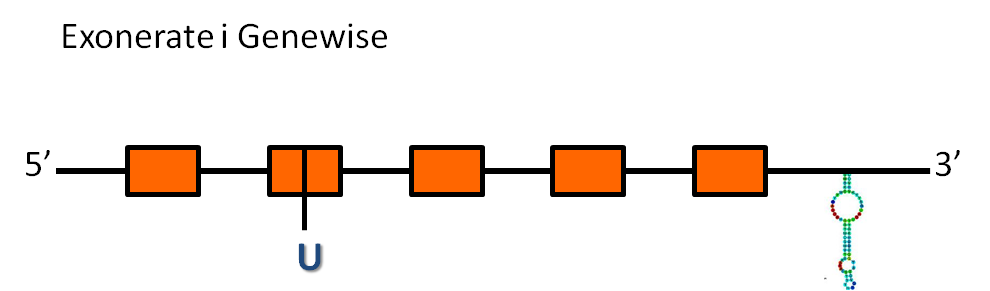
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna GPx3 es troba a l'scaffold GL840403.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
- La predicció d'exonerate té 10197 nucleòtids, que inclouen cinc exons i quatre introns, i una raw score de 878.
- La predicció de genewise té 10197 nucleòtids, que inclouen cinc exons i quatre introns, i té una puntuació de 337.69 bits.
Les dues prediccions comencen i acaben al mateix nucleòtid de l'scaffold, és a dir, els dos prediuen la mateix proteïna.
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el segon exó de les proteïnes predites tan per exonerate com per genewise.
El programa secisSearch ha trobat un possible element secis a l'extrem 3' del gen, cosa que dóna suport a la predicció que es tracta una selenoproteïna conservada.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna GPx3:
GPx4
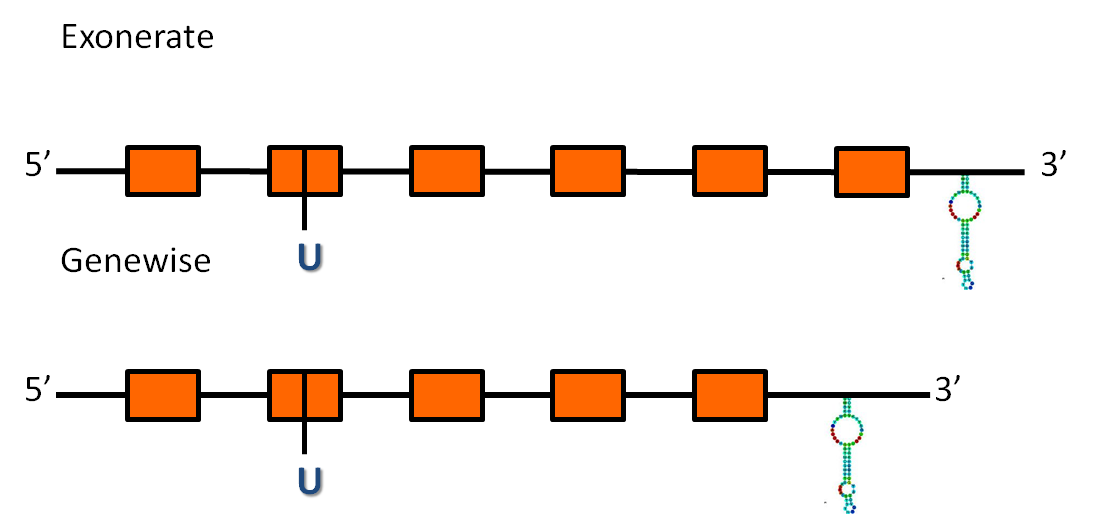
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna GPx4 es troba a l'scaffold GL841423.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
- La predicció d'exonerate té 2427 nucleòtids, que inclouen sis exons i cinc introns, i una raw score de 835.
- LLa predicció de genewise té 2140 nucleòtids, que inclouen cinc exons i quatre introns, i té una puntuació de 296.82 bits.
La predicció de l'exonerate comença tres nucleòtids abans, és a dir, inclou un aminoàcid més a l'inici, així com també inclou un intró i un exó més al final (els dos molt curts). No tenim les eines per discriminar quina de les dues prediccions és millor, però segons l'alineament d'aquestes prediccions amb la seqüència query dut a terme amb el tcoffee, es pot veure que la predicció realitzada per l'exonerate, a diferència de la del genewise, inclou les últimes bases.
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el segon exó de les proteïnes predites tan per exonerate com per genewise.
El programa secisSearch ha trobat quatre possibles elements secis a l'extrem 3' del gen, a diferents distàncies del final del gen, però no tenim el coneixement per discriminar quin dels dos és el real. Tot i això, la presència d'aquests elements secis dóna suport a la predicció que es tracta una selenoproteïna conservada.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna GPx4:
GPx7
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna GPx7 es troba a l'scaffold GL856722.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
- La predicció d'exonerate té 1319 nucleòtids, que inclouen dos exons i un intró, i una raw score de 878.
- La predicció de genewise té 1316 nucleòtids, que inclouen dos exons i un intró, i té una puntuació de 337.69 bits.
La predicció de l'exonerate comença tres nucleòtids abans, és a dir, inclou un aminoàcid més a l'inici, però les dues prediccions acaben al mateix nucleòtid de l'scaffold, és a dir, els dos prediuen gairebé la mateix proteïna. Tot i això, la proteïna query té uns 45 aminoàcids més al principi, que no són predits amb cap dels dos programes, però si mirem la seqüència d'on s'han fet les prediccions, hem pogut veure que on hauria d'haver-hi aquesta regió hi ha un fragment de genoma que conté moltes N (nucleòtids no definits). Aquesta podria ser la causa que no s'hagi pogut realitzar la predicció.
La proteïna d' Homo sapiens no té una selenocisteïna, sinó que té una cisteïna, que es troba conservada en la nostra proteïna en el primer exó predit tan per exonerate com per genewise.
El programa secisSearch ha trobat un possible element secis a l'extrem 3' del gen, cosa que dóna suport a la predicció que podria haver-se tractat d'una selenoproteïna, que ha perdut la selenocisteïna
Considerant tota la informació anterior, es podria predir una estructura de la proteïna GPx7:
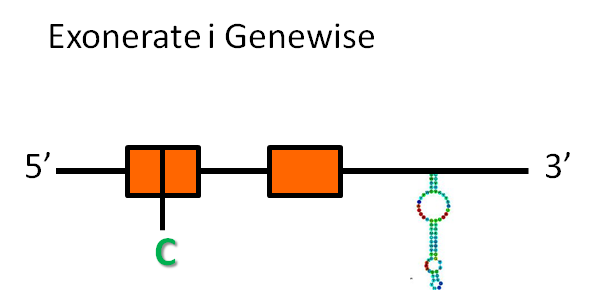
GPx8
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna GPx8 es troba a l'scaffold GL841771.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
- La predicció d'exonerate té 4918 nucleòtids, que inclouen tres exons i dos introns, i una raw score de 914.
- La predicció de genewise té 4918 nucleòtids, que inclouen tres exons i dos introns, i té una puntuació de 390.95 bits.
Les dues prediccions comencen i acaben al mateix nucleòtid de l'scaffold, és a dir, els dos prediuen la mateix proteïna.
La proteïna d'Homo sapiens no té una selenocisteïna, sinó que té una cisteïna, que es troba conservada en la nostra proteïna en el segon exó predit tan per exonerate com per genewis
El programa secisSearch ha trobat un possible element secis a l'extrem 3' del gen, cosa que dóna suport a la predicció que podria haver-se tractat d'una selenoproteïna, que ha perdut la selenocisteïna.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna GPx8:
Selenoprofiles
Per aquesta família de selenoproteïnes, el programa Selenoprofiles només ens troba quatre hits. Els quatre es corresponen amb quatre de les proteïnes trobades per nosaltres, però veiem que n’hem trobat dues de més que el Selenoprofiles ha sigut incapaç d’identificar. Cal esmentar també que en aquesta família de proteïnes, tot i que afirmem que les proteïnes identificades per Selenoprofiles coincideixen a la mateixa posició que les proteïnes trobades per nosaltres; no és del tot cert: sí que cobreixen pràcticament la mateixa extensió però varien lleugerament a l’inici i el final dels alineaments.
La proteïna que anomenem GPx1 es correspon amb la posició trobada pel hit GPx2 del Selenoprofiles. A més, l’únic element secis trobat pel Selenoprofiles és el mateix que el que hem trobat nosaltres.
La proteïna que anomenem GPx2 es correspon amb la posició trobada pel hit GPx3 del Selenoprofiles. Per aquest hit, el Selenoprofiles ens troba un element secis, que coincideix amb un dels dos secis que trobem nosaltres. Aquest element coincident és també el que està més pròxim al final de la proteïna dels dos que havíem trobat.
La proteïna que anomenem GPx3 es correspon amb la posició trobada pel hit GPx10 del Selenoprofiles. Tot i aixi, tenen extensions diferents. La identificada per nosaltres s’extén uns 10.000 nucleòtids aproximadament, mentre que la que identifica el Selenoprofiles, se n’extén uns 3.000. Respecte als elements secis, l’únic que el Selenoprofiles ha identificat és el mateix que hem trobat nosaltres
La proteïna que anomenem GPx4 es correspon amb la posició trobada pel hit GPx13 del Selenoprofiles. Tot i així, cal esmentar que la proteïna trobada pel Selenoprofiles té una llargada de 300 nucleòtids, mentre que la que hem identificat nosaltres té una llargada d’uns 2000. Nosaltres hem pogut identificar 4 secis diferents, mentre que el programa Selenoprofiles només un. Tot i així, aquest coincideix amb el secis més pròxim al final de la proteïna dels que hem trobat nosaltres.
Per les proteïnes que anomenem GPx7 i Gpx8 no s’han trobat resultats amb el Selenoprofiles.