Discussió
Família DI
Les tres proteïnes que formen aquesta família (DI1, DI2 i DI3) han donat els 3 mateixos hits en el BLAST. Utilitzant els valors d'e-value i la informació dels bits del BLAST, i els diferents alineaments del t-coffee, hem pogut assignar un hit a cada una de les proteïnes.
DI1
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna DI1 es troba a l'scaffold GL857025.1 i està codificada en el mateix sentit que l'anotació (forward).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
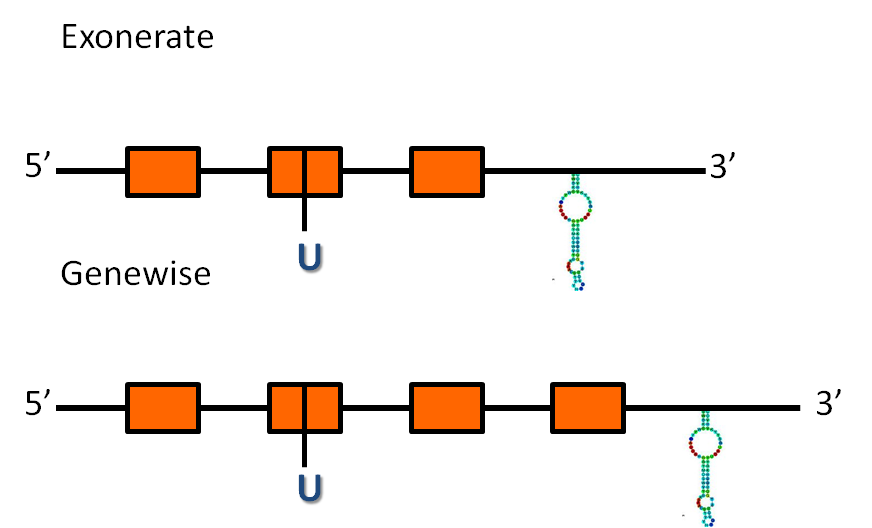
- La predicció d'exonerate té 9824 nucleòtids, que inclouen tres exons i dos introns, i una raw score de 735.
- La predicció de genewise té 14922 nucleòtids, que inclouen quatre exons i tres introns, i té una puntuació de 328.97 bits.
Les dues prediccions comencen al mateix nucleòtid de l'scaffold, però el genewise dóna una predicció més llarga i que, tal com es pot veure en els alineaments del t-coffee corresponents, cobreix una major regió de la proteïna utilitzada com a query.
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el segon exó de les proteïnes predites tan per exonerate com per genewise.
El programa secisSearch ha trobat tres possibles elements secis a l'extrem 3' del gen, a diferents distàncies del final del gen, però no tenim el coneixement per discriminar quin dels tres és el real. Tot i això, la presència d'aquests elements secis dóna suport a la predicció que es tracta una selenoproteïna conservada.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna DI1:
DI2
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna DI2 es troba a l'scaffold GL834538.1 i està codificada en el mateix sentit que l'anotació (forward).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
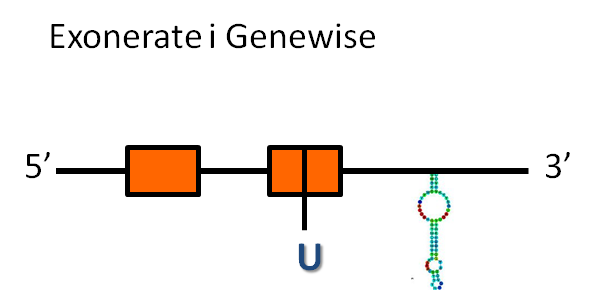
- La predicció d'exonerate té 10413 nucleòtids, que inclouen dos exons i un intró, i una raw score de 1176.
- La predicció de genewise té 10413 nucleòtids, que inclouen dos exons i un intró, i té una puntuació de 523.60 bits.
Les dues prediccions comencen i acaben al mateix nucleòtid de l'scaffold, és a dir, els dos prediuen la mateix proteïna.
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el segon exó de les proteïnes predites tan per exonerate com per genewise.
El programa secisSearch ha trobat un possible element secis a l'extrem 3' del gen, cosa que dóna suport a la predicció que es tracta una selenoproteïna conservada
Considerant tota la informació anterior, es podria predir una estructura de la proteïna DI2:
DI3
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna DI3 es troba a l'scaffold GL834587.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
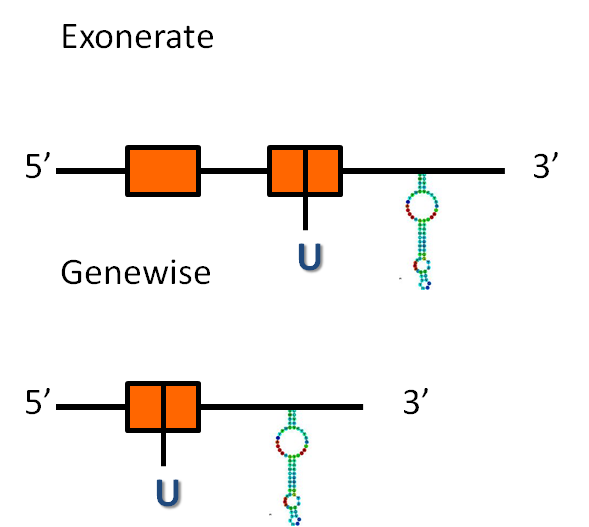
- La predicció d'exonerate té 857 nucleòtids, que inclouen dos exons i un intró, i una raw score de 863.
- La predicció de genewise té 857 nucleòtids, que inclouen només un exó, i té una puntuació de 374.50 bits.
Les dues prediccions comencen i acaben al mateix nucleòtid de l'scaffold, és a dir, els dos programes prediuen la mateix proteïna.
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el segon exó de les proteïnes predites per exonerate i a l'únic exó predit per genewise.
El programa secisSearch ha trobat un possible element secis a l'extrem 3' del gen, cosa que dóna suport a la predicció que es tracta una selenoproteïna conservada.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna DI3:
Selenoprofiles
Per aquesta família de selenoproteïnes, el Selenoprofiles ens troba tres hits diferents, i cada un ens coincideix amb la posició de les tres proteïnes anteriors trobades per nosaltres:
Per la proteïna que nosaltres anomenem DI1, el resultat de Selenoprofiles que concorda amb la mateixa posició és el hit DI.5. Aquest resultat de Selenoprofiles ens prediu només un element secis a la posició 317727 fins a la posició 317820 de l’scaffold, en canvi, nosaltres n’hem trobat tres, però estan més allunyats del final de la proteïna identificada.
Per la proteïna que nosaltres anomenem DI2, el resultat de Selenoprofiles que concorda am la mateixa posició és el hit DI.3. Per aquesta proteïna tampoc hem trobat els mateixos elements secis. El programa Selenoprofiles ha trobat un element secis més proper al final de la proteïna, a les posicions 1060987 fins 1061085 de l’scaffold, i el que hem trobat nosaltres se situa des de la posició 1063363 fins la posició 1063465.
Per la proteïna que nosaltres anomenem DI3, l'últim resultat (hit DI.1) de Selenoprofiles hi quadra perquè es situa al mateix scaffold, tot i així, tot i que els dos resultats sembla que no concordin en les mateixes posicions exactes, solapen a la mateixa regió. A més, els elements secis trobats per Selenoprofiles i nosaltres, tampoc concorden amb la mateixa posició dins de l’scaffold.