Discussió
SelO
La realització d'un BLAST ens va donar lloc a dos hits diferents, un dels quals només donava llocs a alineaments de fragments petits de la proteïna, o bé alineaments amb puntuacions molt baixes. Per tant, hem acabat assignant un sol hit a aquesta proteïna.
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna SelN es troba a l'scaffold GL861545.1 i està codificada en el mateix sentit que l'anotació (forward).
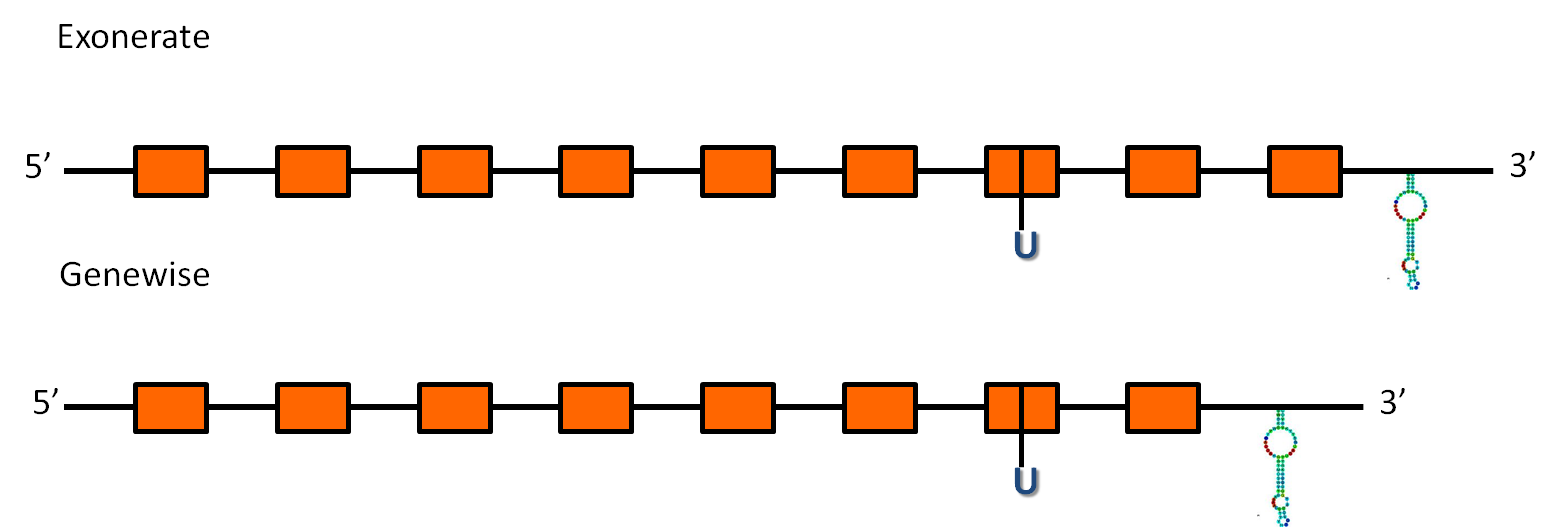
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
- La predicció d'exonerate té 4963 nucleòtids, que inclouen deu exons i nou introns, i una raw score de 1836.
- La predicció de genewise té 4954 nucleòtids, que inclouen onze exons i deu introns, i té una puntuació de 786.80 bits.
Les dues prediccions comencen al mateix nucleòtid i acaben amb nou nucleòtids de diferència. Tot i així, tenen estructures exòniques lleugerament diferents i, a més a més, cap de les dues prediccions inclou els 184 primers aminoàcids de la proteïna utilitzada com a query. Això podria ser degut a una regió amb moltes N que hi ha relativament aprop de l'inici de les prediccions, tal com hem pogut observar manualment.
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el novè exó de la proteïna predita per exonerate, però no en la predita per genewise, que s'acaba a l'aminoàcid anterior. Això podria ser degut a que genewise considera el codó TGA com a codó d'stop i, per tant, acaba la predicció i no detecta la selenocisteïna. Per tant, considerarem que aquesta proteïna predita té la selenocisteïna conservada.
El programa secisSearch ha trobat tres possibles elements secis a l'extrem 3' del gen, a diferents distàncies del final del gen, però no tenim el coneixement per discriminar quin dels tres és el real. Tot i això, la presència d'aquests elements secis dóna suport a la predicció que es tracta una selenoproteïna conservada.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna SelO:
Selenoprofiles
El Selenoprofiles ens troba 3 hits, dels quals, el que concorda amb la posició en la que hem trobat la proteïna nosaltres és el hit SelO.2. Aquest hit és l'únic dels 3 que conté una selenocisteïna. A més, per aquest mateix hit, el Selenoprofiles troba tres elements secis compatibles, a les posicions 519288, 519408 i 519606. En canvi, els elements secis que hem identificat nosaltres no correspondrien amb aquests, ja que es troben a les posicions 662034, 689536 i 757294 de l'scaffold.
Respecte als altres dos hits, un conté una cisteïna (SelO.8) i l'altre no conté una selenocisteïna alineada (SelO.10). Els dos hits corresponen a un mateix scaffold (GL861682.1), però en canvi ens troben a posicions diferents dintre d'aquest: el hit SelO.8 comença a la posició 966389 i acaba a la posició 971430; i en el hit SelO.10 comença a la posició 920157 i acaba la posició 935378 de l'scaffold. El hit SelO.8 consta de dos exons i s’estén 5041 nucleòtids. EL hit SelO.10 consta de 8 exons i s’estén 15.200 nucleòtids. Curiosament, aquest scaffold es troba en un dels hits aconseguits al fer el blast a partir de SelO humana. Aquest hit trobat al blast es va descartar ja que l'alineament que produïa indicava que la proteïna estava poc conservada.
Per cap dels dos altres hits de SelO trobats per Selenoprofiles es troben elements secis compatibles.