Discussió
SelK
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, vam obtenir només un hit amb el BLAST: la proteïna SelK es troba a l'scaffold GL841621.1 i està codificada en el mateix sentit que l'anotació (forward).
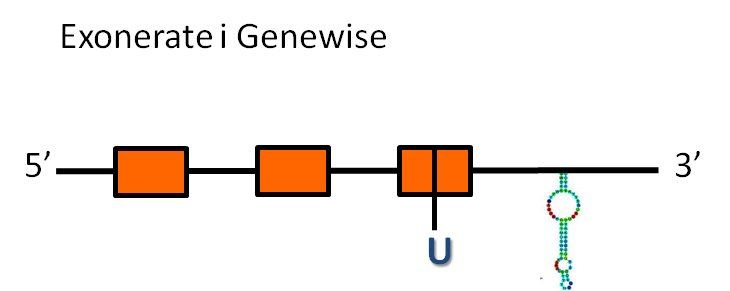
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
- La predicció d'exonerate té 1895 nucleòtids, que inclouen tres exons i dos introns, i una raw score de 355.
- La predicció de genewise té 1886 nucleòtids, que inclouen tres exons i dos introns, i té una puntuació de 139.45 bits.
Les dues prediccions comencen al mateix nucleòtid i tenen la mateixa estructura pel que fa exons i introns, però la predicció de l'exonerate és una mica més llarga pel final (té tres aminoàcids més).
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en el tercer exó de la proteïna predita per exonerate, però no en la predita per genewise, que s'acaba a l'aminoàcid anterior. Això podria ser degut a que genewise considera el codó TGA com a codó d'stop i, per tant, acaba la predicció i no detecta la selenocisteïna. Per tant, considerarem que aquesta proteïna predita té la selenocisteïna conservada.
El programa secisSearch ha trobat un possible elements secis a l'extrem 3' del gen, la presència del qual dóna suport a la predicció que es tracta una selenoproteïna conservada.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna SelK:
Selenoprofiles
El programa Selenoprofiles, només troba un hit per aquesta proteïna i ens concorda amb la posició de la proteïna trobada per nosaltres. El resultat de Selenoprofiles conté una selenocïsteina alineada, i ens troba també un element secis compatible, que és el mateix que hem trobat nosaltres.