Discussió
MsrA
A la taula es pot observar a quin scaffold es troba la proteïna, en quin sentit respecte l'anotació es transcriu, les coordenades de la proteïna predites per l'exonerate i el genewise i, finalment, les coordenades i l'energia lliure dels elements secis.
En aquest cas, la proteïna MsrA es troba a l'scaffold GL834715.1 i està codificada en el sentit contrari que l'anotació (reverse).
Hem obtingut la predicció d'una proteïna tan amb exonerate com amb genewise:
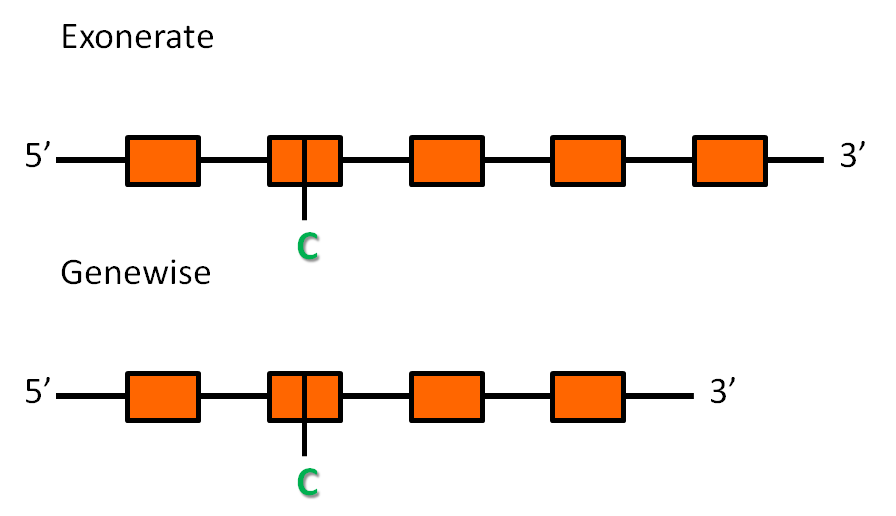
- La predicció d'exonerate té 158702 nucleòtids, que inclouen cinc exons i quatre introns, i una raw score de 645.
- La predicció de genewise té 142793 nucleòtids, que inclouen quatre exons i tres introns, i té una puntuació de 258.09 bits.
Les dues prediccions comencen al mateix nucleòtid de l'scaffold, però tenen llargades i número d'exons diferents. En aquest cas, les dues prediccions només contenen la regió central de la proteïna query, és a dir, els dos extrems de la proteïna no són predits per cap dels dos programes. Això podria ser deguts a la manca de qualitat de la seqüenciació o l'ensamblatge, però no hem aconseguit trobar el fragment predit dins l'scaffold (no sabem perquè) i, per tant, no ho podem confirmar.
La proteïna d' Homo sapiens no té una selenocisteïna, sinó que té una cisteïna, que es troba conservada en la nostra proteïna en el segon exó predit tan per exonerate com per genewise. .
El programa secisSearch no ha trobat cap element secis a l'extrem 3' de la proteïna predita, cosa que tampoc és contradictòria perquè la proteïna no té selenocisteïna i, per tant, no és necessari.
Considerant tota la informació anterior, es podria predir una estructura de la proteïna MsrA:
Selenoprofiles
Per aquesta proteïna, el programa Selenoprofiles només ens troba un hit, que coïncideix en el mateix scaffold que la proteïna trobada per nosaltres. Tot i trobar-se aproximadament a les mateixes posicions dins de l’scaffold, el nostre resultat és bastant més curt que el resultat de Selenoprofiles. La nostra proteïna va de la posició 1319115 a la 1160412 i la trobada pel Selenoprofiles va de la posició 1341667 a la 1017973. Per tant, el nostre resultat té aproximadament uns 164.000 nucleòtids menys.
El Selenoprofiles no ens troba secis, cosa que ens concordaria amb els nostres resultats.
En aquest únic hit del Selenoprofiles, ens el categoritza com a “uga containing”. Això és una mica contradictori, ja que esperaríem trobar una cisteïna, tal com hem trobat amb els nostres resultats.