Selenoproteïnes
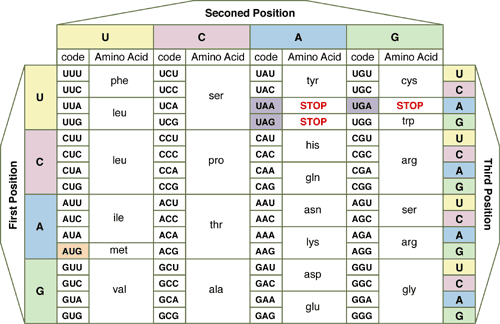
Les selenoproteïnes són proteïnes que incorporen l’aminoàcid selenocisteïna en la seva seqüència. La selenocisteïna (Sec o U), correspon a l’aminoàcid 21 i presenta la mateixa estructura que la cisteïna però conté un àtom de seleni enlloc d’un de sofre.
El seleni és un element poc abundant que constitueix un nutrient essencial en la dieta dels animals, microorganismes i alguns eucariotes. Ha demostrat ser un dels agents quimioprotectors contra el càncer més prometedors i hi ha evidències que té també un paper important en la reducció de l’expressió viral, en la prevenció de problemes cardíacs i cardiovasculars i trastorns musculars.
A més a més, la major part de selenoproteïnes són enzims redox, i per tant, poden tenir gran capacitat de protecció antioxidant. La selenocisteïna és més àcida que la cisteïna, és a dir, l’anió de la selenocisteïna és més estable que el de la cisteïna. Aquest fet implica que l’energia necessària per ionitzar-se és menor i tenen doncs una major capacitat d’atraure substàncies electròfiles. Això explica perquè pràcticament totes les selenoproteïnes descrites en eucariotes participen en reaccions d’oxidació-reducció.
Les funcions fisiològiques de les selenoproteïnes deriven doncs de les propietats que el seleni els confereix en formar part de la seva estructura.
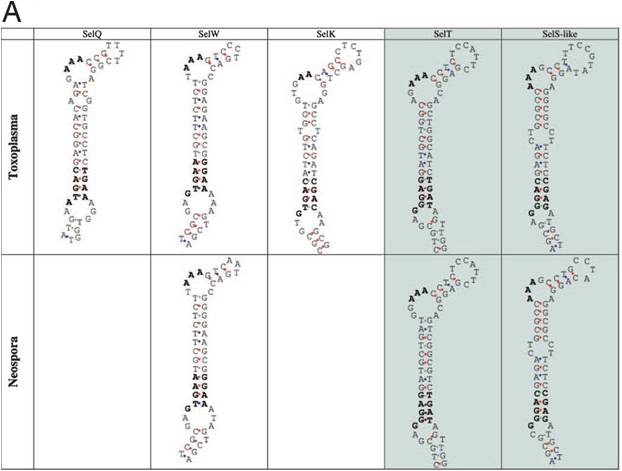
Les selenoproteïnes es van descobrir al 1973, i encara avui es segueix identificant, caracteritzant i investigant la seva estructura, metabolisme i fisiologia. S’han descrit selenoproteïnes als tres regnes Eucarya, Archaea i Prokarya i se n’ha identificat més de 30 famílies diferents.
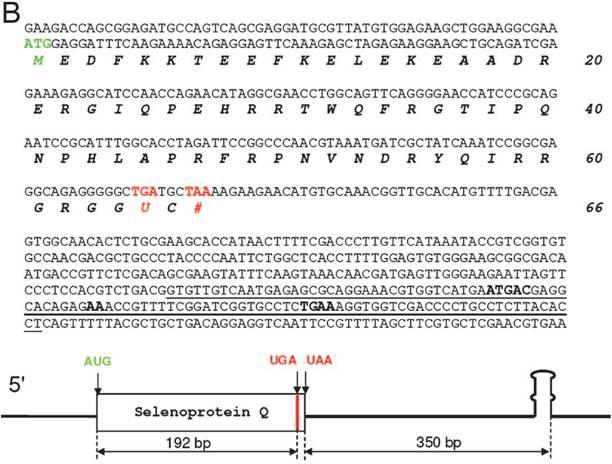
El codó que codifica per la selenocisteïna és l’UGA, que normalment codifica per a un codó STOP.