Selenoproteïnes en Plasmodium vivax
Clara Conill, Estel Enreig, Albert Gil, Carlos González, Jordina Guillén
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
SBP2
La SECIS-binding protein 2 (SBP2) és una proteïna de maquinària de selenoproteïnes. La seva funció és unir-se a l'element SECIS en la regió 3'-UTR dels mRNA que codifiquen per selenoproteïnes. Així doncs, en aquest cas no hem de trobar cap selenocisteïna, sinó que simplement hem d'anotar el gen de SBP2 en el genoma de P. vivax i comprovar que s'expressa.
RECERCA DE SBP2 EN Plasmodium vivax
D'entrada, cal advertir que la cerca d'aquesta proteïna en el genoma de P. vivax ha sigut força complicada.
Fent el TBLASTN amb la seqüència de la SBP2 humana obteníem un e-value de 10-10 en el ctg_7187, però fent posteriorment el GeneWise obteníem un alineament molt curt i poc satisfactori. Per tant, vam decidir buscar SBP2 partint d'un homòleg més proper: vam buscar la referència per a SBP2 en l'article The Plasmodium selenoproteome. Seguint l'accession number per l'NCBI de SBP2 en P.vivax (Pv_6635.phat_70) no vam arribar a la seqüencia, però seguint la de P.falciparum sí (NP_473214). Per tant, la cerca de SBP2 la vam continuar a partir de l'homòleg en P.falciparum.
El TBLASTN amb el genoma de P.vivax usant SBP2 de P.falciparum com a query va donar el següent resultat:
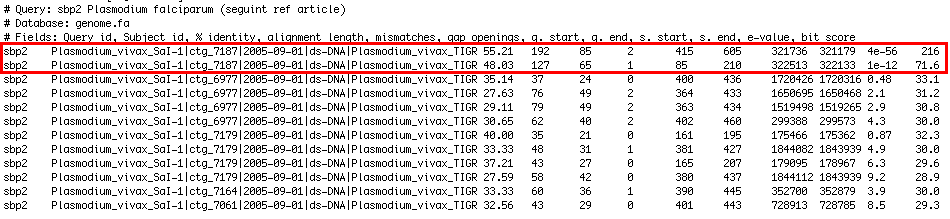 |
| Figura 1. Resultats TBLASTN |
| Descarrega't l'arxiu |
Vam usar el GeneWise per predir la seqüència de la proteïna (aquí). Llavors, vam agafar la seqüència d'aquesta proteïna predita i vam fer un TBLASTN contra els mRNA de P.vivax i vam obtenir un alineament perfecte (e-value 0.0) a partir del residu 65 de la proteïna predita amb el GeneWise:
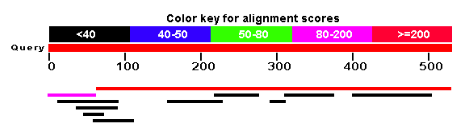 |
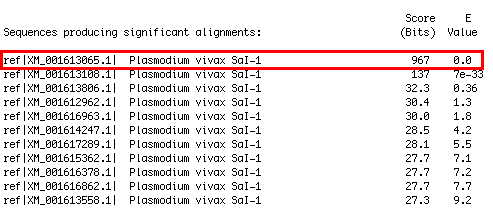 |
| Figura 2. Resultats TBLASTN mRNA |
| Descarrega't l'arxiu |
Paral.lelament, vam descarregar-nos tots els transcripts de P.vivax a partir de la web PlasmoDB, i buscàrem la seqüència del fragment d'e-value 0.0 en aquests transcripts. Així vam trobar el cDNA de la SBP2 hipotètica de P.vivax a partir d'un transcript processat (present en el ctg_7187).
Tot seguit, vam comprovar que aquesta proteïna s'expressa en P.vivax fent un TBLASTN contra els mRNA d'aquest organisme, on vam obtenir un alineament perfecte (e-value 0.0), com és lògic.
Finalment, vam predir l'estructura del gen d'SBP2 amb GeneWise i Exonerate, utilitzant la proteïna hipotètica contra el genoma de P.vivax:
Com a resum de l'estructura del gen:
# seqname source feature start end score strand frame attributes Plasmodium_vivax_SaI-1|ctg_7187|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome gene 321164 322585 2474 - .
gene_id 1 ; sequence sbp2 ; gene_orientation . Plasmodium_vivax_SaI-1|ctg_7187|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 321164 322585 . - . Plasmodium_vivax_SaI-1|ctg_7187|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 321164 322585 . - .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_7187|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome similarity 321164 322585 2474 -
. alignment_id 1 ; Query sbp2 ; Align 322586 1 1422
| Figura 3. Resultats Exonerate (.gff) |
Per tal d'acabar de corroborar que la proteïna predita és homòloga a la proteïna de P.falciparum, i que té els dominis necessaris per a la seva funció, analitzàrem la seqüència amb l'InterProScan:
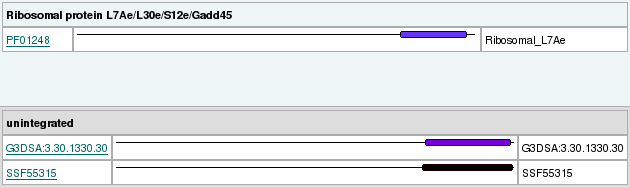 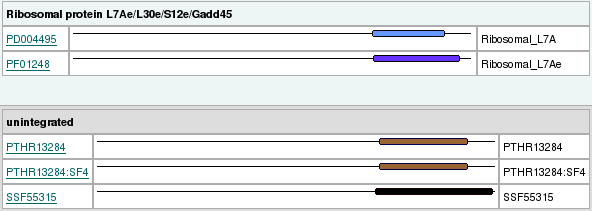 |
| Figura 4. Dominis SBP2 en P.falciparum (esquerra) i en P.vivax (dreta) |
Així doncs, l'estructura de SBP2 queda definida així:
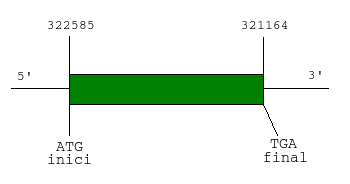 |
| Figura 5. Esquema d'SBP2 |