Selenoproteïnes en Plasmodium vivax
Clara Conill, Estel Enreig, Albert Gil, Carlos González, Jordina Guillén
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
A common problem with the databases nowadays is the incorrect annotation of selenoproteins, due to TGA codon only being considered as a stop signal. Recent characterization of selenoproteins in several organisms will permit solving this lack. The purpose of this study is to identify all the selenoproteins in the Plasmodium vivax genome, as well as all the components essential for the Sec insertion machinery. Homologs in other species have been the starting point for these sequences search. At the same time, a general screening in the whole genome has been done, which has allowed us to dismiss the existance of other possible selenoproteins with no homologs in other species. This research has lead to the localization of the four unique Plasmodium selenlenoproteins in P. vivax genome and its machinery.
Un problema actual de les bases de dades és la mala anotació de les seqüències de les selenoproteïnes, degut a que tradicionalment s'ha considerat que el codó TGA únicament codifica per un stop. La recent caracterització de les selenoproteïnes en diversos organismes permetrà resoldre aquesta mancança. La finalitat d'aquest estudi ha estat localitzar totes les selenoproteïnes presents en el genoma de Plasmodium vivax, així com tots els gens de la maquinària, que permeten la síntesi de selenocisteïna i la seva incorporació en la cadena proteica creixent. Concretament, la presència d'homòlegs en altres espècies ha servit de base per a la cerca d'aquestes seqüències. Simultàniament, hem realitzat una cerca general en tot el genoma que ens ha permès descartar l'existència d'altres possibles selenoproteïnes sense cap homòleg conegut en altres espècies. Tot aquest estudi ha possibilitat la localització de les quatre selenoproteïnes de P. vivax (exclusives del gènere Plasmodium) i la maquinària necessària per la seva síntesi.
Tornar a l'índexL'objectiu d'aquesta investigació és determinar la presència de selenoproteïnes en el genoma de Plasmodium vivax mitjançant mètodes computacionals.
A continuació presentem informació rellevant sobre l'espècie d'estudi i les selenoproteïnes.
Plasmodium vivax
Plasmodium vivax és una espècie del gènere Plasmodium. Concretament segueix la següent classificació científica:
| Domini | Eucarya |
|---|---|
| Regne | Protista |
| Filum | Apicomplexa |
| Classe | Aconoidasida |
| Ordre | Haemosporida |
| Família | Plasmodiidae |
| Gènere | Plasmodium |
| Espècie | Plasmodium vivax |
És un dels cuatre paràsits causants de la malària en humans (P.falciparum, P.vivax, P.malariae, P.ovale). Tot i que P.falciparum és el causant de la majoria de morts per aquesta malaltia, P.vivax és l'espècie més prevalent. Malgrat no ser tan mortal, causa febre, calfreds, sudoració, mal de cap i ossos i un decaïment general, simptomatologia que té una important repercusió en la qualitat de vida del malalt.
El cicle de vida de P.vivax és semblant al de les altres tres espècies que causen malària. A grans trets podem dir que el vector de la malaltia és la femella del mosquit Anopheles, on el paràsit es reprodueix sexualment i produeix sporozoits, els quals es transmetran per la saliva a l'hoste durant la picada. Els hostes són diferents vertebrats, en els que el Plasmodium es reprodueix asexualment i infecta les cèl·lules hepàtiques i sanguínies, causant els símptomes característics de la malaltia. Un fet important és que en les cèl·lules sanguínies es produeixen gametòcits que seràn captats pel mosquit quan es produeixi una picada, començant així un nou cicle.
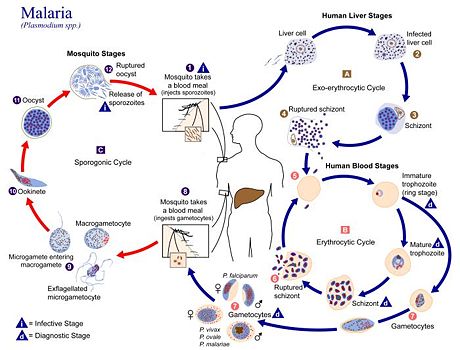
|
| Figura 1.Cicle de vida de Plasmodium |
Les cuatre espècies de Plasmodium que causen malària en humans, igual que altres espècies del gènere que afecten altres animals, presenten un genoma amb 14 cromosomes, alguns dels quals han estat seqüenciats recentment. Aquest és el cas del genoma de Plasmodium vivax, sobre el qual treballarem.
SELENOPROTEÏNES
Les selenoproteïnes són proteïnes que contenen l'aminoàcid selenocisteïna (Sec), un anàleg de la cisteïna (Cys) que presenta seleni enlloc de sofre. Es tracta de l'aminoàcid número 21, codificat pel codó TGA, el qual normalment significa la finalització de la traducció. El fet de que sigui un codó stop el que indiqui la incorporació de Sec fa difícil la detecció de selenoproteïnes mitjançant els mètodes de predicció de gens convencionals, motiu pel qual moltes selenoproteïnes es troben mal anotades en els genomes. El codó TGA codificarà per Sec sempre i quan hi hagi un element SECIS (Selenocysteine insertion sequence) a l'extrem 3' UTR del mRNA. Els elements SECIS no presenten un patró de seqüència gaire conservat, però tots ells formen una estructura terciària característica (hairpin-like structure) que fa possible la inserció de Sec.
Gràcies a la seqüenciació de molts genomes i a l'avenç de la bioinformàtica s'estan identificant selenoproteïnes en molts organismes dels tres dominis de la vida: Bacteria, Archaea i Eukarya.
 |
| Figura 2.Estructura general d'una selenoproteïna |
Tornar a l'índex
Per a la realització d'aquesta investigació hem seguit un protocol establert anteriorment, el qual es mostra a continuació. Cal tenir present que es tracta d'un protocol general que ha hagut de ser modificat lleugerament degut a problemes sorgits durant l'avenç del treball.
En l'apartat de Resultats i Discussió estan explicats els passos addicionals que ha calgut fer en cada cas.
PROTOCOL PER LA CERCA DE SELENOPROTEÏNES EN UN GENOMA
1. Obtenció de la seqüència genòmica de l'espècie problema
Primer de tot buscarem la nostra seqüència en una de les bases de dades (les més usades són l'Ensembl, NCBI i Santa Cruz). Tot i així hi ha seqüències de microorganismes que no es troben disponibles en aquestes bases de dades, pel que les haurem de buscar en altres de menys utilitzades. Un cop obtingudes les seqüències les guardarem en format FASTA.
2. Caracterització de l'espècie i cerca d'estudis sobre selenoproteïnes en aquesta
Buscarem informació sobre l'espècie d'estudi i mirarem si hi ha estudis previs sobre selenoproteïnes per tal d'orientar la nostra investigació.
Cerca de selenoproteïnes ja conegudes en el genoma:
3. Cerca de selenoproteïnes en el SelenoDB
Entrarem en la base de dades de selenoproteïnes SelenoDB, on sabem que hi ha disponible la seqüència tan genòmica com aminoacídica de les selenoproteïnes eucariotes descobertes fins ara. Aquesta pàgina web també proporciona informació sobre els elements Secis i molècules relacionades. A més, separa les proteïnes segons l'espècie, i dins de cada espècie les classifica segons si són selenoproteïnes, homòlegs amb cisteïna, homòlegs amb algun altre aminoàcid o maquinària relacionada amb les selenoproteïnes.
Com que l'espècie humana és la més estudiada i la que té més selenoproteïnes caracteritzades, inicialment utilitzarem aquestes seqüències, tot i que la cerca es pot ampliar si és convenient.
Ens interessa tenir aquesta informació per a buscar seqüències homòlogues en el nostre genoma. En aquest cas, agafarem la seqüència aminoacídica per a posteriorment fer el Blast que considerem més adequat.
Per altra banda, durant la recerca d'informació sobre el nostre organisme hem trobat un article al Pubmed titulat 'The Plasmodium selenoproteome' (Nucleic Acids Research, 2006, Vol. 34, No. 2), que s'ha convertit sens dubte en l'article de referència a l'hora de realitzar el nostre treball, perquè ens ha proporcionat molta informació rellevant. Els autors van fer un estudi per caracteritzar les selenoproteïnes en protozous apicomplexes, ja que van constatar que en eucariotes només havien estat estudiats els selenoproteomes d'animals i algues. En aquest article s'explica que els autors han identificat quatre gens codificants per selenoproteïnes (de Sel 1 a Sel 4), que són específics del gènere Plasmodium i que no presenten cap homòleg detectable en altres espècies. Sabent els resultats d'aquest estudi, també buscarem aquests gens en el genoma de P.vivax, lògicament partint dels homòlegs de P.falciparum, ja que en la base de dades d'H.sapiens de SelenoDB no hi seran.
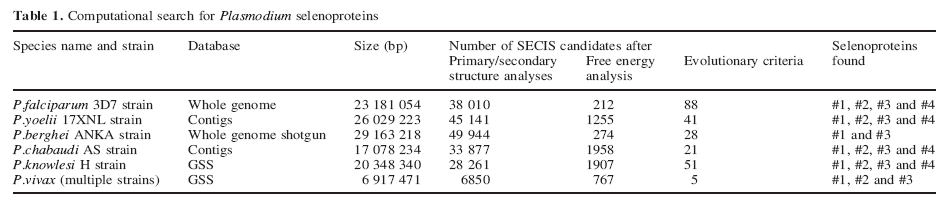 |
En aquesta taula, extreta de l'article, veiem que en P.vivax els autors només han identificat Sel 1, Sel 2 i Sel 3. Pel que fa a Sel 4, intentarem buscar-la igualment, perquè cal tenir en compte que l'article és del 2006, de manera que potser nosaltres tenim accés a bases de dades més actualitzades.
Un altre punt clau de l'article és que els autors afirmen que en Plasmodium vivax no hi ha homòlegs de selenoproteïnes conegudes que es mantinguin com a tal. Tot i això, nosaltres considerem important buscar-les igualment (mitjançant el protocol descrit a continuació), per tal de verificar personalment la validesa d'aquesta afirmació.
Per descarregar l'article, clica aquí4. Comparació de les seqüències aminoacídiques obtingudes amb el genoma d'estudi: TBLASTN
Un cop tenim les seqüències d'aminoàcids de les selenoproteïnes humanes, utilitzarem el programa BLAST (Basic Local Alignment Search Tool), el qual ens permetrà buscar les seqüències homòlogues en el nostre organisme. Hi ha diferents tipus de BLAST, depenent de què es vulgui alinear. En aquest cas utilitzarem TBLASTN, que ens compara una query de proteïna contra seqüències de nucleòtids de la base de dades. Considerem que és el més útil perquè evitarem falsos mismatches produïts pel biaix en l'ús de codons.
De tots els paràmetres del BLAST que es poden modificar a l'hora de fer alineaments, n'hi ha un que ens interessa especialment: l'e-value. Aquest és el nombre esperat d'alineaments que podem obtenir amb un score igual o superior per atzar en un alineament múltiple. D'aquesta manera, com menor sigui l'e-value, més significant és l'alineament. En el nostre cas, ens interessa una gran significància i, per tant, delimitarem un e-value de 10-10.
El BLAST el podrem utilitzar des de la pàgina web, des de la finestra terminal del Linux o amb la Blastmachine (ens permetria córrer totes les seqüències d'un sol cop) segons ens convingui.
5. Selecció dels alineaments significatius i extracció del fragment de la seqüència genòmica d'interès
De tots els alineaments ens quedarem amb aquells que tinguin un e-value significatiu.
A partir d'aquí utilitzarem la seqüència genòmica dels alineaments que considerem vàlids i l'allargarem uns 3000pb per tal d'englobar l'element Secis. Creiem que la manera més pràctica de fer-ho és a través de la finestra terminal del Linux.
6. Deducció de l'estructura exònica del nostre fragment de genoma: GeneWise
Per a poder conèixer l'estructura exònica haurem de comparar el nostre fragment de genoma amb la selenoproteïna que estem estudiant. Això ho podem fer mitjançant una advanced query del programa GeneWise , el qual dedueix els exons i introns de la seqüència genòmica. En aquesta predicció seria ideal que coincidís la selenocisteïna amb un codó stop o amb una cisteïna del nostre genoma. A partir del resultat del GeneWise podrem veure si realment els codons que codifiquen selenocisteïna o cisteïna es troben dins un exó, la qual cosa ens indicaria que estem davant una selenoproteïna o un hoòleg d'aquesta.
7. Comprovació de l'existència de les selenoproteïnes predites: Exonerate
Per a comprovar que la predicció de les selenoproteïnes és correcta farem servir el programa Exonerate a través de la finestra terminal del Linux. Gràcies a aquest programa farem un alineament entre la seqüència protèica de la selenoproteïna predita i el contig corresponent del genoma de l'espècie en estudi. Així, obtindrem un resultat en format gff que ens informarà sobre l'estructura genètica de la selenoproteïna trobada: l'inici i el final del gen, els llocs d'splicing, la pauta de lectura...
8. Cerca d'elements Secis en les selenoproteïnes trobades
Un cop trobades les possibles selenoproteïnes utilitzarem el programa SECISearch per a trobar l'element SECIS en la regió 3' de la seqüència (recordem que abans l'hem allargat amb aquest propòsit). Aquest programa ens permetrà no tan sols localitzar dominis potencials d'elements SECIS sinó que també ens informarà de l'estabilitat termodinàmica d'aquests. De fet, només acceptarem aquells SECIS que presentin un score major de 15 (que es considera el llindar de validesa)
Cerca de noves selenoproteïnes en el genoma:
Tant si hem trobat selenoproteïnes homòlogues en el nostre genoma com si volem extendre més la nostra cerca, podem buscar la presència de noves selenoproteïnes en el nostre genoma seguint els següents passos:
9. Cerca d'elements SECIS
Podem intentar determinar elements SECIS en tot el nostre genoma gràcies al programa SECISearch. Seleccionarem aquelles prediccions d'elements SECIS amb un score superior a 15 i extraurem el fragment de seqüència del genoma corresponent per tal d'englobar el possible gen.
També podríem emprar el programa PatScan per a aquesta cerca, però en aquest cas hauriem d'utilitzar també el programa RNAfold per l'estudi de l'estabilitat termodinàmica.
10. Limitació dels SECIS a regions codificants per possibles selenoproteïnes
Com que del punt anterior esperem obtenir un nombre considerable d'elements SECIS potencials hem de limitar la nostra cerca condicionant aquests elements SECIS a la presència d'una seqüència codificant prèvia amb un codó per a selenocisteïna. Amb el programa geneid es poden determinar aquestes seqüències. Hem de tenir en compte que haurem d'utilitzar una versió modificada d'aquest programa que tingui en compte que un codó stop TGA pot ser codificant.
11. Anàlisis posteriors necessaris
Del procés anteriorment descrit ja n'obtindríem un nombre bastant limitat de selenoproteïnes. És en aquest punt on cal comparar els possibles candidats amb altres gens ja coneguts i descartar aquelles seqüències que presentin incompatibilitats amb les estructures generals dels EST i/o mRNA. A més a més cal tenir en compte la viabilitat de les estructures secundàries de les selenoproteïnes predites. Amb tots els passos anteriors esperaríem una reducció encara més gran de les selenoproteïnes candidates. Per tal de comprovar que el gen predit codifica per a una selenoproteïna s'hauria de fer un BLAST entre la seqüència predita i el genoma d'un organisme proper per a veure si hi ha conservació més enllà del codó TGA que creiem que codifica per selenocisteïna. Finalment, caldria verificar que la seqüència de les selenoproteïnes que hem trobat no és cap de les que ja havien estat predites anteriorment.
Per acabar, només falta corroborar l'homologia entre els dominis dels dos homòlegs (proteïna de partida vs. proteïna trobada en el genoma d'estudi). Aquest últim pas es farà gràcies al programa InterProScan.
 |
| Figura 3.Esquema del protocol |
Tornar a l'índex
La taula que es mostra a continuació recull els resultats, tan positius com negatius, que hem obtingut de la cerca de selenoproteïnes en el genoma de Plasmodium vivax. En la primera columna hi ha representades totes les selenoproteïnes que hem estudiat, així com la maquinària necessària per a la seva síntesi. En la segona columna s'especifica l'organisme del qual hem obtingut les seqüències utilitzades per a buscar homòlegs en Plasmodium vivax. Finalment, els resultats de la nostra investigació estan plasmats en la tercera columna de la taula.
Per tal d'accedir a una informació més detallada dels resultats cal clicar sobre el link d'interès. S'ha de tenir en compte que, en cas de tractar-se d'una família de selenoproteïnes, el link dirigeix a una de les selenoproteïnes que formen part de la família en qüestió.
* Totes les selenoproteïnes no significatives són aquelles en les quals els e-values són superiors a 10-10. Com s'ha comentat a materials i mètodes, per sobre d'aquest valor hem considerat que la probabilitat de trobar un alineament com el que tenim per atzar és massa elevada, i per tant, la nostra predicció podria ser errònia.
| Homòleg patró | Plasmodium vivax | |
| eEFSec | Homo sapiens | SI |
| SECp43 | Drosophila melanogaster | SI |
| SLA/LP | Drosophila melanogaster | SI |
| SPS | Homo sapiens | SI |
| SBP2 | Plasmodium falciparum | SI |
| tRNAsec | Plasmodium falciparum | SI |
| TR | Homo sapiens | SI |
| Sel 1 | Plasmodium falciparum | SI |
| Sel 2 | Plasmodium falciparum | SI |
| Sel 3 | Plasmodium falciparum | SI |
| Sel 4 | Plasmodium falciparum | SI |
| GPx | Homo sapiens | No significatiu* |
| DI | Homo sapiens | No significatiu* |
| Sel 15 | Homo sapiens | No significatiu* |
| Sel H | Homo sapiens | No significatiu* |
| Sel I | Homo sapiens | No significatiu* |
| Sel K | Homo sapiens | No significatiu* |
| Sel M | Homo sapiens | No significatiu* |
| Sel N | Homo sapiens | No significatiu* |
| Sel O | Homo sapiens | No significatiu* |
| Sel P | Homo sapiens | No significatiu* |
| Sel R | Homo sapiens | No significatiu* |
| Sel S | Homo sapiens | No significatiu* |
| Sel T | Homo sapiens | No significatiu* |
| Sel U | Homo sapiens | No significatiu* |
| Sel V | Homo sapiens | No significatiu* |
| Sel W | Homo sapiens | No significatiu* |
| MsrA | Homo sapiens | No significatiu* |
Finalment, pel que fa a la recerca de selenoproteïnes no descrites fins ara, vàrem realitzar un screening general a nivell del genoma complet, els resultats del qual es poden veure aquí.
Tornar a l'índex
Principalment, el treball que hem realitzat ens ha permès confirmar i ampliar els aspectes essencials de l'article de referència (Lobanov AV et al.) sobre el selenoproteoma de Plasmodium vivax , a més d'aprofundir en el seu estudi.
En primer lloc, hem verificat l'afirmació de l'article que no existeixen homòlegs de selenoproteïnes caracteritzades en el SelenoDB que es mantinguin com a tal en P. vivax. Així doncs, en dur a terme el protocol en les selenoproteïnes humanes no n'hi ha cap que haguem trobat conservada com a selenoproteïna. De fet, pel que fa a homòlegs amb algun altre aminoàcid enlloc de la U només n'hem identificat una en P. vivax (concretament, la TR1).
A més, hem confirmat la presència de Sel 1, Sel 2 i Sel 3 en el genoma de P.vivax, que ja estaven descrites en l'article. Fins i tot, hem localitzat Sel 4, que ja estava caracteritzada en altres genomes del gènere Plasmodium però no en P. vivax. Cal tenir en compte, però, que l'article de referència és de l'agost del 2005, any en el qual només disposaven d'una base de dades GSS del genoma de P. vivax en PlasmoDB. La GSS és una base de dades similar a les de EST, tot i que està formada per seqüències genòmiques enlloc de cDNA (mRNA). Per tant, cal considerar que els fenòmens d'splicing no hi estan contemplats. Els inconvenients principals d'aquest tipus de bases de dades és que la informació recollida prové de moltes fonts diferents (Alu PCR sequences o transposon-tagged sequences, entre d'altres), no està completa, pot tenir errors o talls enmig d'una seqüència d'interès, etc. En canvi, nosaltres hem disposat d'un conjunt de contig que representen tot el genoma de P. vivax, que és una informació que va ser incorporada en la web de PlasmoDB durant el setembre del 2007. Lògicament, el fet de disposar de tot el genoma, sencer i ordenat en contigs, facilita enormement la caracterització de gens, fent obsoleta la base de dades de GSS. Per tant, en disposar d'una base de dades molt més adequada hem pogut localitzar el gen de Sel 4 en P. vivax, que en l'article no estava descrit (veure punt 3 de l'apartat Materials i Mètodes).
Per altra banda, també hem confirmat la presència de la maquinària de selenoproteïnes en P. vivax (en trobar homòlegs de gens coneguts de maquinària d'H. sapiens, D. melanogaster i P. falciparum), tal i com ja s'afirmava en l'article de referència. Aquest fet també ens serveix per a verificar la presència real de selenoproteïnes en P. vivax, ja que els gens de maquinària són imprescindibles per a la síntesi d'aquestes.
També ha estat realitzada una cerca global d'elements SECIS a tot el genoma (posant com a query tots els contigs). Tot i que el programa SECISearch ens ha predit una gran quantitat d'elements SECIS només un petit nombre d'aquests ha obtingut puntuacions significatives. Concretament, només hem obtingut scores majors de 15 en els elements SECIS corresponents a les quatre selenoproteïnes identificades per homologia i en una zona del genoma contigua a la proteïna subtilisin-like protease. Davant la possibilitat de que la subtilisina fos una selenoproteïna mal anotada, hem realitzat un estudi de la seva seqüència. Finalment, hem pogut descartar que aquesta proteïna sigui una selenoproteïna degut a que en el seu transcrit no hem estat capaços de trobar ni elements SECIS hipotètics prou propers ni aminoàcids conservats després del codó TGA.
Com s'ha pogut veure en els resultats de Sel 1 i Sel 3, vam detectar un parell d'errors en la seqüència aminoacídica d'aquestes dues selenoproteïnes annotada a PlasmoDB. Caldria, doncs, revisar aquestes seqüències i corregir tals errors en cas de ser confirmats.
Pel que fa als reptes de futur, creiem que és molt important continuar la recerca en aquesta direcció.
Per començar, caldria validar experimentalment la presència de selenoproteïnes en P. vivax, mitjançant les proves de marcatge amb 75Se. També seria interessant investigar les possibles funcions de les selenoproteïnes de Plasmodium, perquè de moment no s'han pogut inferir. Els autors de l'article ja van realitzar cerques de BLAST exhaustives, sense trobar homòlegs en altres espècies (a partir dels quals haguéssin pogut deduir la seva funció). A més, nosaltres hem buscat els dominis d'aquestes proteïnes mitjançant InterProScan i, tot i que els dominis entre cadascuna de les selenoproteïnes específiques i l'homòleg del qual hem partit comparteixen els dominis, no hi hem trobat cap domini que ens permeti hipotetitzar sobre la funció. De fet, potser l'estudi de la funció d'aquestes proteïnes es podria enfocar experimentalment (per exemple, mitjançant la realització de KO). Un altre punt rellevant seria fer un alineament múltiple de totes les selenoproteïnes de les diferents espècies de Plasmodium, i d'altres Apicomplexes en general, per tal de comparar-ne l'homologia i poder generar un arbre filogenètic (per exemple, usant el programa Mega3), per estudiar-ne la història evolutiva.
Finalment, el que caldria a llarg termini seria identificar les selenoproteïnes de totes les espècies (en Bacteria, Archaea i Eukarya), per tal de poder corregir-ne totes aquelles que actualment es troben mal anotades en les grans bases de dades. Així, la comunitat científica podria disposar, per fi, de bases de dades que consideren la presència d'aquestes selenoproteïnes.
Tornar a l'índexCastellano S, Morozova N, Morey M, Berry MJ, Serras F, Guigó R et al. In silico identification of novel selenoproteins in the Drosophila melanogaster genome. EMBO Rep. 2001; 2(8):697-702.
Lobanov AV, Delgado C, Rahlfs S, Novoselov SV, Kryukov GV, Gromer S et al. The Plasmodium selenoproteome. Nucleic Acids Research. 2006; 34 (2): 496-505.
Papp LV, Lu J, Holmgren A, Khanna KK. From selenium to selenoproteins: synthesis, identity and they role in human health. Antioxid Redox Signal. 2007; 9(7):775-806.
Xu XM, Mix H, Carlson BA, Grabowski PJ, Gladyshev VN, Berry MJ, Hatfield DL. Evidence for direct roles of two additional factors, SECp43 and soluble liver antigen, in the selenoprotein synthesis machinery. J Biol Chem. 2005; 280(50):41568-75.
Tornar a l'índexClara Conill: clara.conill01@campus.upf.edu
Estel Enreig: estel.enreig01@campus.upf.edu
Albert Gil: albert.gil01@campus.upf.edu
Carlos González: carlos.gonzalez01@campus.upf.edu
Jordina Guillén: jordina.guillen01@campus.upf.edu
Tornar a l'índex