

Anàlisi
Estudi de la distribució de caixes que corresponen
a llocs de reconeixement del factor de transcripció CREB
Taula de continguts:
Introducció
La proteïna CREB (cyclic AMP-responsive element-binding
protein) és un factor de transcripció clau que estimula
l’expressió de nombrosos gens en resposta a
factors de creixement, hormones, neurotransmissors, fluxes
iònics i senyals d’estrès. Aquests elements extracel·lulars
activarien un conjunt de missatgers secundaris que transduirien la senyal via rutes quinasa
fins l’activació de CREB, que té localització nuclear. Aquesta
activació es porta a terme mitjançant la fosforilació de la serina 133
d’aquest factor. Tant CREB com els seus paràlegs ATF1 i CREM actuen formant
dímers i activant la transcripció de gens corresponents a un ampli ventall de
processos biològics, incloent-hi el metabolisme glucídic i la supervivència
cel·lular i complexes funcions neuronals com la memòria i l’aprenentatge.
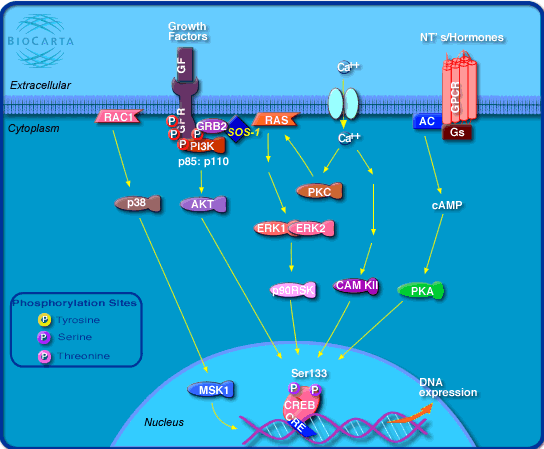 Figura 1: vies d'activació de CREB
Figura 1: vies d'activació de CREB
Els membres de la familia CREB s’uneixen al DNA per mitjà d’un domini
proteic bàsic anomenat leucine zipper (bZIP). Aquest motiu és
capaç de reconèixer una seqüència palindròmica curta present
en el genoma: l’element CRE (cAMP response element).
Aquesta seqüència es localitza freqüentment a la regió promotora proximal
al lloc d’inici de transcripció (TSS: transcription starting site).
Estudis realitzats sobre nombrosos promotors que responen a AMPc han permès la
identificació d’un palíndrom consens -TGACGTCA- que és reconegut pel
dímer CREB amb alta afinitat.
Altres estudis indiquen que CREB està unit constitutivament als motius CRE fins i tot en
absència d’agonistes. Aquesta unió es produeix molt sovint molt a prop de
les capces TATA del mateix promotor. Per aquest motiu, la unió de CREB al
promotor sembla estimular interaccions cooperatives amb altres factors de transcripció
per tal de facilitar el reclutament dels complexes RNA polimerasa II.
Tornar a Taula de continguts
Objectius
L’objectiu del nostre estudi és analitzar la possible cooperació
funcional entre els factors d’unió als motius CRE i TATA del DNA. Volem
estudiar aquesta interacció putativa comparant la distribució dels dos motius
en diferents seqüències corresponents a diferents regions gèniques humanes.
Tornar a Taula de continguts
Materials i Mètodes
Per realitzar el nostre estudi hem utilitzat dos fitxers que contenen seqüències
corresponents a diferents regions gèniques en format FASTA:
- hs_EPD.fasta (seqüències promotores): aquest fitxer conté
seqüències de gens humans corresponents al promotor proximal de cada gen.
Inclou des de la posició -674 respecte a l'inici de transcripció (TSS)
fins a la +325.
- hs_cod.fasta (seqüències codofocants): aquest fitxer conté
seqüències codificants del mateix tamany(984 pb) a partir del codó
d'inici de la traducció ATG.
Per tal d’analitzar la distribució dels motius CRE i TATA sobre aquestes
seqüències, hem utilitzat dues expressions regulars que defineixen la
seqüència consens de cadascun dels motius. Degut a que els elements de
resposta són seqüències relativament curtes, hem precisat les expressions
per tal de limitar la búsqueda sobre les seqüències. Així, les
expressions regulars utilitzades en format Perl són les següents:
- TATAAAA per al motiu TATA.
- TGACGT[AC][AT] per al motiu CRE.
Hem realitzat la búsqueda d’aquests motius sobre les seqüències
mitjançant el programa motius.pl, descrit detalladament a la secció PROGRAMA. Els
resultats obtinguts al fitxer de sortida s’han processat i representat en forma
d’histogrames a partir del programa SPSS v11.0.
Tornar a Taula de continguts
Resultats
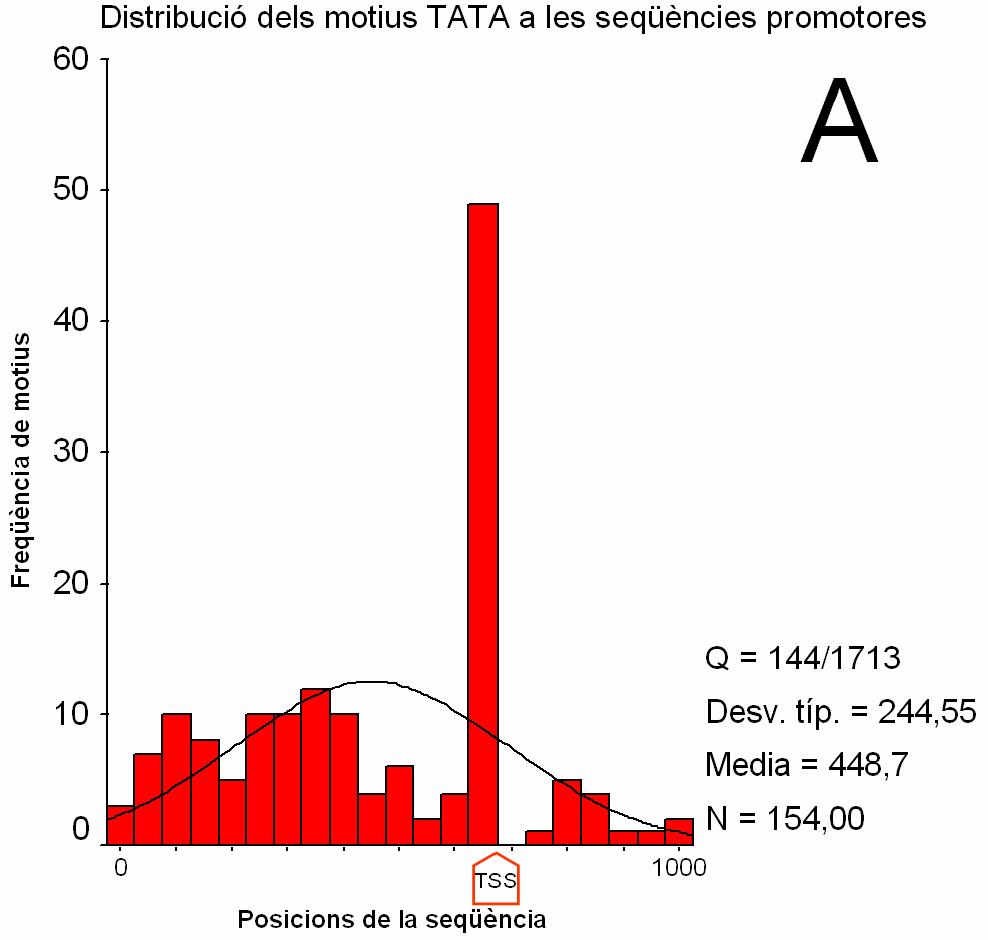 |
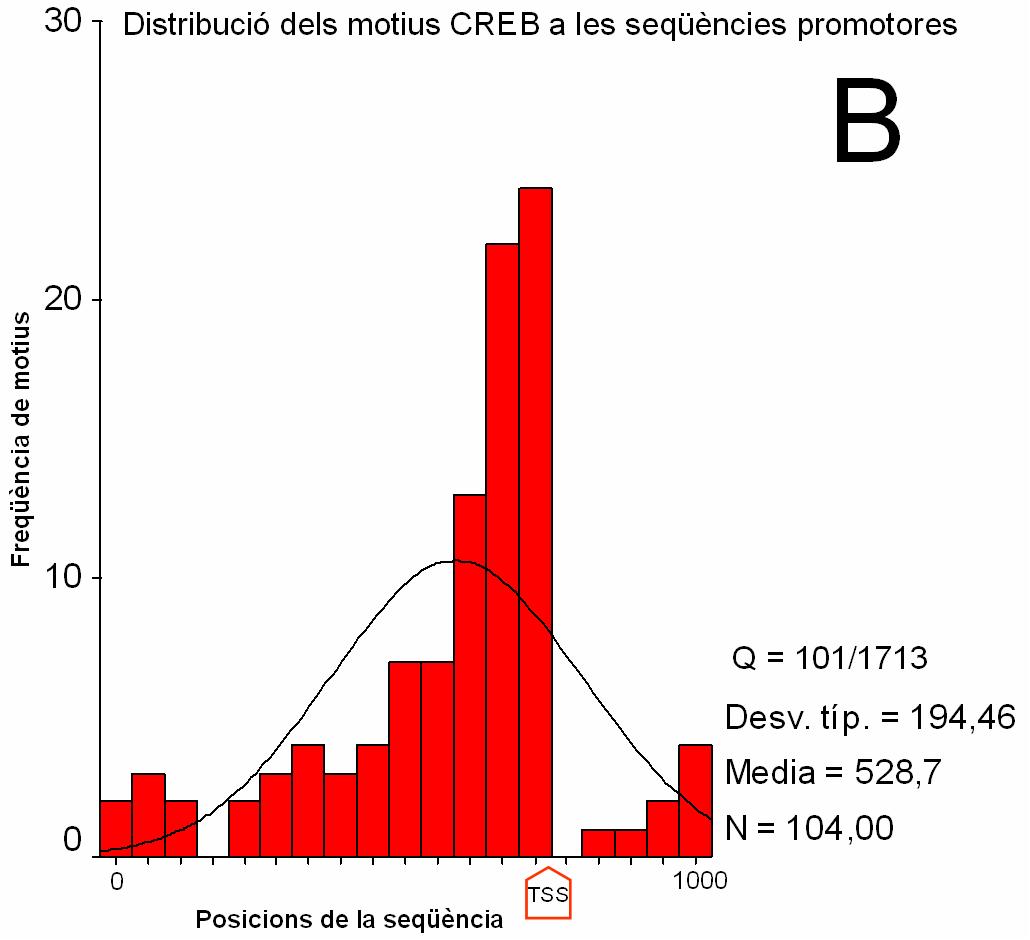 |
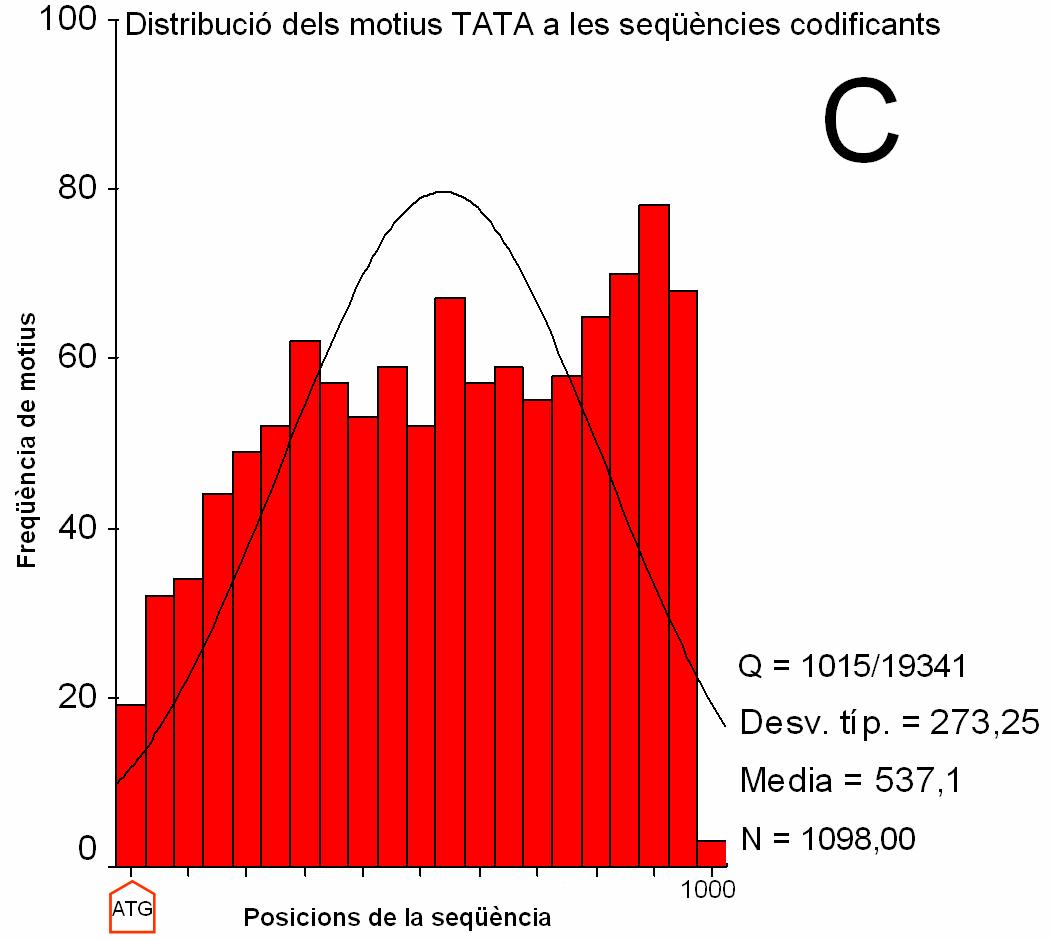 |
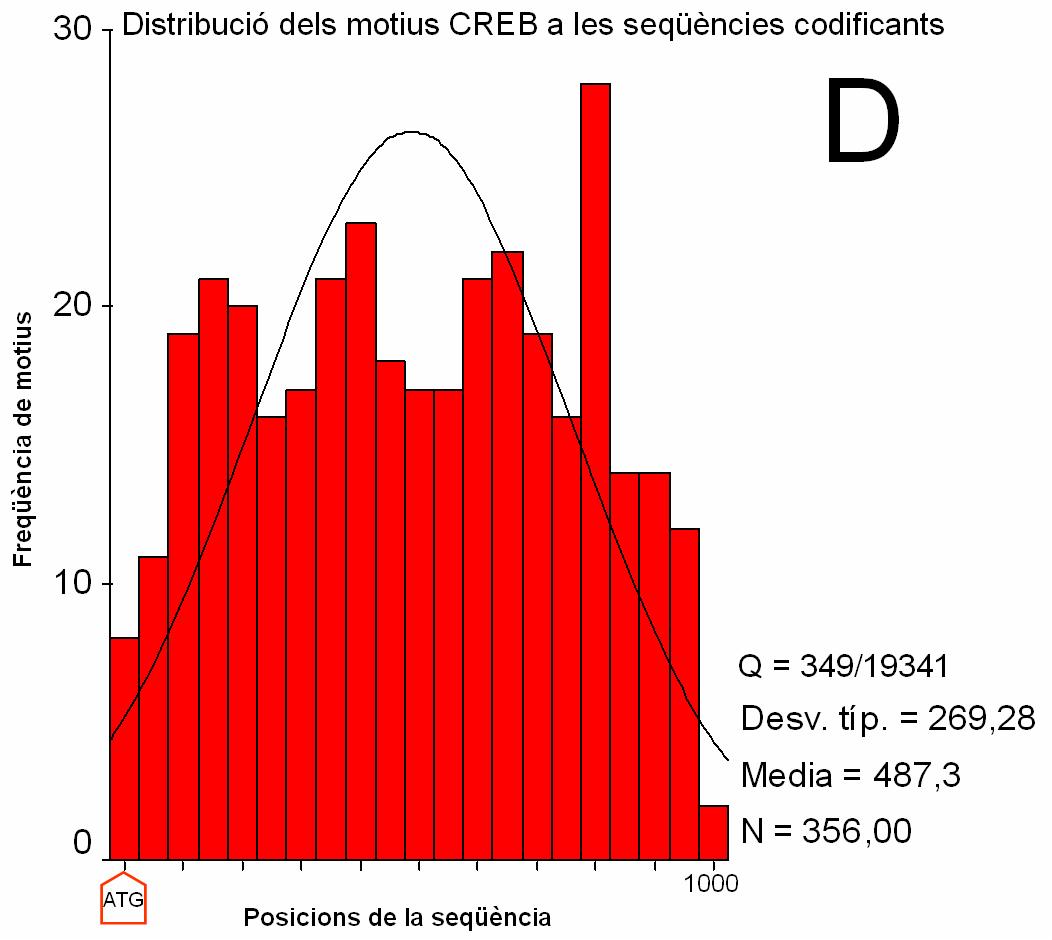 |
Figura 2.
Distribució dels motius CRE i TATA en seqüències
promotores i codificants (clica per veure els gràfics ampliats).
(A) i (B) Histograma que recull la freqüència de match
registrada a cada intèrval de posició de les
seqüències promotores. Els intèrvals són de
100 nucleòtids en A i 5 nucleòtids en B. S’indica amb una
fletxa la posició del TSS. La posició 0 a l’eix d’abcises
correspon al nucleòtid –674 respecte el TSS. La posició
1000 correspon al nucleòtid +325.
(C) i (D) Histograma que recull la freqüència de match
registrada a cada intèrval de posició de les
seqüències codificants. Els intèrvals són de
100 nucleòtids. La posició 0 correspon a l’ATG (indicat
amb una fletxa).
Q: nombre de seqüències amb match respecte el total de seqüències.
Desv. típ: desviació estàndard
Media: mitjana
N: nombre total de match
|
En el cas de les seqüències promotores, el nombre total de
match ocorreguts sobre les seqüències és prou
coincident en ambdós motius. Analitzant visualment el llistat
registrat al fitxer de sortida trobem que la major part de les
seqüències promotores que contenen TATA també
contenen CRE (fitxers de sortida de
A i
B).
Els resultats suggereixen una tendència dels motius CRE i TATA a
distribuir-se en regions coincidents. La figura 2A mostra una clara
prevalència del motiu TATA a localitzar-se a la regió
inmediatament anterior al lloc d’iniciació de
transcripció. Aquest fet coincideix amb la funció
d’aquest motiu relacionada amb el reclutament de la maquinaria de
transcripció. Addicionalment, a la figura 2B s’aprecia com la
distribució predominant del motiu CRE es dóna a les 100
posicions precedents al TSS. Tenim, per tant, que tant les capses TATA
com les capses CRE es localitzen molt properes al lloc d’iniciació
de transcripció, i que en la majoria dels casos apareixen alhora.
En l’anàlisi realitzat sobre les seqüències
codificants, trobem que el nombre de match és molt superior que
en el cas anterior, especialment amb el motiu TATA (apareixen match
vàries vegades per seqüència). A més, no
és possible establir un patró de distribució dels
motius al llarg de les seqüències a partir dels histogrames.
Així, l’aparició de coincidències al llarg
d’aquestes seqüències seria deguda a l’atzar, que
permetria l’aparició dels motius en gran quantitat degut a la
curta longitud d’aquests. Els resultats serveixen de control negatiu
respecte els resultats anteriors, donat que permeten descartar una
distribució funcional sobre seqüències no
promotores (fitxers de sortida de
C i
D).
Tornar a Taula de continguts
Discussió
Els estudis realitzats fins al moment sobre la proteïna CREB i la
seva localització nuclear han permès afirmar que es tracta
d’un factor de trancripció activat per múltiples vies
i que s’uneix amb alta afinitat a determinades regions promotores
del genoma. És de vital importància reconèixer els
patrons d’activació i de cooperació que segueix aquest
factor per tal d’esbrinar detalladament quin ampli ventall de gens
és capaç d’activar i en quines circumstàncies. Un
primer pas en aquesta labor és determinar quan CREB interacciona
amb factors d’unió a TATA i en quins promotors ho fa. Una
aproximació a aquesta cooperació és realitzar un
anàlisi computacional que permeti relacionar el patró de
distribució de CRE en determinades regions del genoma amb el de TATA.
Aquest estudi, realitzat sobre una mostra representativa de seqüències
promotores, és el que s’ha realitzat en aquest treball.
Els resultats obtinguts inviten a pensar que realment es produeix una bona
correlació entre la distribució d’ambdós elements.
Aquesta dependència de localització suggereix una funció
cooperativa entre els factors que s’uneixen a aquests elements per impulsar la
transcripció d’un subconjunt específic de gens activats per CREB.
Aquest fet permetria limitar o, si més no, encaminar la búsqueda de quins
gens són activats per CREB i per què.
Tornar a Taula de continguts















 Figura 1: vies d'activació de CREB
Figura 1: vies d'activació de CREB