Per tal de caracteritzar les selenoproteïnes exitents a Taenyopigia guttata hem fet un anàlisi d’homologia amb un organisme que té les selenoproteïnes ben caracteritzades (Gallus gallus). El resultat d’aquesta comparació el trobem a les següents línies.
Iodotironina deionidases (DIO)
La família iodotironina deionidases (DIO) està formada per tres proteïnes paràlogues en mamífers (DIO1, DIO2 i DIO3), que intervenen en la regulació de l'activitat de l'hormona tiroidea per la deionidació reductiva. Les deionidases són selenoproteïnes integrals de membrana caracteritzades per un plec de tiorredoxina. Els tres selenoenzims de deionidasa contenen un únic domini transmembrana i formen una estructura homodimèrica. Aquesta família té un paper important en el manteniment dels nivells d'hormona tiroïdal i la seva activitat tant per activar la seva prohormona com per degradar la T3 biològicament activa. [13][14]
DIO1
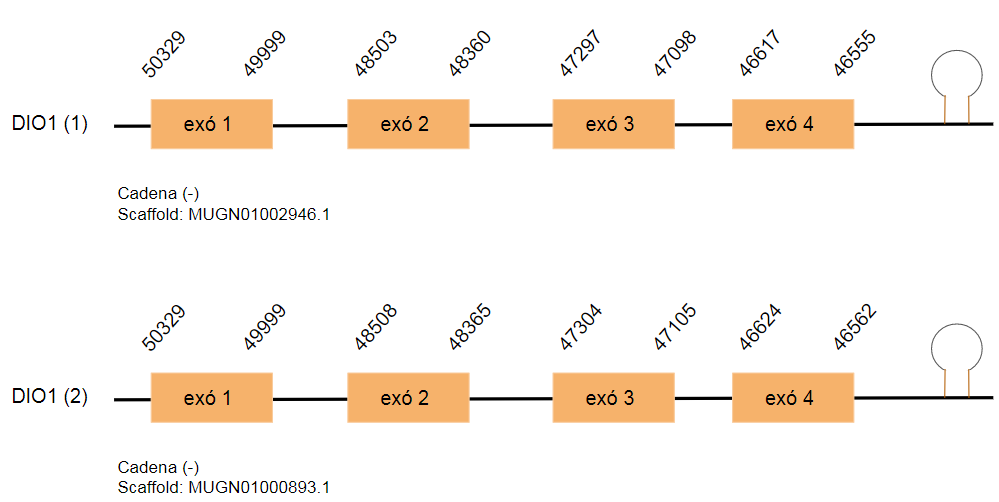
És responsable de l'eliminació del iode de l'anell exterior de T4 per convertir-la en T3 plasmàtica i, a més a més, catalitza la inactivació de T3. Per tant, juga un paper important com a proveïdor de T3 al plasma en els teixits perifèrics com fetge, ronyó i a la glàndula tiroides.
Aquesta proteïna al genoma de Gallus gallus conté 246 aminoàcids. S’han predit dos hits al genoma de Taenyopigia guttata a que es troben a dos scaffolds diferents. El primer d’ell es troba a l’scaffold MUGN01002946.1 i el segon es troba a l’scaffold MUGN01000893.1. i cada un està format per quatre exons. La proteïna en els dos casos es localitza a la cadena reversa.
Per altra banda, s’ha predit un SECIS per a cada scaffold però es troben en una posició molt allunyada del 3’UTR, en concret, es troba a més de 1000 bases de l’UGA. Per aquest motiu, no es pot dir que aquest SECIS formi part de la DIO1.
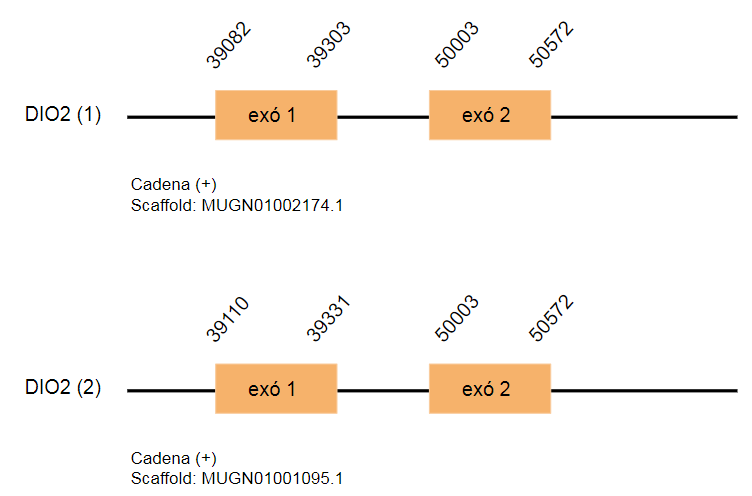
DIO2
S'encarrega exclusivament de l'activació de l'hormona tiroidea mitjançant la desiodació de l'anell tirosil exterior, és essencial per aportar al cervell els nivells suficients de T3 durant el desenvolupament.
La DIO2 està formada per 264 aminoàcids al genoma de Gallus gallus. S’han predit dos hits al genoma estudiat que es troben a dos scaffolds diferents: MUGN01002174.1 i MUGN01001095.1. Els dos hits s’han trobat a la cadena positiva i cada un està format per dos exons. En aquest cas, es creu que hi ha una duplicació ja que el resultat obtingut després de realitzar el t-coffee és idèntic en els dos scaffolds.
Pel que fa a la predicció de SECIS, s’ha predit un SECIS però com que la seva posició està molt allunyada de la seqüència codificant, no es considera com una selenoproteïna.
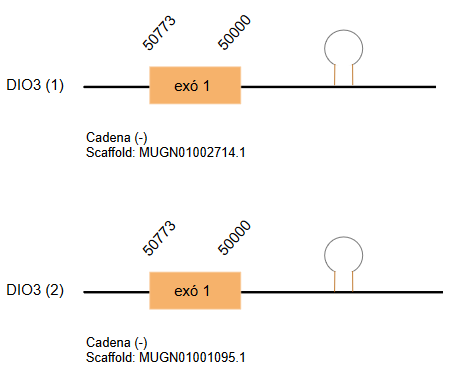
DIO3
Catalitza l’eliminació de l’anell intern de iode, portant a la formació d’una T2 inactiva i una T3 reversa (rT3). És localitza al còrtex cerebral i a la pell, i està expressada en nivells alts a la placenta i al úter durant l’embaràs.
Aquesta proteïna està formada per 258 aminoàcids. S’ha predit un hit a dos scaffolds diferents: MUGN01002714.1 i MUGN01001095.1. Es troben la cadena reversa i estan formats per un exó. Aquest cas també es creu que es tracta d’una duplicació ja que els resultats obtinguts del t-coffee son idèntics.
S’han predit elements SECIS però no es troben a una distància raonable del gen, per tant, no es pot concloure que aquest element formi part de la proteïna DIO3.
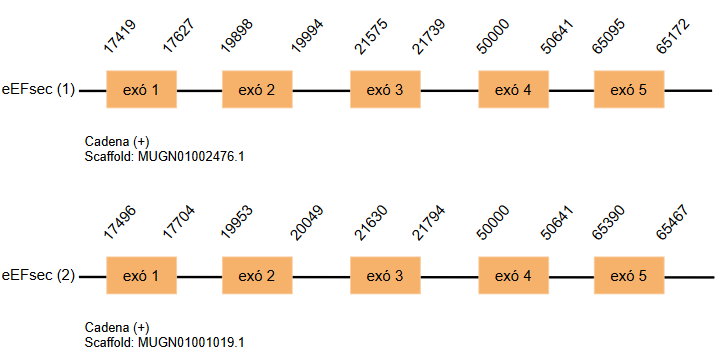
Eukaryotic elongation factor (eEFsec)
És una proteïna de la maquinària relacionada amb la síntesis de selenoproteïnes. Es tracta d’un factor d’elongació eucariota, que té funcions en la detoxificació del peròxid d’hidrogen i és un dels enzims antioxidants més importants dels humans. eEFsec interacciona amb el complex format per l’element SECIS i la SBP2 i fa que es promogui la unió del tRNA-Sec a l’espai A del ribosoma. [15]
En el genoma de Gallus gallus està codificada per 531 aminoàcids. S’han trobat dues còpies d’aquest gen en el genoma de Taenyopigia guttata en dos scaffolds: MUGN01002476.1 i MUGN01001019.1. La proteïna està formada per cinc exons i es troba a la cadena positiva.
No s’han predit cap element SECIS.
Glutatió peroxidases (GPx)
És la família de selenoproteïnes més gran en vertebrats i la més ben estudiada. El rol més important d’aquests enzims és la participació en les vies de protecció de l'organisme enfront l’estrès oxidatiu. La funció bioquímica del glutatió peroxidasa és la de catalitzar la reducció del peròxid o lipoperòxid utilitzat com a reduït el glutatió.
Aquesta família està integrada per 8 proteïnes homòlogues en mamífers (GPx1-8) de les quals GPx1-4 i GPx6 són selenoproteïnes en humans i, en canvi, GPx5, GPx7 i GPx8 contenen homòlegs de cisteïna. En aus, concretament al genoma de Gallus gallus, trobem tres isoformes de l’enzim glutatió peroxidasa: GPx3, GPx7 i GPx8. [16]
GPx3
GPx3 es secreta principalment al ronyó i és la isoforma de GPx més abundant en plasma. S’utilitza com a marcador dels nivells de seleni.
En el genoma de Gallus gallus, aquesta proteïna està codificada per 207 aminoàcids. S’han predit quatre hits, un a cada scaffold diferent: MUGN01001966.1, MUGN01001067.1, MUGN01002945.1 i MUGN01001019.1. Els dos primers hits són una duplicació, es troben a la cadena positiva i estan formats per quatre exons. Els altres dos hits es troben a la cadena reversa i estan formats per dos i un exó respectivament.
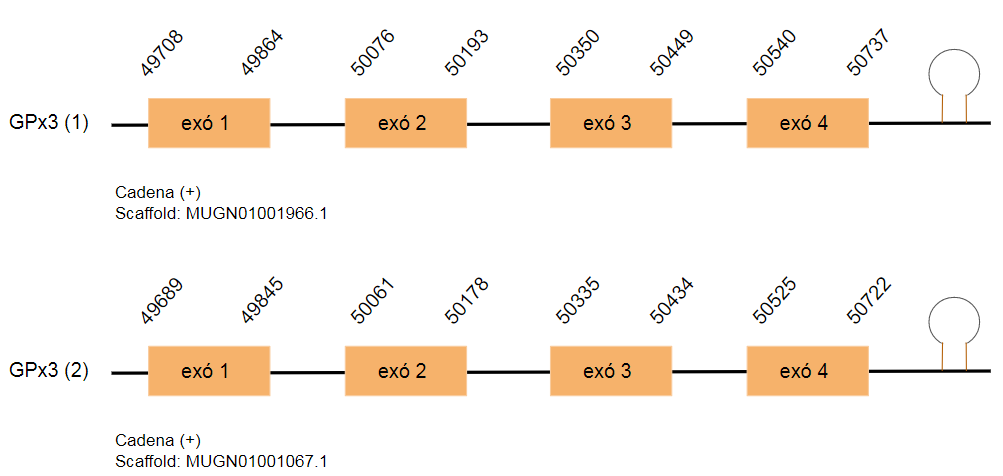
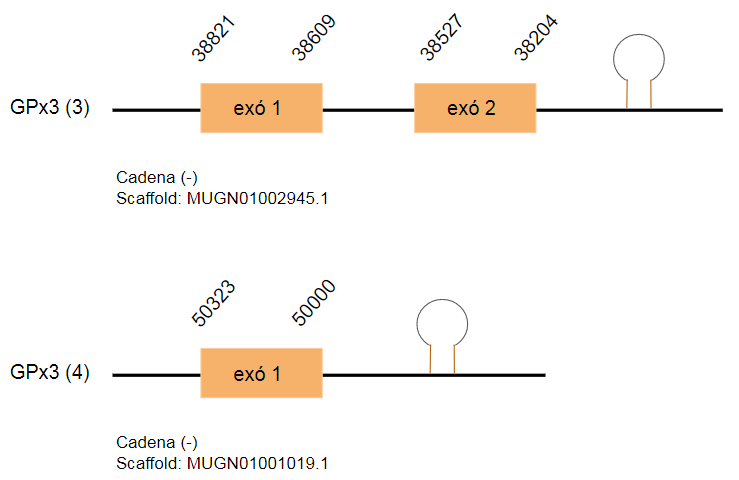
Pel que fa a la predicció de SECIS, s’ha predit un SECIS a la cadena corresponent i que es troba a una distància considerable del gen.
GPx7
Aquesta proteïna redueix l’estrès cel·lular oxidatiu induït per peròxid i àcids grassos poliinsaturats. [17]
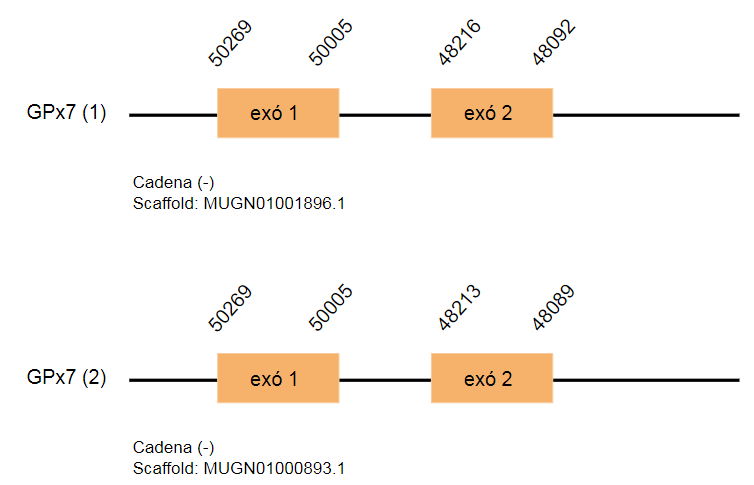
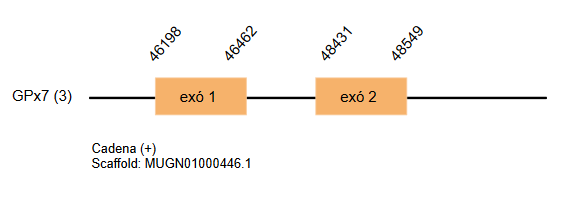
La GPx7 està codificada per 128 aminoàcids en el genoma de Gallus gallus. S’han trobat tres hits que es troben en tres scaffolds diferents: MUGN01001896.1, MUGN01000893.1 i MUGN01000446.1. Els dos primers hits es tracten d’una duplicació, es troben a la cadena reversa i contenen dos exons. L’últim hit anomenat es troba a la cadena positiva i conté dos exons. Però aquest hit, també codifica per la següent proteïna, la GPx8, i en aquest cas ho fa amb més significança. Per tant, no considerarem que el hit MUGN01000446.1 codifiqui per la GPx7.
No s’ha pogut predir cap element SECIS.
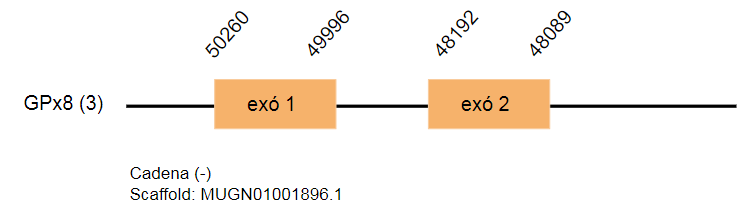
GPx8
Aquesta isoforma també té la funció de reduir l’estrès cel·lular oxidatiu.
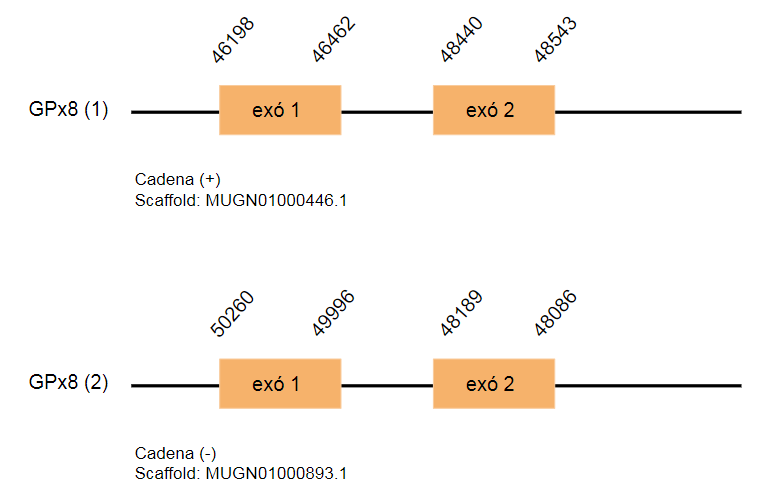
En el genoma de Gallus gallus està codificada per 123 aminoàcids. S’han localitzat tres hits localitzats en tres scaffolds: MUGN01000446.1, MUGN01000893.1 i MUGN01001896.1. El primer hit es troba a la cadena positiva i està format per dos exons. Els dos últims son una duplicació i es troben a la cadena reversa i també estan formats per dos exons.
Pel que fa a la predicció de SECIS, no s’han obtingut resultats.
Com es pot observar, els tres scaffolds identificats codifiquen tant per la GPx7 com per la GPx8 i no es pot saber amb seguretat quin scaffold correspon a cada isoforma. Aquest fet pot ser degut a la divergència evolutiva d’aquesta família de proteïnes.
Metionina sulfoxi reductasa A (MsrA)
MsrA és un enzim que catalitza la reducció enzimàtica de sulfoxi-metionina a metionina. En alguns organismes com els eucariotes unicel·lulars i els bacteris anaeròbics, MsrA té un residu Sec situat al lloc actiu mentre que en altres organismes, com els vertebrats, aquest residu és una Cys. S'ha demostrat que el residu Sec proporciona avantatges catalítics en els enzims redox-actius. [13]
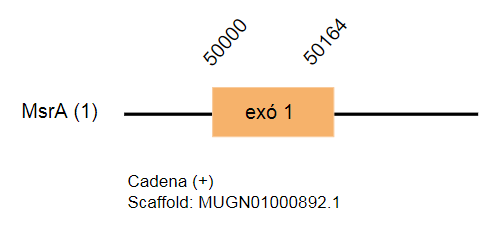
La proteïna MsrA està codificada per 125 aminoàcids en el genoma de Gallus gallus. En l’espècie estudiada, la MsrA s’ha predit com a homòloga a cisteïna. S’ha trobat un hit (MUGN01000892.1) que es troba a la cadena positiva i està format per un exó.
S’ha predit un SECIS però es troba molt allunyat de la regió 3’UTR.
Selenoproteïna R (MSRB)
Aquesta proteïna pertany a la família de les sulfoxi-metionina reductases (Msr) que tenen la funció de reduir els residus de metionina oxidats en les proteïnes, protegint la cèl·lula de l’estrès oxidatiu. [13]
MSRB1
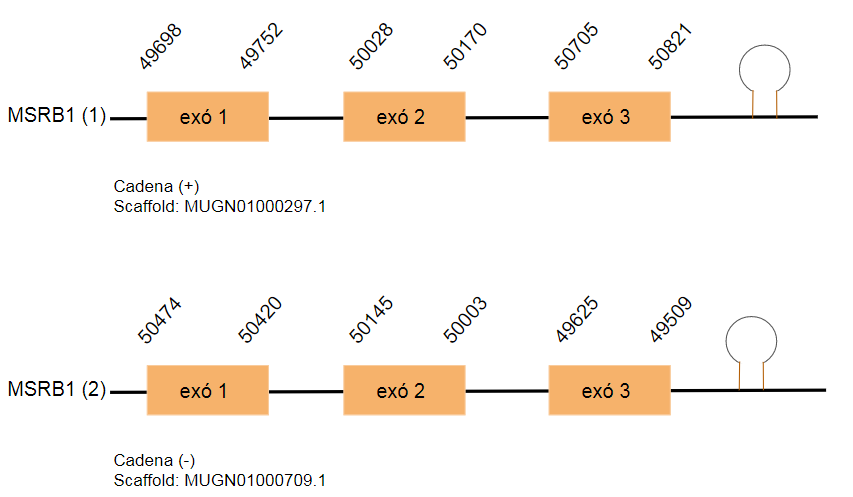
En el genoma de Gallus gallus aquesta proteïna està formada per 107 aminoàcids. S’han predit dos hits situats en els següents scaffolds: MUGN01000297.1 i MUGN01000709.1 que contenen un residu de selenocisteïna. El primer hit s’ha trobat a la cadena positiva i té 3 exons. El segon, es troba a la cadena la reversa i també consta de tres exons.
S’ha predit un SECIS per a cada hit, però en ambdós casos està molt lluny de l’extrem 3’UTR
Els dos gens predits provenen de una duplicació en el genoma, ja que el resultat obtingut després de realitzar el t-coffee és igual en els dos casos.
MSRB3
Aquesta proteïna actua com a un enzim de funció equivalent a MSRB1. Necessita zinc que actuï com a cofactor. S’han trobat dues isoformes diferents en aquest gen, una d’elles es localitza al mitocondri i l’altra al reticle endoplasmàtic.
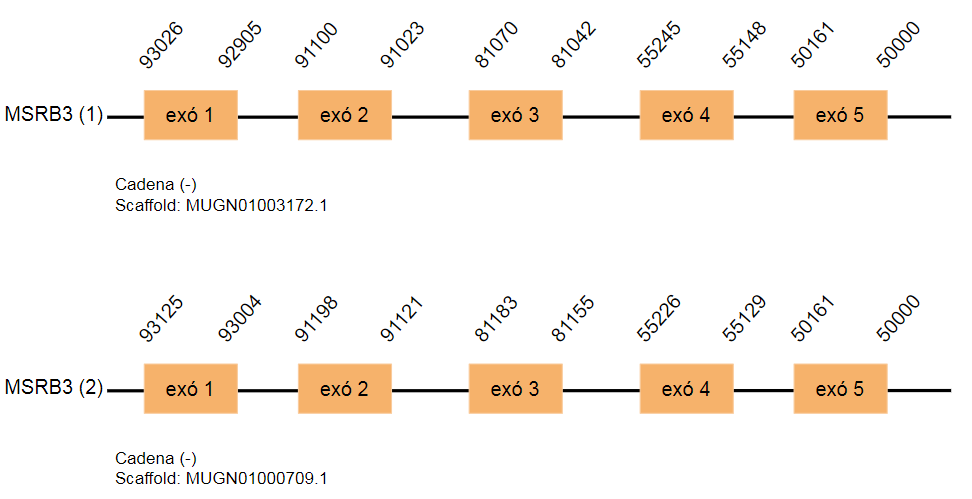
En aquest cas, s’han trobat dos hits, que contenen homòlegs a la selenocisteïna, situats en els següents scaffolds respectivament: MUGN01003172.1 i MUGN01000619.1. Els dos hits s’han trobat a la cadena reversa i tenen cinc exons cadascun.
No s’han trobat SECIS en cap dels dos hits. El resultat dels dos scaffolds és idèntic, fet que porta a determinar que s’han format a partir de una duplicació.
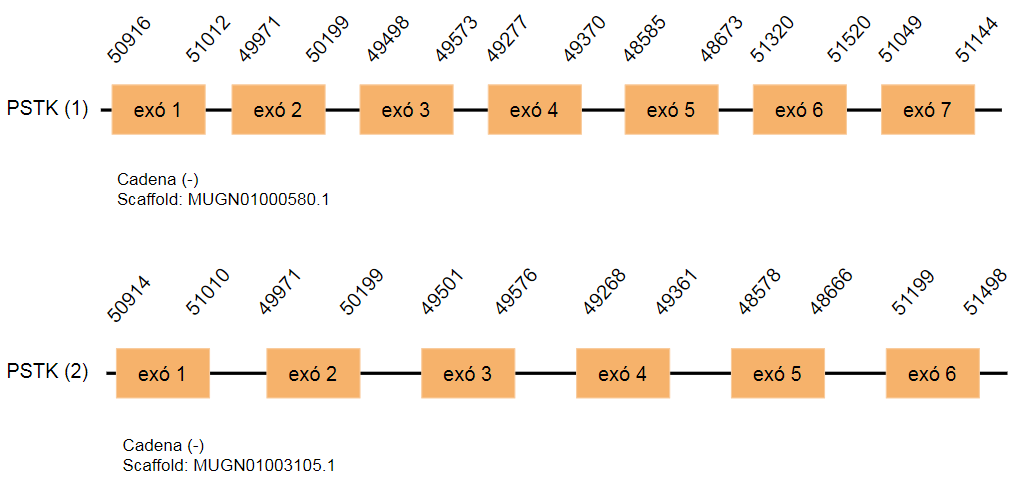
Phosphoseryl-tRNA kinase (PSTK)
La phosphoseryl-tRNA kinase (PSTK) és una proteïna quinasa implicada en catalitzar la reacció de seril-tRNA[Ser]Sec a la forma fosforilada fent servir una molècula d’ATP. Està formada per dos dominis independents connectats, el domini catalític N-terminal (NTD) i el domini C-terminal (CTD). La seva acció és essencial per la codificació específica UGA de les selenocisteïnes. [18]
No s’ha trobat cap SECIS en cap dels dos hits, tot i que, en el primer s’han trobat dos SECIS, un dels quals s’ha rebutjat directament ja que es troba en la cadena positiva i l’altre no forma part de la proteïna estudiada. En el segon hit s’ha trobat un SECIS però tampoc forma part de la proteïna.
SECIS binding protein 2 (SBP2)
SBP2 està associada de manera estable als ribosomes i conté un domini d'unió de RNA L7Ae diferent que se sap que uneix els elements SECIS amb una gran afinitat i especificitat. De manera que permet incorporar l’aminoàcid selenocisteïna durant el procés de traducció. [13]
Totes les isoformes estudiades contenen un residu de selenocisteïna.
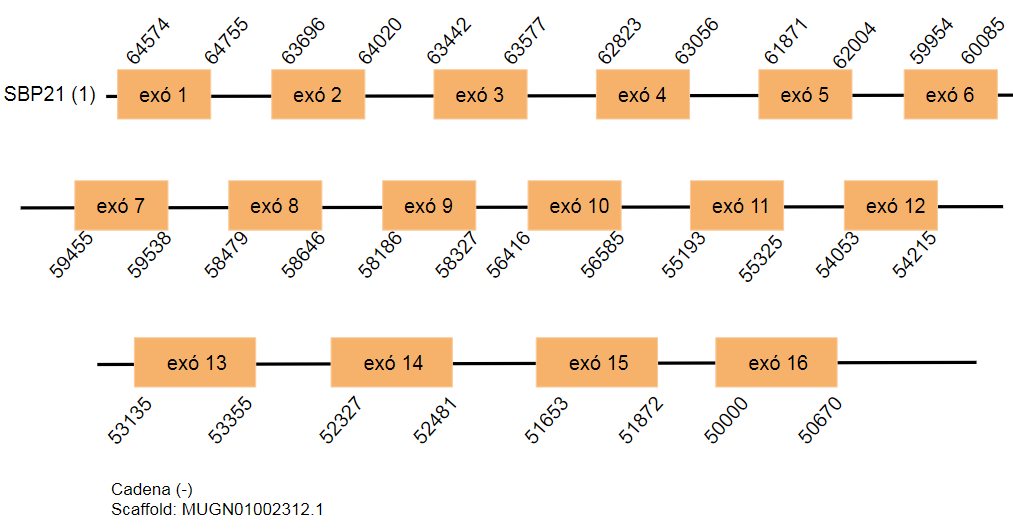
SBP2(1)
Aquesta proteïna es troba en el genoma Gallus gallus codificada per 1090 aminoàcids. S’han determinat dos hits al genoma de Taenyopigia guttata que es troben a diferents scaffolds. El primer hit es troba a l’scaffold MUGN01002312.1 i el segon es troba a l’scaffold MUGN01000131.1. En ambdós casos la proteïna es localitza en la cadena reversa i està formada per setze exons.
En aquest cas s’han trobat dos SECIS a la cadena no reversa i per aquest motiu s’han rebutjat.
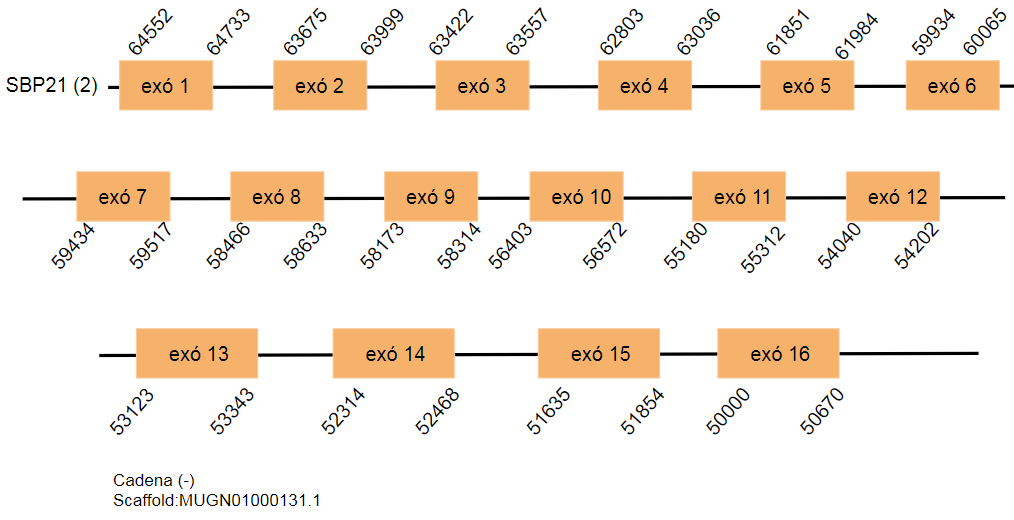
SBP2(2)
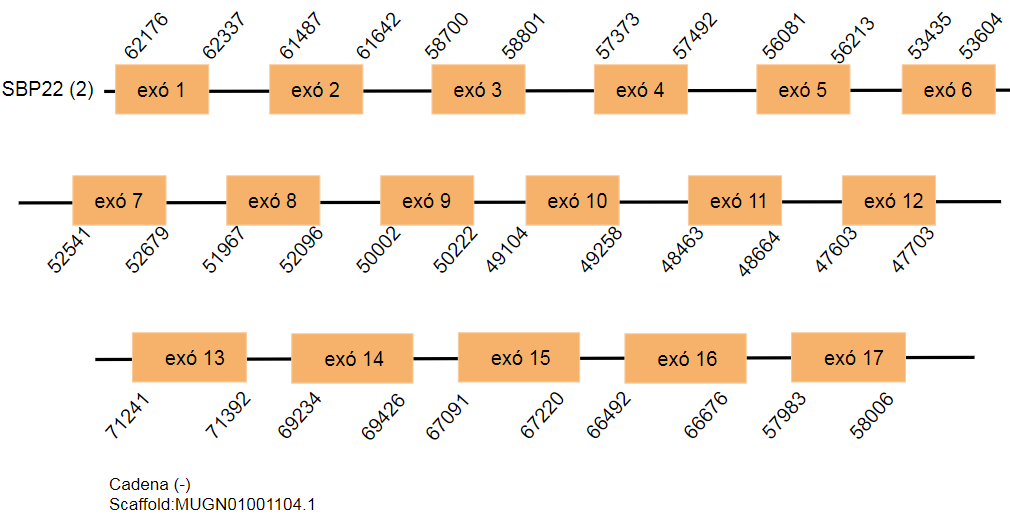
Aquesta isoforma de la SBP2 que es troba al genoma Gallus gallus codifica per 906 aminoàcids. S’han predit dos hits al genoma Taenyopigia guttata que es troben el primer hit a l’scaffold MUGN01002073.1 i el segon hit a l’scaffold MUGN01001104.1. La proteïna es troba en la cadena reversa i formada per disset exons en els dos hits.
En el primer hit s’ha determinat un element SECIS però s’ha rebutjat perquè no forma part de la proteïna estudiada perquè es troba a una distància massa allunyada del gen. En el segon hit s’han trobat dos SECIS, un dels quals s’ha rebutjat directament ja que es troba en la cadena positiva i l’altre no forma part de la proteïna.
Es creu que els hits estudiats són una duplicació, ja que el resultat del t-coffee és idèntic.
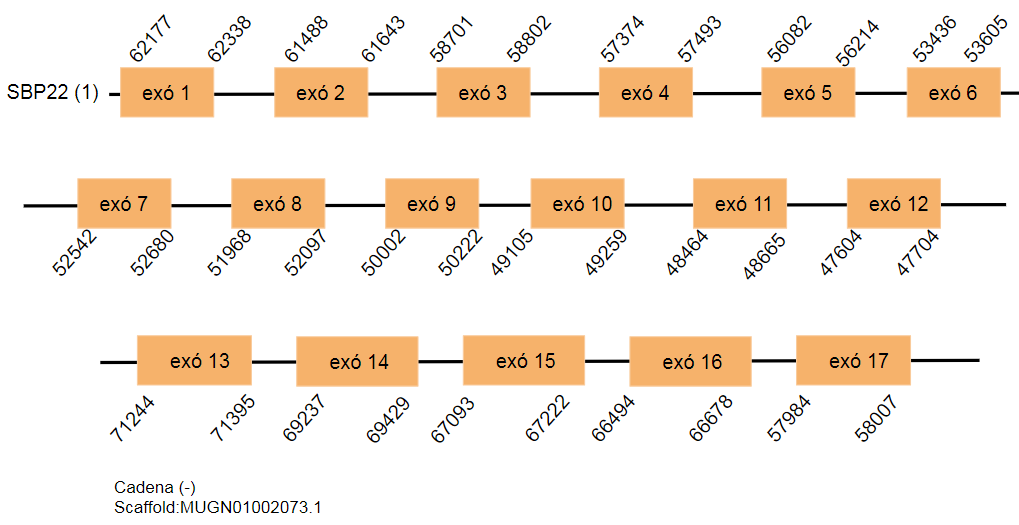
SBP2(3)
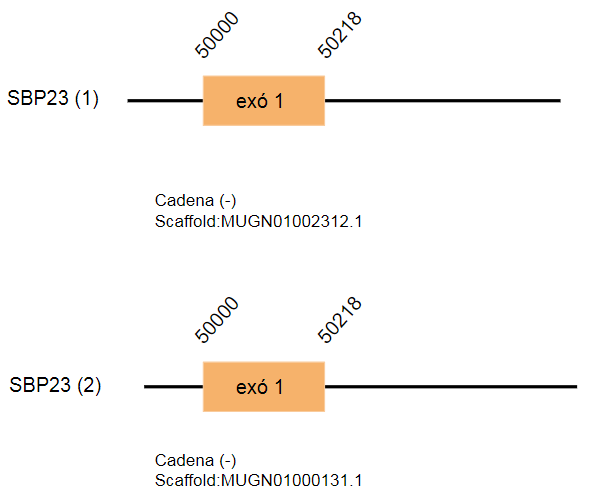
En el genoma del Gallus gallus aquesta la isoforma està codificada per 73 aminoàcids. S’han localitzat dos hits que es troben a diferents scaffolds. El primer hit es localitza a l’scaffold MUGN01002312.1 i el segon hit a l’scaffold MUGN01000131.1. Es localitzen a la cadena reversa i tant sols contenen un exó.
S’han determinat dos elements SECIS a la cadena positiva i per aquest motiu son rebutjats.
Els dos hits predits són una duplicació del gen que codifica per la isoforma 1, ja que es localitzen als mateixos scaffolds i a una localització similar.
tRNA Sec 1 associada a proteïna 1(SECp43)
La funció d’aquesta proteïna és regular l’expressió de les selenoproteïnes. Juntament amb altres molècules, forma part del complex proteic selenocisteïna sintasa que incorpora selenofosfat a l’aminoàcid. [19]
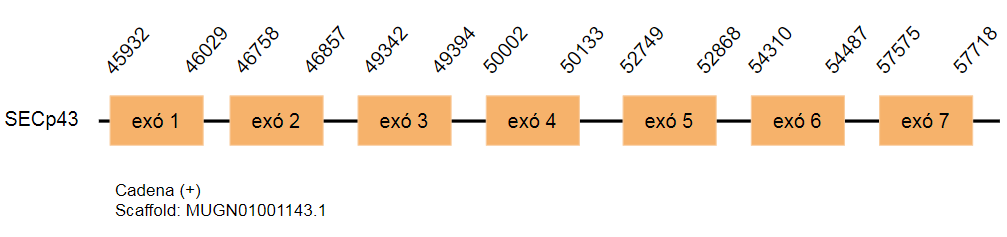
La seqüència de proteïna codificada per SECp43 al genoma de Gallus gallus conté 310 aa. Al genoma deTaenyopigia guttata s’ha trobat un hit a l’scaffold MUGN01001143.1 que es localitza a la cadena positiva i està format per set exons.
Pel que fa a la predicció de SECIS no se n’ha trobat cap.
Selenocisteïna sintetasa (SecS)
L'aminoàcid selenocisteïna és l'únic aminoàcid que no té la seva pròpia tRNA sintetasa. En lloc d'això, aquest aminoàcid es sintetitza en el seu propi tRNA en un procés de tres passos. La proteïna codificada per aquest gen catalitza el tercer pas del procés, la conversió de O-fosfoseril-tRNA (Sec) al selenocisteinil-ARNt (Sec). [20]
SecS1
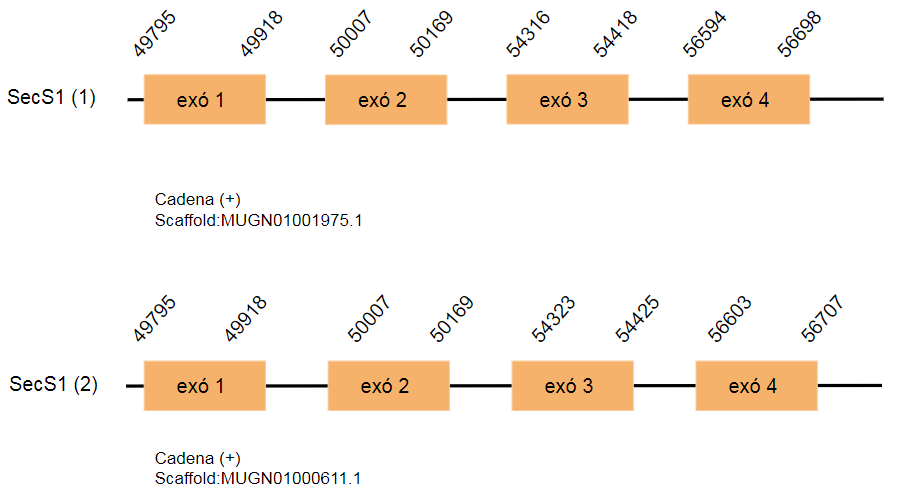
Al genoma del Gallus gallus aquesta proteïna codifica per 467 aminoàcids. S’han predit dos hits al genoma Taenyopigia guttata que es troben a diferents scaffolds, al MUGN01001975.1 i al MUGN01000611.1., respectivament. SecS1 tenen quatre exons que es localitzen a la cadena positiva. Els dos hits trobats es tracten d’una duplicació del gen.
A l’hora de predir els SECIS s’ha trobat un SECIS al primer hit però s’ha rebutjat perquè es troba a la cadena reversa. En el segon hit no s’ha trobat cap element SECIS.
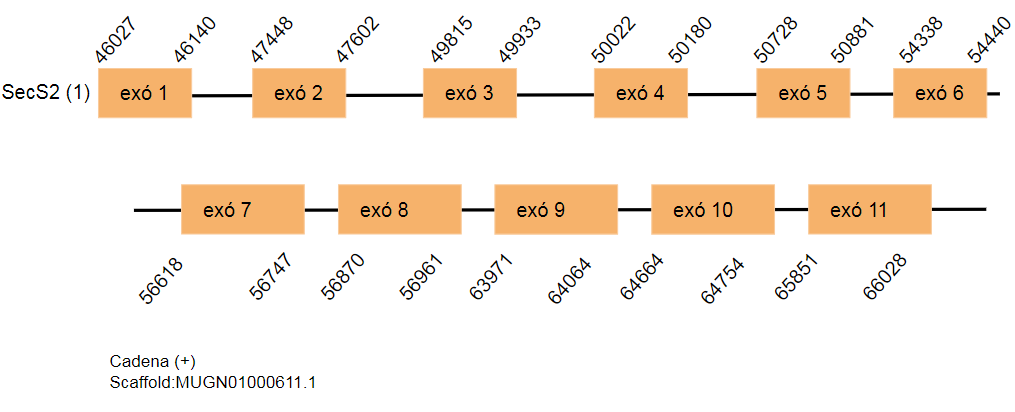
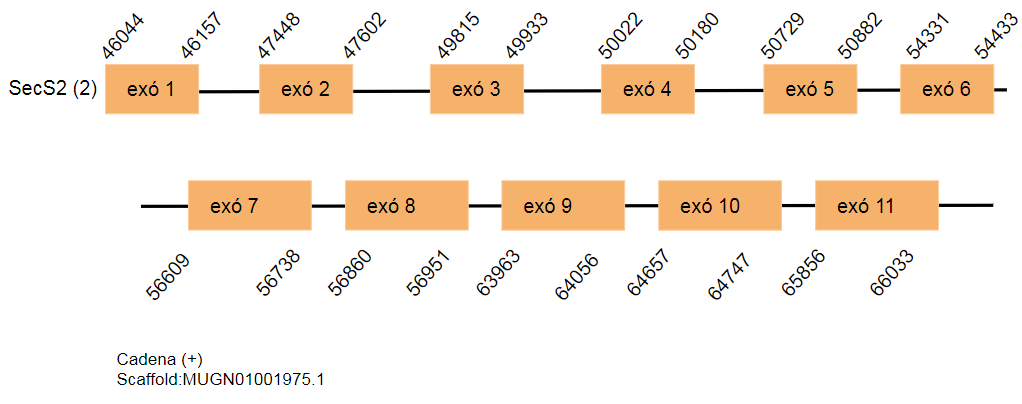
SecS2
En aquest cas 165 aminoàcids són els que codifiquen per aquesta proteïna en el genoma Gallus gallus. S’han trobat dos hits al genoma Taenyopigia guttata, els hits es localitzen a l’scaffold MUGN01000611.1 i a l’scaffold MUGN01001975.1. Els dos scaffolds es troben a la cadena positiva i consten d’onze exons.
En el primer hit no s’ha predit cap element SECIS i en el segon hit se n’ha trobat un però s’ha rebutjat perquè es troba a la cadena reversa.
Els dos hits predits són una duplicació del gen que codifica per la proteïna estudiada.
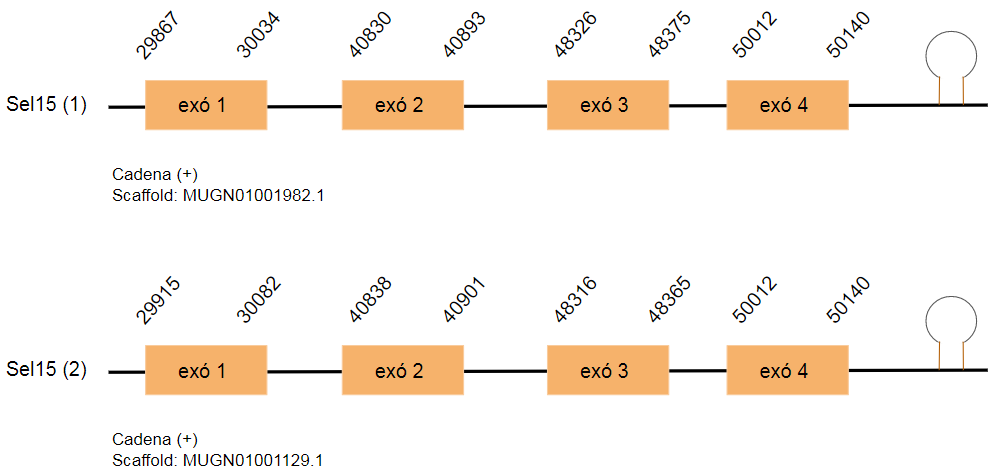
Selenoproteïna 15-kDa (Sel15)
La selenoproteïna de 15-kDA (Sel15) és una proteïna residual del reticle endoplasmàtic, la seva funció específica d’aquest a proteïna encara no es coneix, tot i que participa en el control de la qualitat del plegament de la glicoproteïna. [21]
Aquesta proteïna està formada per 137 aminoàcids en el genoma de Gallus gallus. S’han predit dos hits al genoma de Taenyopigia guttata que contenen un residu de selenocisteïna. Cada un està format per quatre exons i es troben a la cadena positiva. Els scaffolds son: MUGN01001982.1 i MUGN01001129.1.
S’ha predit un element SECIS que es troba a l’extrem 3’UTR de la cadena corresponent.
Selenoproteïna H (SelH)
La SelH és essencial per a la viabilitat i la defensa antioxidant de les cèl·lules. És una proteïna receptora de DNA que respon a la redox de la família AT-hook i funciona en la regulació dels nivells d'expressió de gens implicats en la síntesi de novo de glutatió i la desintoxicació de la fase II en resposta a l'estat de redox. [22][23]
Aquesta proteïna al genoma de Gallus gallus codifica per 96 aa. Al comparar amb el genoma de l’espècie problema no s’ha predit cap hit, per la qual cosa es creu que aquesta no és present en el genoma de Taenyopigia guttata.
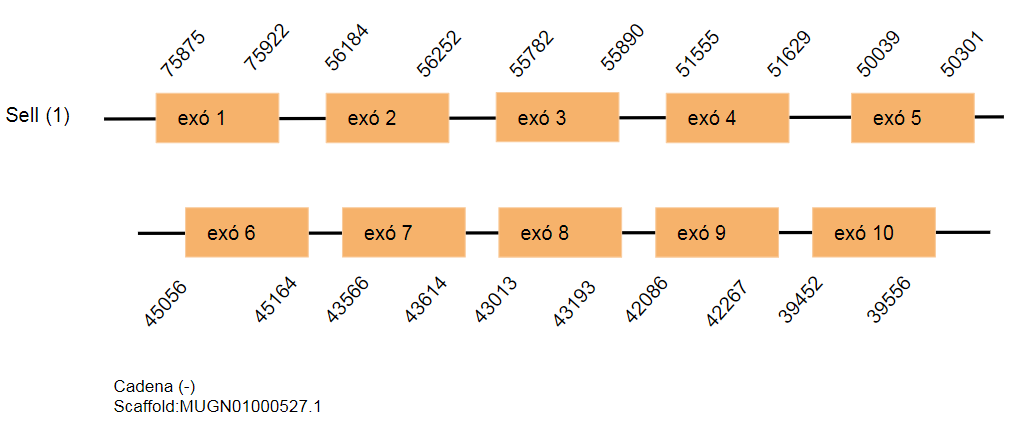
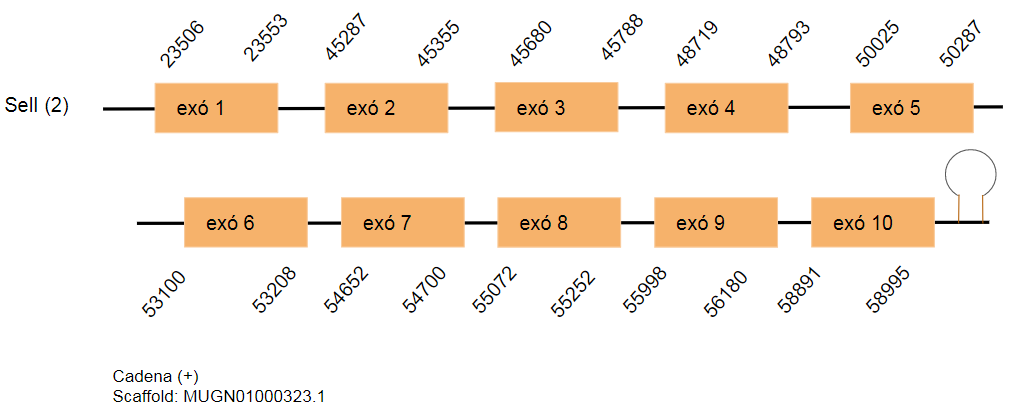
Selenoproteïna I (SelI)
És una selenoproteïna transmembrana que pertany a la família de les CDP-alcohol fosfatidiltransferases de classe I. Es catalitza la transferència de fosfoetanolamina de CDP-etanolamina a diacilglicerol per produir fosfatidiletanolamina, que participa en la formació i manteniment de membranes vesiculars, la regulació del metabolisme dels lípids i el plegament de proteïnes. [24]
Aquesta proteïna en el genoma de Gallus gallus està codificada per 400 aminoàcids. S’han determinat dos hits al genoma de Taenyopigia guttata que es troben en els scaffolds: MUGN01000527.1 i MUGN01000323.1.. Tots dos contenen deu exons i el primer scaffold es troba a la cadena reversa i el segon a la cadena positiva.
En el primer hit no s’ha predit cap SECIS i en el segon hit s’ha predit un SECIS que compleix les condicions esmentades a la metodologia.
Selenoproteïna K (SelK) i Selenoproteïna S (SelS)
SelK i SelS són membres del complex de degradació associada al reticle endoplasmàtic (ERAD), que s'encarrega de translocar proteïnes del reticle endoplasmàtic (ER) al citosol per degradació. A més a més s’ha determinat que SelK s'uneix i estabilitza la palmitoil transferasa DHHC6 i, per tant, contribueix a la palmitoilació. SelK i SelS resideixen a la membrana ER per mitjà d'una sola hèlix transmembrana. [25]
SelK1
Conté 95 aminoàcids en el genoma Gallus gallus. Després de córrer el programa no s’ha obtingut cap resultat. Això indica que aquesta proteïna no es troba al genoma de Taenyopigia guttata
SelK2 i selK3
Són dues isoformes de la SelK1 que tampoc s’han pogut trobar al genoma de l’espècie estudiada, ja que el programa no donava resultats per aquestes. De tal manera que es creu que aquestes isoformes no formen part de Taenyopigia guttata.
SelS
SelS, igual que amb les proteïnes anteriors, tampoc s’ha trobat al genoma de l’espècie estudiada.
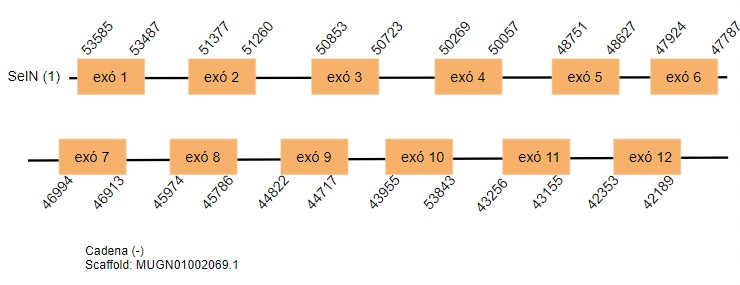
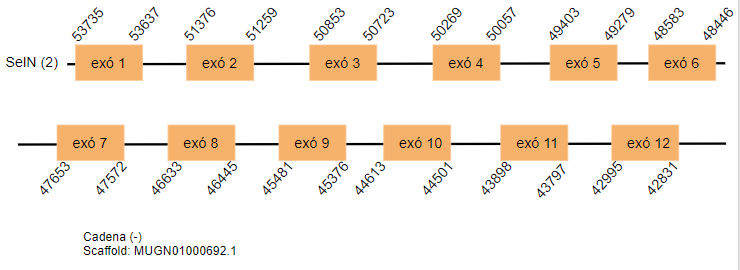
Selenoproteïna N (SelN)
SelN és una glicoproteïna de membrana relacionada amb el desenvolupament embrionari i sobretot del múscul esquelètic, s’expressa en altes concentracions durant l’embriogènesi i la seva expressió baixa en l’adult. [26][27]
Aquesta proteïna en el genoma de Gallus gallus conté 530 aminoàcids. S’han predit dos hits al genoma de Taenyopigia guttata que contenen dotze exons cada un, els hits es localitzen a l’scaffold MUGN01002069.1 i a l’scaffold MUGN01000692.1. Està localitzada en tots dos casos a la cadena reversa.
S’ha pogut predir un SECIS a cada scaffold però es troben a una distància massa allunyada del 3’UTR, per tant, no poden ser de la proteïna estudiada.
Els dos scaffolds trobats son molt similars però no son una duplicació. No es pot afirmar que siguin pseudogens ja que no s’ha fet un estudi transcripcional.
Selenoproteïna O (SelO)
SelO és una selenoproteïna que es troba en mamífers amb ortòlegs que es troben en una gran varietat d'organismes, incloent bacteris i llevats. És una selenoproteïna mitocondrial activa en diverses funcions redox de sistemes biològics. [28]
En totes les isoformes estudiades, s’han trobat scaffolds molt similars entre ells. Tot i així, no es poden considerar pseudogens perquè no s’ha realitzat un estudi més profund de la seva transcripció.
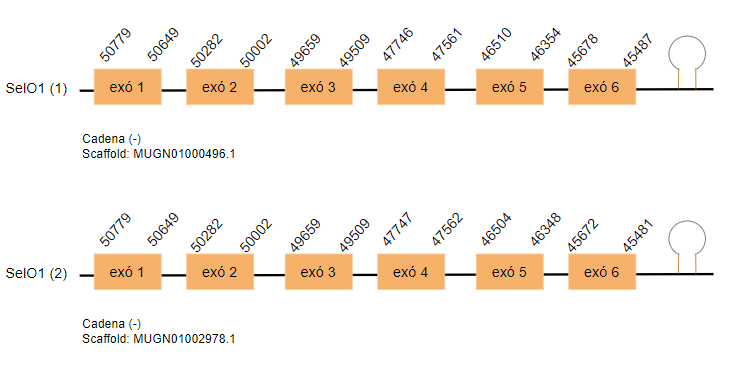
SelO1
Aquesta isoforma es troba codificada per 364 aminoàcids al genoma de Gallus gallus. En el cas de l’espècie problema s’han predit dos hits que es troben en dos scaffolds diferents. Un hit es troba en l’sacffold MUGN01000496.1 i l’altre al MUGN01002978.1. Tots dos estan localitzats a la cadena reversa i estant formats per sis exons i contenen un residu Sec.
S’ha predit un element SECIS a cada hit a la cadena reversa a una distància considerable de l’extrem 3’UTR.
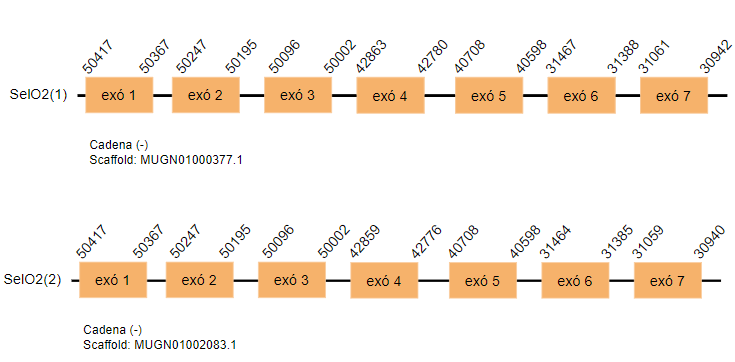
SelO2
La isoforma SelO2 que es troba al genoma de Gallus gallus codificada per 198 aminoàcids. S’han predit dos hits codificats per set exons a la cadena reversa localitzats als scaffolds MUGN01000377.1 i MUGN01002083.1. Aquesta, és una homòloga de cisteïna.
Els SECIS d’aquests scaffolds es troben molt allunyats de l’extrem 3’UTR.
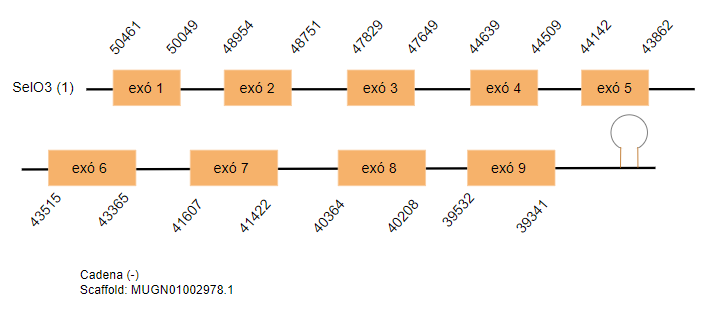
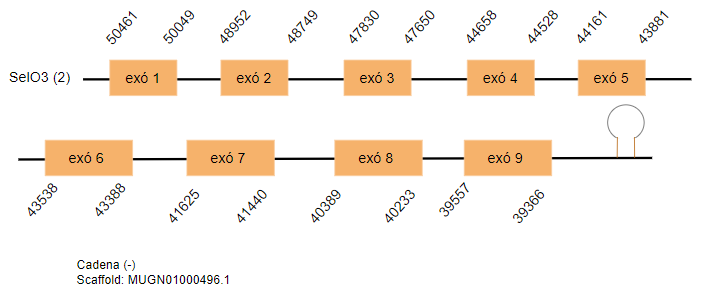
SelO3
En Gallus gallus la isoforma està codificada per 629 aminoàcids. En l’espècie estudiada s’han predit dos hits localitzats en dos scaffolds diferents MUGN01000496.1 i MUGN01002978.1, tots dos estan formats per nou exons i es troben a la cadena reversa. La isoforma conté un residu Sec en tots dos hits.
S’ha predit un SECIS de cada scaffold, una distància considerable de l’extrem 3’UTR.
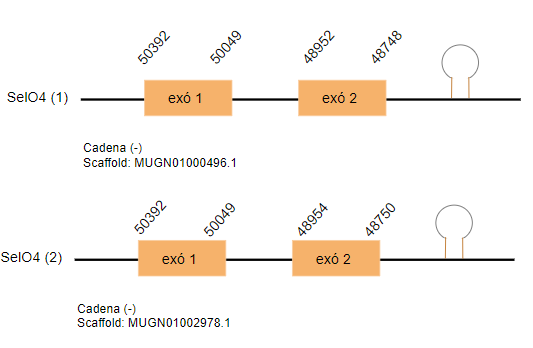
SelO4
182 aminoàcids són els que codifiquen per aquesta isoforma al genoma de Gallus gallus. Al genoma de Taenyopigia guttata s’han trobat dos hits localitzats als scaffolds MUGN01000496.1 i MUGN01002978.1 respectivament. Estan formats per dos exons i localitzats a la cadena reversa i són homòlegs de cisteïna.
Els SECIS han estat predits i en tots dos casos es localitzen a la mateixa distància de l’extrem 3’UTR.
Selenoproteïna P (SelP)
La SelP és la principal transportadora de seleni en el sèrum. A més, juga un paper com a proteïna local d'emmagatzematge i reciclatge al cervell, on manté la homeòstasi del seleni. [29]
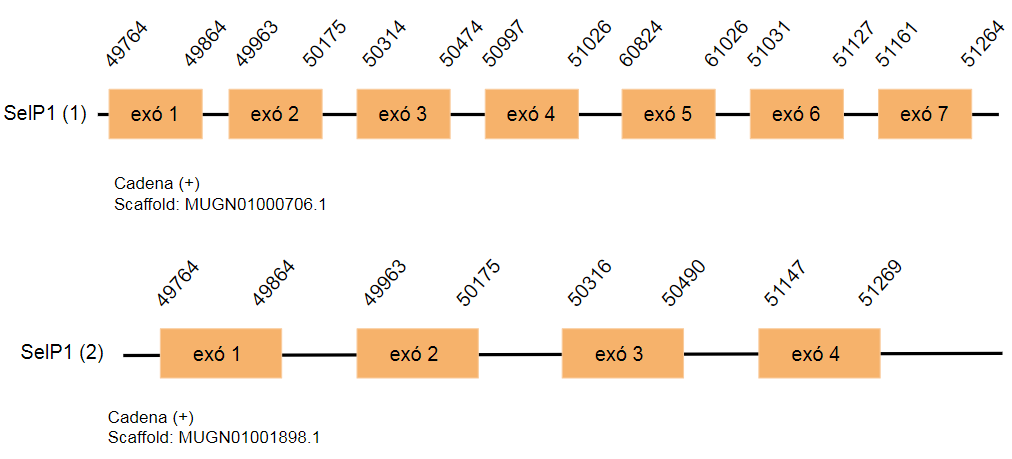
SelP1
Al genoma del Gallus gallus aquesta proteïna codifica per 267 aminoàcids. Al genoma estudiat s’han predit dos hits als scaffolds MUGN01001898.1 i MUGN01000706.1, el primer de set exons i el segon de quatre. Tots dos es troben a la cadena positiva i conté un residu homòleg a la cisteïna.
Els SECIS es troben a més de 800 posicions de l’extrem 3’UTR, per tant no pot pertànyer a la proteïna estudiada.
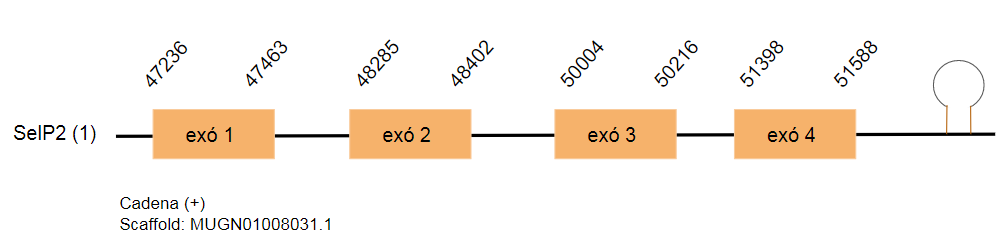
SelP2
Aquesta proteïna codifica per 251 aminoàcids al genoma de Gallus gallus. S’ha trobat un hit per aquesta proteïna en l’scaffold MUGN01000803.1 a la cadena reversa i format per quatre exons que conté un residu Sec.
El SECIS predit compleix els criteris esmentats en els mètodes.
Selenoproteïna T (SelT)
Es tracta d’una proteïna present a l'aparell de Golgi i al reticle endoplasmàtic (RE). La seva deficiència altera l'adhesió cel·lular i millora l’expressió de gens oxido-reductors per tant, està implicada en els processos d'organització i ancoratge cel·lular. [30]
En el genoma de Gallus gallus està codificada per 400 aminoàcids. En l’espècie estudiada no s’ha pogut predir cap hit ja que el programa després de realitzar el BLAST no realitza la resta de passos per analitzar la proteïna. Això vol dir, que no s’ha pogut estudiar
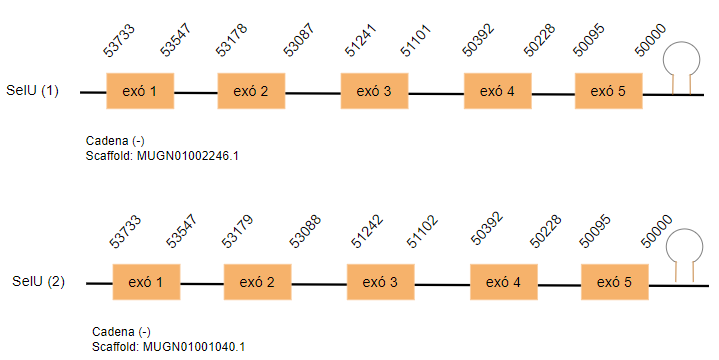
Selenoproteïna T (SelU)
Aquesta proteïna s’expressa a l’os, cervell, fetge i ronyó. La seva funció no està ben descrita, però podria estar relacionada amb la superfamília de les tioredoxines-like. [31]
En Gallus gallus està codificada per 227 aminoàcids. S’han trobat dos hits per SelU als scaffolds MUGN01002246.1 i MUGN01001040.1. Ambdós es troben a la cadena reversa i tenen cinc exons i un residu Sec.
S’ha predit un SECIS per cada un dels hits a poca distància del 3’UTR.
Els resultats idèntics d’aquests dos hits, permeten dir que són una duplicació.
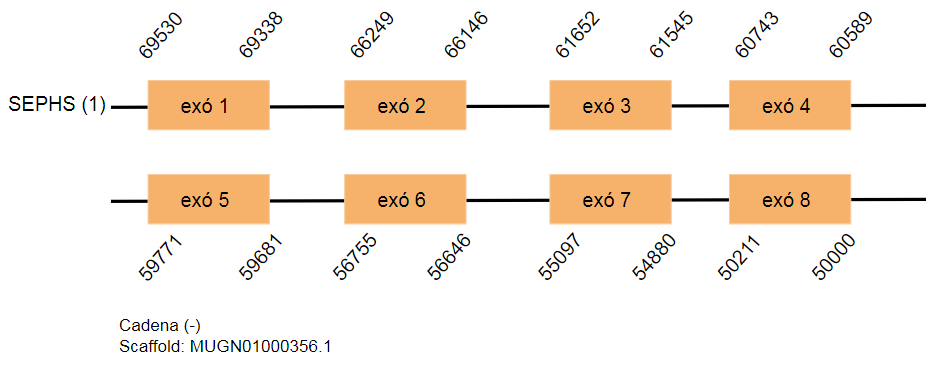
Selenofosfat sintetasa (SEPHS)
Aquest gen codifica per un enzim que sintetitza el selenofosfat de selenida i ATP. El selenofosfat és el donant de seleni utilitzat per sintetitzar selenocisteïna. [32]
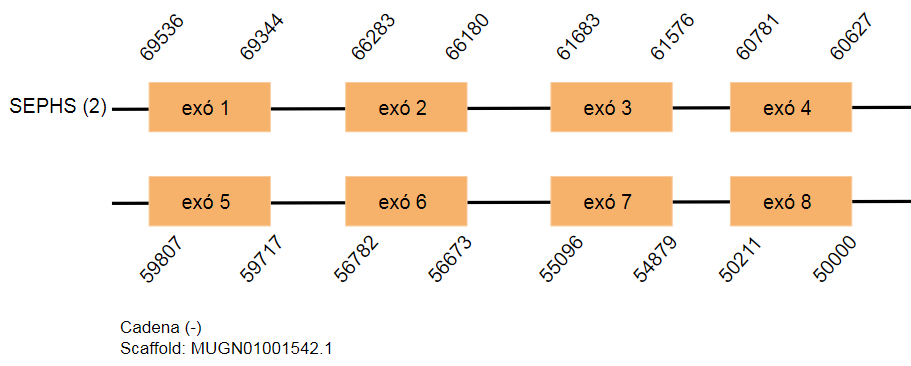
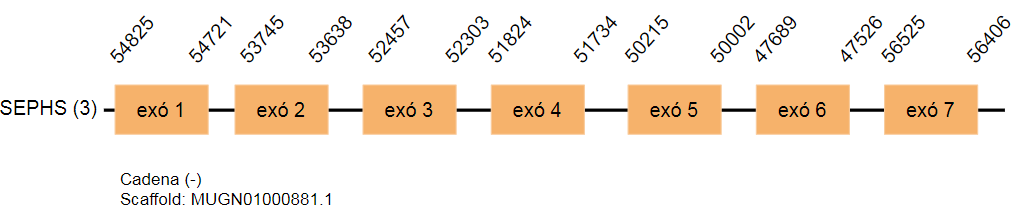
Aquesta proteïna es troba en el genoma Gallus gallus codificada per 397 aminoàcids. S’han determinat tres hits al genoma de Taenyopigia guttata que es troben a diferents scaffolds. El primer hit es troba a l’scaffold MUGN01000356.1 i conté vuit exons, el segon es troba a l’scaffold MUGN01001542.1 i conté vuit exons i el tercer hit a l’scaffold MUGN01000881.1 i conté set exons. SEPHS es localitza en la cadena reversa en els tres casos.
En els tres hits s’ha determinat un element SECIS però com que es troben a una posició allunyada del 3’UTR i per tant no poden ser de la proteïna estudiada.
Els dos primers hits són idèntics i això podria determinar que s’han format a partir d’una duplicació.
Thioredoxin reductasa (TXNRD)
Aquest gen codifica per un membre de la família de les oxidoreductases de piridina. Aquestes tenen la funció de reduir tioredoxines i altres substàncies. També té un paper important en el metabolisme del seleni i la protecció envers l’estrès oxidatiu. L’enzim actua en forma de homodímer utilitzant com a cofactor FAD (dinucleòtid de flavina i adenina). Cada subunitat conté selenocisteïna que li confereix activitat catalítica. [33]
Totes les isoformes estudiades contenen un residu de selenocisteïna.
TXNRD1
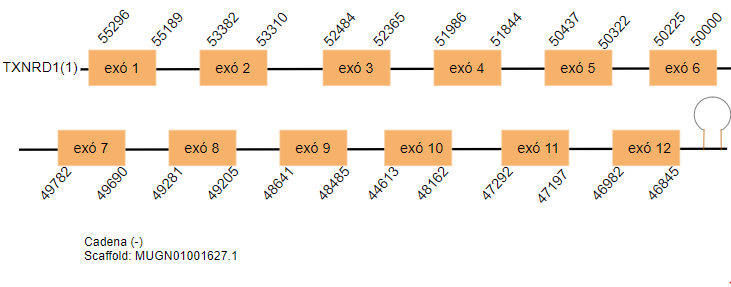
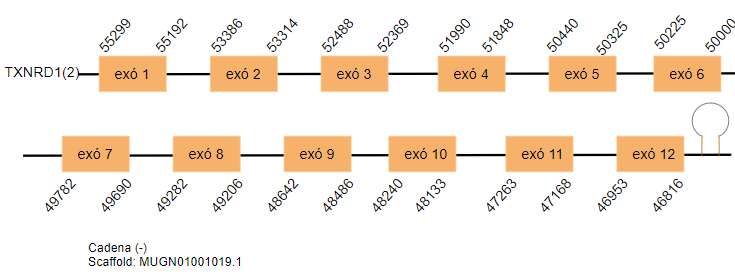
En el genoma de Gallus gallus aquesta proteïna està formada per 511 aminoàcids. En l’espècie estudiada s’han trobat quatre hits significatius situats als scaffolds següents: MUGN01001627.1, MUGN01001019.1, MUGN01002868.1 i MUGN01000779.1. Tots ells es situen a la cadena reversa. Els dos primers hits contenen dotze exons i els altres dos tretze exons.
S’ha predit un element SECIS en tots els hits obtinguts.
El primer i segon hit són una duplicació entre ells i el tercer i el quart també.
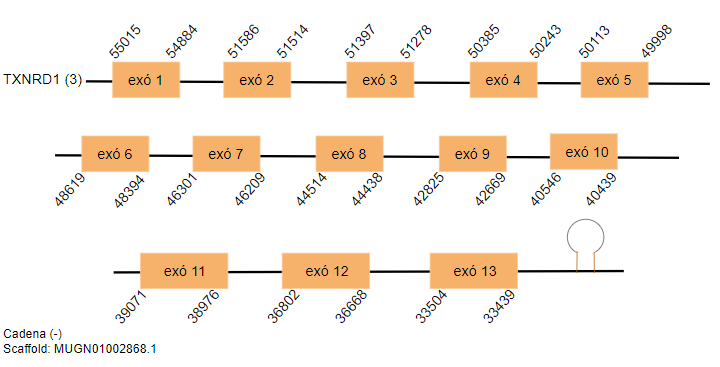
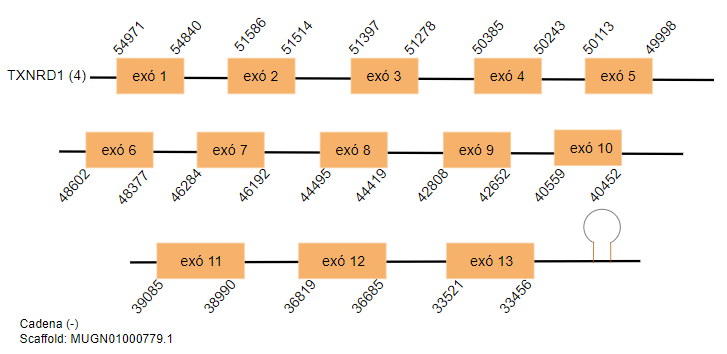
TXNRD2
517 aminoàcids constitueixen la proteïna TXNRD2 al genoma de Gallus gallus. S’han pogut predir 2 hits al genoma de Taenopygia gutatta. Aquests, es troben als scaffolds MUGN01002432.1 i MUGN01000423.1. Es troben a la cadena no reversa i estan formats per tretze exons. S’ha predit un SECIS per cada un dels hits tot i que a posicions massa allunyades de l’extrem 3’UTR.
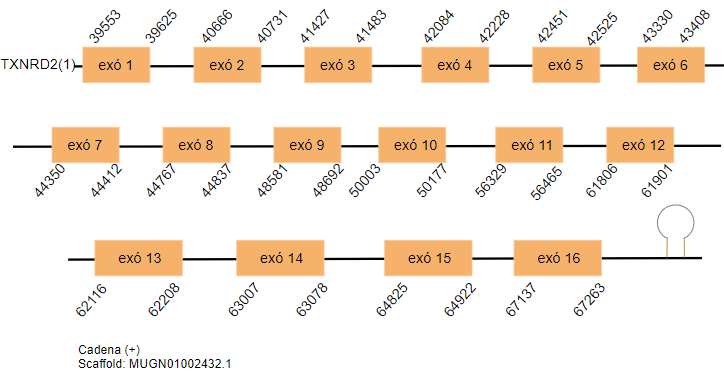
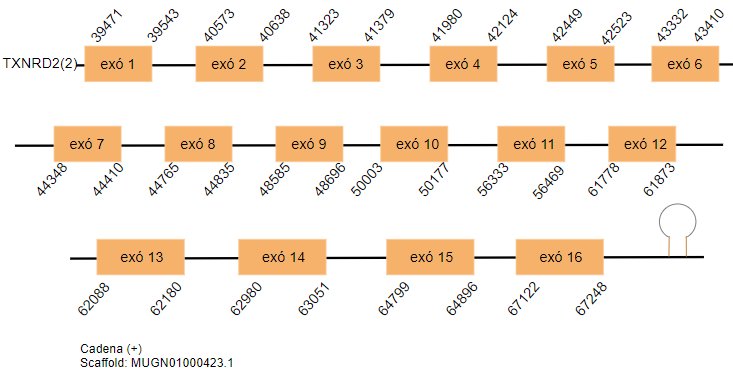
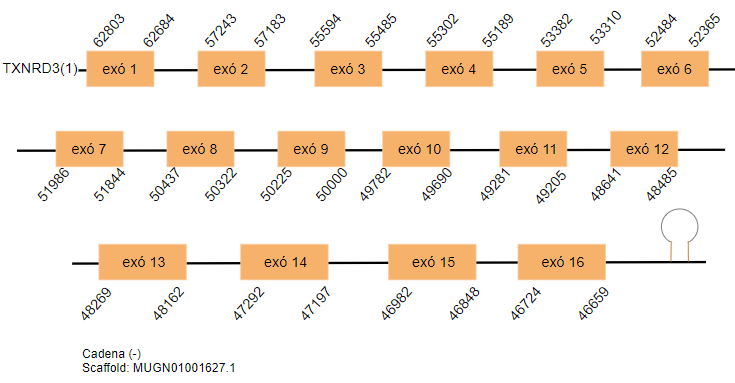
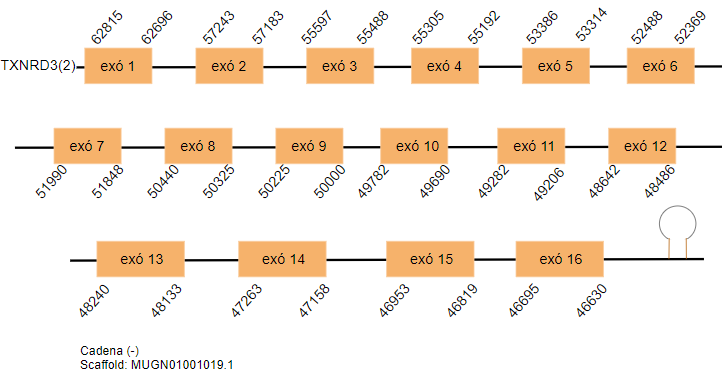
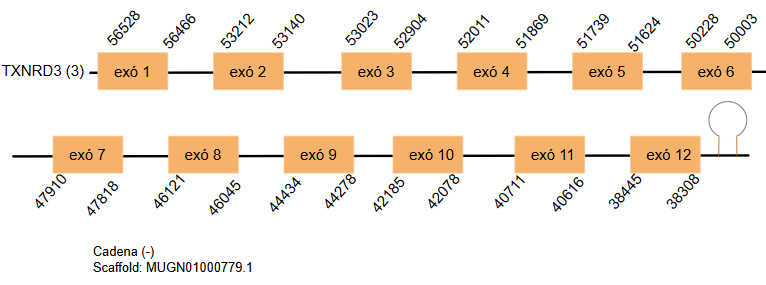
TXNRD3
Aquesta proteïna consta de 606 aminoàcids al genoma de Gallus gallus. S’han pogut predir tres hits significatius localitzats als scaffolds següents: MUGN01001627.1, MUGN01001019.1 i MUGN01000779.1. Tots ells es troben a la cadena reversa. El primer i segon hit tenen 16 exons cada un, el tercer hit conté 13 exons.
El SECIS s’ha predit a tots tres hits amb les condicions esmentades en el mètodes.
El primer i el segon hit són una duplicació.