INTRODUCCIÓN
las selenoproteínas:
El selenio fue descubierto por el químico sueco Jöcob Berzelius en el año 1817, y es un micronutriente esencial para los humanos y animales, ya que su déficit puede provocar enfermedades como la enfermedad de Keshan. Por otra parte, un exceso de selenio puede resultar tóxico. El selenio forma parte de la selenocisteína (Sec, U), el aminoácido 21 que podemos encontrar en las selenoproteínas, y está codificado por el codón de terminación UGA en el DNA. No existe un pool de Sec libre en las células, por lo que se utiliza un mecanismo inusual para descodificar el codón STOP UGA del mRNA e incorporar la Sec de manera cotranslacional al polipéptido que se está sintetizando. Una de las razones por las que no existe este pool de Sec libre es que existe un riesgo de que por error se incorpore Sec en vez de cisteína (Cys). Otra razón es que la Sec, al igual que otros compuestos de selenio como el selenito, reacciona con el oxígeno y promueve la formación de especies reactivas del oxígeno (ROS). En el genoma humano han sido identificados 25 genes que codifican para las diferentes selenoproteínas. [1]
La Sec se diferencia de la Cys en un átomo, es decir, contiene selenio en vez de azufre. Presentan propiedades químicas similares, pero la Sec presenta un valor de pKa menor y una nucleofilicidad mayor, cosa que hace que sea mucho más reactiva. [2]
Familias de selenoproteínas:
Las selenoproteínas actúan en las reacciones redox, en la defensa antioxidante, en la fertilidad, en el metabolismo de la hormona tiroidea, en el desarrollo del músculo y en las respuestas inmunitarias. Por ello, se han relacionado con enfermedades como el cáncer, la infertilidad, las infecciones virales y anormalidades del sistema inmunitario o del funcionamiento de la hormona tiroidea. Sin embargo, la función exacta de muchas selenoproteínas es aún desconocida.
Las familias de selenoproteínas más estudiadas y mejor caracterizadas son las glutation peroxidasas (GPx), tioredoxin reductasas (TR) y yodotironin deyodinasas (DI). Participan en las reacciones redox, y la Sec es un residuo del centro activo esencial para la actividad catalítica. Dentro de estas familias, se ha visto que TR1, TR2 y GPx4 tienen además un papel en la embriogénesis.
TRs: Como hemos dicho participan en la defensa antioxidante. Se han identificado 3 TRs en mamíferos: la TR1 en el citosol o núcleo, la TR3 en la mitocondria, y la tioredoxin glutation reductasa en los testículos. El mecanismo de acción propuesto consiste en la reducción del disulfuro N-terminal por la NADPH a través de la FAD, y la consiguiente transferencia de electrones del grupo tiol del N-terminal al C-terminal y de aquí a los diferentes sustratos. Así, las TRs están involucradas en el control de la proliferación celular, la viabilidad y la apoptosis mediante el control de la actividad de la tioredoxina y su estado redox. De hecho, son las únicas enzimas capaces de reducir la tioredoxina oxidada. La tioredoxina es la encargada de transferir electrones a la rebonucleótido reductasa, que convierte los ribonucleótidos en deoxiribonucleótidos, paso esencial para la síntesis de DNA. Además, también participa en muchas vías de señalización controlando la actividad de factores de transcripción que contienen cisteínas en su dominio de unión al DNA.
GPx: Son la familia más grande de selenoproteínas en vertebrados, y son los componentes más importantes de la defensa antioxidante humana. Existen 8 tipos distintos en humanos: las GPx1-3 catalizan la reducción del peróxido de hidrógeno y los hidroperóxidos orgánicos, mientras que la GPx4 reduce los fosfolípidos y los hidroperóxidos de colesterol. Además, la GPx4 está involucrada en la maduración de los espermatozoides y la fertilidad de los hombres, ya que se ha visto que es el componente principal de la cápsula mitocondrial de los espermatozoides maduros.
DIs: Son proteínas integrales de membrana de 29-33 kDa y comparten similitud estructural. Existen 3 tipos: DIO1 y DIO3 se localizan en la membrana plasmática mientras que la DIO2 reside en la membrana del retículo endoplasmático. Las DIO1 y DIO2 catalizan la desyodación de la hormona tiroidea T4 a la hormona activa T3, y también son capaces de transformar la T3 reversa en 3,3-diyodotironina. Por otra parte, la DIO3 transforma la T4 en T3 reversa inactiva y también la T3 en 3,3-diyodotironina.
Selenoproteína de 15 kDa (Sel15): Implicada en el plegamiento de las glicoproteínas en el retículo endoplasmático. [1]
Selenoproteína H (Sel H): Se sitúa en el núcleo y se cree que tiene un papel como antioxidante.
Selenproteína I (Sel I): Presenta actividad etanolamina fosfotransferasa, pero no se conoce del todo su función.
Selenoproteína K (Sel K): Se expresa predominantemente en el corazón y en el músculo esquelético pero otros tejidos como el páncreas, el hígado o la placenta también tienen niveles detectables de SelK. Está localizada en el retículo endoplasmático y la membrana plasmática y su sobreexpresión disminuye los niveles de ROS y protege a los cardiomiocitos del estrés oxidativo. Por ellos, se cree que tiene un papel antioxidante en el corazón. [3]
Selenoproteína M (Sel M): Se expresa principalmente en el cerebro donde puede tener una función redox.
Selenproteína N (Sel N): Es la única selenoproteína que está directamente relacionada con una enfermedad. Varias formas de miopatías de aparición temprana se deben a mutaciones en el locus del gen de SelN (SEPN1). Entre ellas están la distrofia muscular con espina rígida, la enfermedad multiminicore y la miopatía relacionada con desmina. Por tanto, podemos decir que juega un papel vital en el tejido muscular. [3]
Selenoproteína O (Sel O): Se desconoce su función.
Selenoproteína P (Sel P): Es una selenoproteína con múltiples residuos de Sec por subunidad proteica, y la encontramos en el plasma humano. Su papel principal es el transporte de selenio desde el hígado hasta los tejidos, principalmente al cerebro y a los testículos. Un papel adicional sería el de quelante de metales pesados o antioxidante. [1]
Methionine-R-sulfoxide reductase (Sel R): Cataliza la reducción de los residuos de metionina oxidados. Esta actividad es esencial para evitar el estrés oxidativo y el daño proteico. [1]
Selenoproteína S (Sel S): Participa en el control de la respuesta inflamatoria mediante la regulación de la producción de citoquinas por parte de los macrófagos. También se relaciona con la diabetes tipo 2 y enfermedades cardiovasculares. [3]
Selenoproteína T (Sel T): No se conoce su función.
Selenoproteína U (Sel U): En humanos distinguimos 3 tipos: SelU1, SelU2 y SelU3. La primera se expresa en hueso, hígado, riñón y cerebro.
Selenenoproteína V (Sel V) y Selenoproteína W (Sel W): Se desconocen sus funciones, pero la primera sólo se expresa en los testículos mientras que la segunda se expresa en diferentes órganos pero sobretodo en el músculo esquelético y en el corazón. Además, existen 2 tipos de Sel W en humanos: la SelW1 y la SelW2. [4]
Methionine sulfoxide reductase A (MsrA): Se encarga de la reducción del sulfóxido de metionina a metionina, de manera que su función podría ser repara el daño oxidativo de las proteínas. Se expresa mayoritariamente en el riñón y en el tejido nervioso.
Las siguientes forman parte de la maquinaria de síntesis de selenoproteínas que explicaremos en el siguiente punto:
Proteína de unión a RNA de 43 kDa (SECp43)
Selenocistein sintasa (SecS) Phosphoseryl-tRNA kinase (Pstk) Selenofosfato sintetasa 1 y 2 (SPS 1 y 2) SECIS binding protein 2 (SBP2) Factor de elongación Sec-específico (eEFsec)Síntesis de selenoproteínas:
El selenito y el selenato de los alimentos y del agua son usados por las células de los mamíferos como fuentes de selenio, y el selenito es reducido a seleniuro por las glutaredoxinas y las tioredoxinas. El selenurio también puede ser formado a partir de la selenometionina y la Sec de la dieta gracias a la acción de la liasa, y por tanto, también puede ser usado como fuente de selenio para la biosíntesis de Sec.
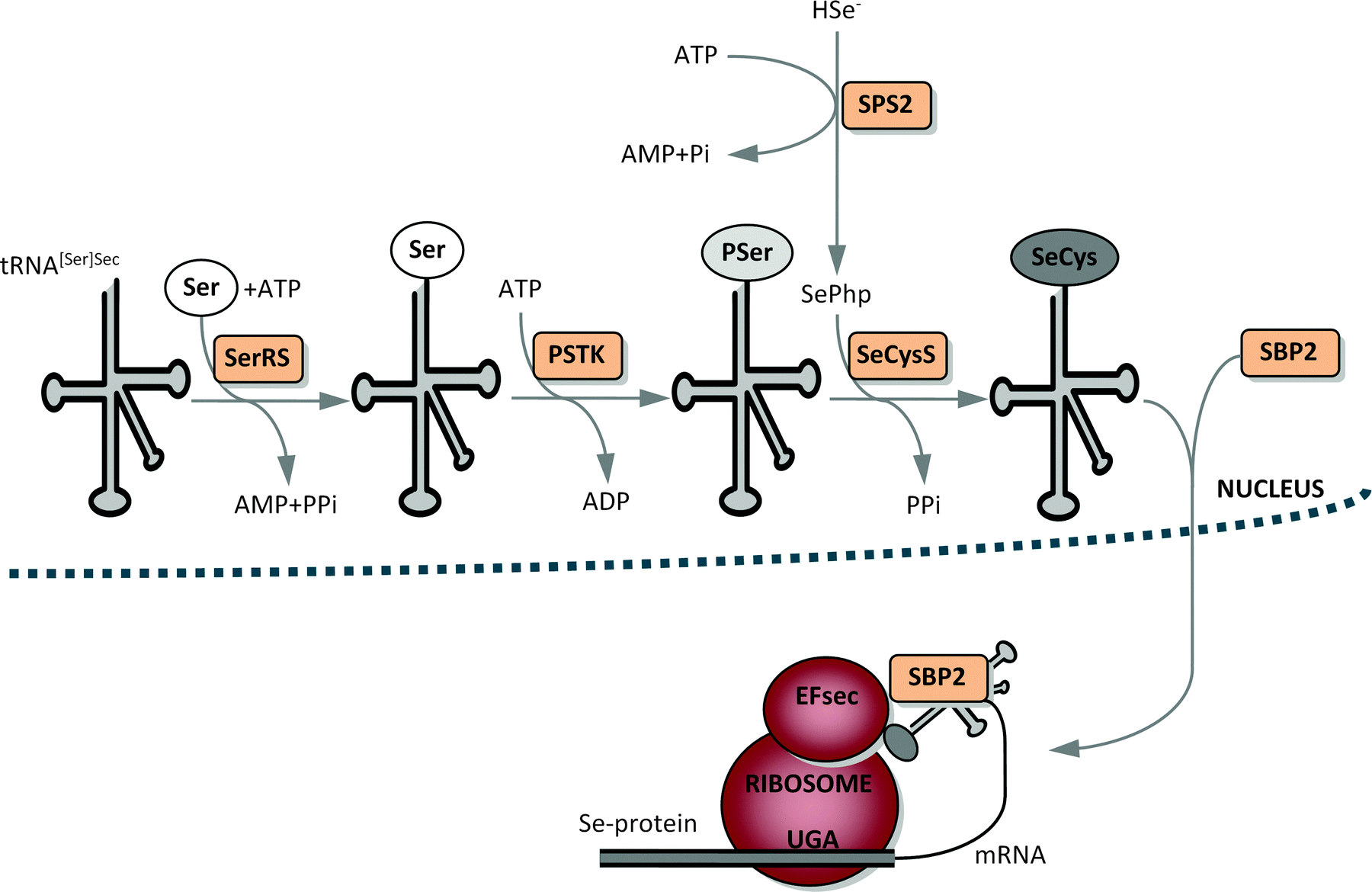
En primer lugar, la seril-tRNA sintetasa añade una serina al Sec-tRNA de manera que se formará la seril-tRNAsec. La SPS2 convierte el selenito o el selenurio en monoselenofosfato, que es el donador activo de selenio en el proceso de conversión de la seril-tRNAsec en Sec-tRNAsec por la SecS. [1]
Después, encontramos diferentes elementos que actúan como maquinaria de síntesis de selenoproteínas. Éstos son los encargadas de incorporar la Sec al polipéptido en el lugar codificado por el codón UGA:
- Elementos SECIS (SElenoCysteine Insertion Sequence): Es una estructura secundaria constituida por 2 hélices específica de la región 3' untranslated (UTR) del mRNA de las selenoproteínas. El pie de la hélice II está constituido por 4 pares de bases consecutivos que no siguen las reglas de Watson y Crick denominados cuarteto, y son esenciales para la incorporación de la Sec. Existen 2 estructuras secundarias posibles para los SECIS y dan lugar a las formas 1 y 2. La segunda contiene una hélice III adicional, pero presenta un loop apical más corto. La forma 2 es más abundante, pero no podemos asegurar que proporcione ninguna ventaja a la hora de incorporar selenocisteínas. De hecho, los mRNA que codifican la misma selenoproteína pueden presentar la forma 1 o la 2 dependiendo de la especie animal.
- SBP2 y Proteína ribosomal L30: Se unen al RNA SECIS mediante el mismo dominio de unión. El complejo SBP2/SECIS recluta al eEFsec, encargado de llevar la Sec-tRNAsec al centro decodificador del ribosoma. Así, la Sec se incorpora a la cadena peptídica y se continúa la traducción.
- Proteína de antígeno soluble del hígado/hígado pancreas (SLA/LP) y SPS1: Interaccionan y son redistribuidas hacia el núcleo gracias a la SECp43.
- Pstk: permite la fosforilación de la serina. [5]
La síntesis de selenoproteínas está regulada mediante el control del estado redox del SBP2, y las tioredoxinas y glutaredoxinas son las encargadas de dar electrones al SBP2 oxidado. [1]
La filogenia de las selenoproteínas:
Las selenoproteínas existen en las arqueas, las bacterias y los eucariotas, pero no en todas las especies de estos reinos. Las levaduras y las plantas superiores por ejemplo no presentan selenoproteínas, y en su lugar encontramos homólogos de cisteína. [1]
Las selenoproteínas de los mamíferos pueden clasificarse en 2 grupos: las relacionadas con el estrés y las housekeeping. Estas últimas están menos influenciadas por la cantidad de selenio de la dieta y participan en funciones críticas para la supervivencia celular. En cambio, las selenoproteínas relacionadas con el estrés no son esenciales para la supervivencia y presentan una expresión disminuida en situaciones de déficit de selenio. [4]
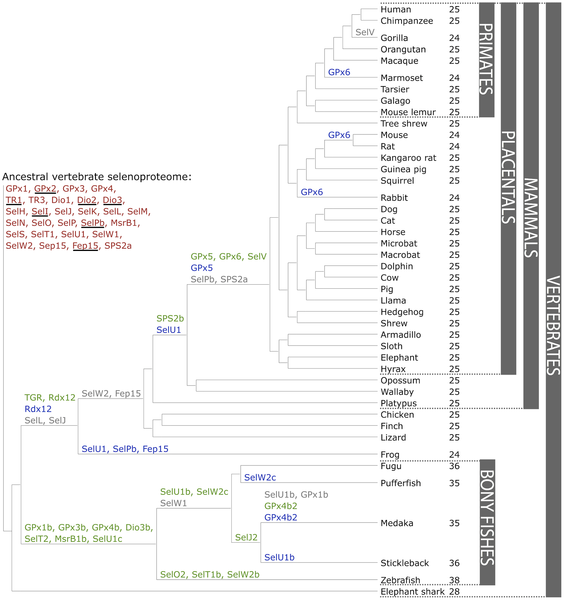
Encontramos 28 selenoproteínas ancestrales de vertebrados (y 24/25 de mamíferos). Concretamente, existen 21 selenoproteínas que se encuentran en todos los vertebrados: GPx1-4, TR1, TR3, DI1, DI2, DI3, SelH, SelI, SelK, SelM, SelN, SelO, SelP, MsrB1, SelS, SelT1, SelW1, Sel15. El resto de selenoproteínas las encontramos sólo en determinados linajes, lo que indica que se han generado nuevos genes de selenoproteínas por duplicación (verde), o se han perdido (gris), o bien han reemplazado su Sec por Cys (azul). Este reemplazamiento se consigue por una sola mutación puntual que transforma el codón UGA de la Sec en un codón de Cys.
Por otra parte, hay diferentes eventos evolutivos destacables en el selenoproteoma de los mamíferos:
- La pérdida de SelPb.
- La conversión de Sel U1, DI3 y GPx6 a homólogos de cisteína en diversos linajes.
- La duplicación de la TR1 encontrada en peces para generar la TR1 y la tioredoxin glutation reductasa de los mamíferos.
- La duplicación de la Sel W y la GPx3 para dar lugar a la Sel V y la GPx6 respectivamente. Además, esta última ha perdido la Sec varias veces.
- La generación de GPx7 y GPx8 a partir de la GPx4 (la más antigua) antes de la separación entre mamíferos y peces.
- El reemplazamiento del gen multiexónico SPS2a por el gen de único exón SPS2b.
Además, cabe destacar la pérdida de Sel L y Sel J en todos los vertebrados excepto en peces, y la pérdida de Sel W2 excepto en peces y ranas.
Para acabar, un dato interesante es que la Sel T es la selenoproteína más conservada en los mamíferos, mientras que la menos conservada es la Sel V. [4]
Identificación de selenoproteínas
El codón que codifica para el aminoácido selenocisteína, es el codón UGA, usualmente un codón STOP. Por ello, los métodos computacionales tradicionales de predicción de genes no sirven para identificar selenoproteínas y estas suelen estar mal anotadas.
Mediante la búsqueda de elementos SECIS se han podido identificar nuevas selenoproteínas, aunque es difícil cuando se aplica a secuencias genómicas ya que el patrón SECIS se encuentra en una alta frecuencia. Para reducir el número de falsos positivos, Castellano et al, desarrollaron un método mediante la correlación de la predicción de elementos SECIS y genes donde el codón bias característico de las regiones codificantes va más allá del codón UGA. [7]
Nosotros, nos basaremos en la búsqueda de homología entre selenoproteínas ya anotadas en genomas de otras especies y nuestra especie (Eidolon helvum). Los genomas utilizados serán Pteropus vampyrus, ya que esta especie se encuentra evolutivamente próxima a Eidolon helvum, y Homo sapiens, ya que es la especie donde las selenoproteínas están mejor anotadas.
Cabe tener en cuenta, que a la hora de buscar homología, tenemos que utilizar el DNA genómico de la especie que queremos estudiar, ya que es posible que la secuencia de proteínas esté mal anotada, ya que se puede encontrar truncada debido al codón STOP.
Así que compararemos la secuencia proteica de la proteína conocida con la secuencia genómica de nuestra especie. Para llevarlo a cabo ejecutaremos un BLAST que realizará alineaciones locales entre las dos secuencias. En este punto, deberemos elegir el Scaffold con el mejor hit, pero no solo podemos tener en cuenta, el mejor e-value sino que tenemos que estar seguros que contiene nuestro aminoácido U (previamente cambiado por X).
Una vez seleccionado el Scaffold tenemos que pasar el DNA genómico a cDNA y este a proteína. Aquí nos encontramos con otra dificultad ya que deberemos elegir entre las 6 pautas posible de lectura (3 forward y 3 reverse).
Después ya podemos correr los programes de alineamiento t coffe y genewise. Aquí nos encontramos con la dificultad de que en una misma familia de proteínas, al ser estas muy homólogas entre sí, pueden compartir los mismos hits (scaffolds). Sin embargo, no puede ser ya que se trata de proteínas diferentes. Así que aunque tengamos un scaffold muy bueno para dos proteínas diferentes de la misma familia deberemos elegir a qué proteína pertenece.
La limitación principal de la búsqueda de selenoproteínas por homología es que sólo podemos encontrar selenoproteínas que ya han sido identificadas en otros genomas, en ningún caso selenoproteínas de novo. Para ello se han desarrollado Nuevos métodos computacionales como SECISearch3 y Seblastian. El primer método es una combinación de los nuevos métodos de predicción de estructures de RNA (Infernal y Covels) y el original SECISearch. Seblastian sirve para predecir selenoproteínas en secuencias nucleotidicas, es capaz de predecir tanto selenoproteínas conocidas como nuevas selenoproteínas. [8]
Nosotros utilizaremos estos métodos para corroborar las selenoproteínas que encontremos conservadas en el genoma de Eidolon helvum.
Eidolon helvum
Taxonomía:
| Reino: Animalia Filo: Chordata Clase: Mammalia Orden: Chiroptera Suborden: Megachiroptera Género: Eidolon Espécie: Eidolon helvum |
 |
En general, los murciélagos se dividen en dos subórdenes; los megamurciélagos (megachiroptera), que a grandes rasgos se caracterizan por ser herbívoros o bien frugívoros, usar su vista y olfato para localizar el alimento y poseer orejas simples, y los micromurciélagos (microchiroptera), que se distinguen de los megamurciélagos por seguir una dieta más variada (pueden ser también carnívoros), poseer orejas complejas y grandes y por localizar el alimento a través de la ecolocación (que consiste en la emisión de sonidos e interpretación del eco procedente de los objetos que se encuentran alrededor).
Eidolon helvum, nuestra especie de murciélago objeto de estudio, pertenece al suborden de los megamurciélagos, al igual que Pteropus vampyrus, otra especie de murciélago próxima filogenéticamente a Eidolon helvum, que hemos usado como modelo para identificar las selenoproteínas de Eidolon helvum. [9]
Características generales, descripción física:
Eidolon helvum es una de las especies más grandes de entre las especies de megamurciélagos frugívororos; típicamente mide entre 12-20cm de largo, y sus alas pueden llegar a medir hasta 91cm. Esta especie presenta cierto dimorfismo sexual, siendo los machos ligeramente mayores que las hembras.
Como curiosidad, Eidolon en griego significa fantasma, haciendo referencia a la aparición nocturna de esta grande especie de murciélago, y helvum en latín significa pardo, haciendo alusión al color del pelaje de este mamífero.
A pesar de su nombre, el color de esta especie no sólo varía en función de las distintas partes corporales de un mismo individuo sino que también es variable entre distintos individuos, aunque en general Eidolon helvum posee un color que va desde el amarillo grisáceo hasta un color sepia grisáceo más oscuro, siendo el cuello y la espalda más típicamente amarillenta y la parte ventral más marronosa.
Las alas de este murciélago se caracterizan por ser muy largas, de color negro amarroneado, acabadas en punta y sobretodo muy finas, la cual cosa les permite recorrer grandes distancias sin necesidad de aletear demasiado ahorrando así energía.
E. helvum posee un pelo fino en casi todo el cuerpo y más grueso en el cuello, aunque las orejas, las alas y la zona alrededor de los ojos están ausentes de pelaje. La esperanza de vida de este animal es de aproximadamente 15 años [10].
Alimentación:
Eidolon helvum es hervíboro, típicamente frugívoro, es decir, básicamente se alimenta del jugo de las frutas y también de flores. Se alimenta de noche y en grupo, usando la vista y el olfato para localizar el alimento, y su dieta no varía a lo largo del año. Debido a su alimentación, esta especie puede ser nociva para las plantaciones y cultivos.
Depredadores:
Aunque hay muchos y distintos animales que pueden cazarlos, como algunas serpientes, algunos mamíferos o pájaros carnívoros como el halcón, en general la depredación de Eidolon helvum no suele amenazar sus poblaciones. En algunas zonas del oeste de África y en la República Democrática del Congo Eidolon helvum es consumido por humanos. [11] [12]
Hábitat/distribución:
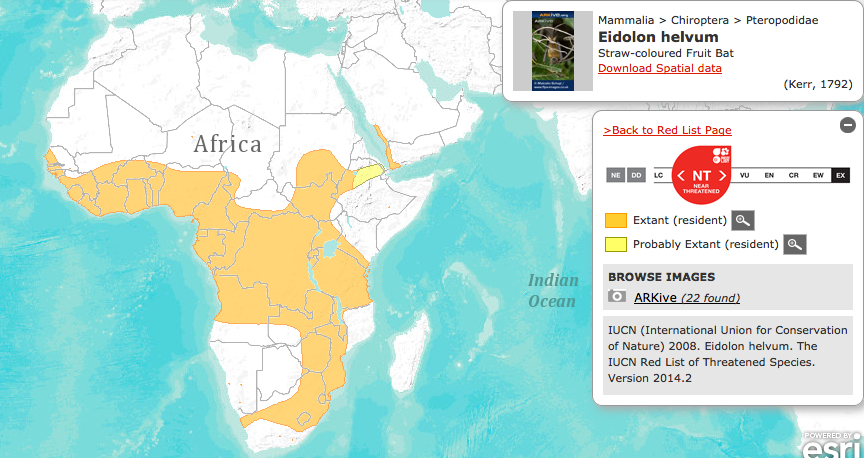
Eidolon helvum es el murciélago frugívoro más ampliamente extendido en África. Básicamente se encuentra en el centro y sureste africano, concretamente en zonas forestales tropicales y en sabanas subsaharianas, aunque también lo encontramos en el suroeste de la península arábiga y en Madagascar.
Es un murciélago migratorio, que habitualmente se localiza en bosques tropicales del centro de África, y migra a regiones circuncidantes más hacia el norte o hacia el sur en función de la disponibilidad de alimentos. [9]