Selenoproteïnes en Plasmodium vivax
Clara Conill, Estel Enreig, Albert Gil, Carlos González, Jordina Guillén
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
SECp43
SECp43 és una proteïna involucrada en la maquinària de síntesi de selenoproteïnes. SECp43 conté dos dominis d'unió a ribonucleoproteïnes mitjançant els quals es reconeix RNA. Així, es podrà unir a tRNA Sec. Per altra banda, SECp43 també s'uneix a SLA formant un triple complex. Sembla ser que SECp43 normalment resideix al nucli, però SLA té unes senyals putatives de NLS i NES que li permeten translocar-se. D'aquesta manera, quan SECp43 està al nucli, unit a SLA i tRNA Sec, forma un complex multiproteic que permet la síntesi de selenoproteïnes.
Experiments recents han demostrat que SECp43 també estaria involucrat a la regulació dels nivells de metilació de tRNA Sec, com també s'ha vist que podria tenir un paper important a la formació o estabilització del complex EFSec-SBP2-tRNASec i promoure la formació i localització citoplasmàtica (gràcies també a SLA) del complex SPS1/SLA/SECp43.
RECERCA DE SECp43 EN Plasmodium vivax
SECp43 és una proteïna de les menys conegudes en maquinària de síntesi de selenoproteïnes, i per tant, no la vam trobar a SelenoDB. Mitjançant una recerca acurada a l'NCBI, vam trobar la SECp43 de Drosophila melanogaster (NP_608837). Amb aquesta proteïna vam fer un TBLASTN contra el nostre genoma des de la terminal. Els resultats obtinguts van ser els següents, on el resultat més significatiu va ser el primer, amb un e-value de 9e-10.
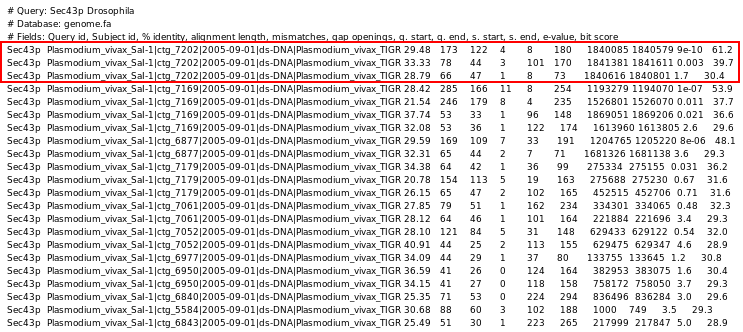
|
| Figura 1. Resultats TBLASTN SECp43 |
| Descarrega't l'arxiu |
Fixant-nos en l'output del BLAST, veurem com el tres primers corresponen al mateix contig (ctg_7202). Això ens va fer pensar que el gen ocupava una regió bastant gran, la qual cosa ens va fer tallar uns 5000 aminoàcids del contig per poder passar aquesta regió al GeneWise. Tot seguit s'adjunta l'output del GeneWise on es mostra la predicció del gen. Així, veiem com la predicció és d'un gen amb dos exons i dos introns.
Un cop feta la predicció, vam voler comprovar si hi havia SECIS o no, tot i que SECp43 és un proteïna de maquinària que en cap espècie és selenoproteïna. Efectivament, passant el SECISearch a la seqüència tallada (suficientment llarga com per abarcar el SECIS) no n'hi vam trobar cap.
Arribats a aquest punt vam estudiar amb més deteniment la proteïna predita pel GeneWise. Primer de tot, el que veiem és que no comença amb metionina, cosa que ens fa pensar que deu estar abans. De fet, dos aminoàcids abans hi ha una metionina, i set més endarrere en tenim una altra. Per altra banda, mirant la seqüència genòmica corresponent intentem buscar el codó STOP més proper, i la nostra sorpresa és que acaba molt més enllà de la predicció. De totes maneres segur que haurà de ser aquest ja que no n'hi ha cap altra abans. Així, el que fem és descarregar-nos de PlasmoDB l'arxiu de tots els transcrits de Plasmodium vivax i procedim a fer un TBLASTN des de la terminal amb la nostre proteïna predita contra la base de dades de tots els transcrits.
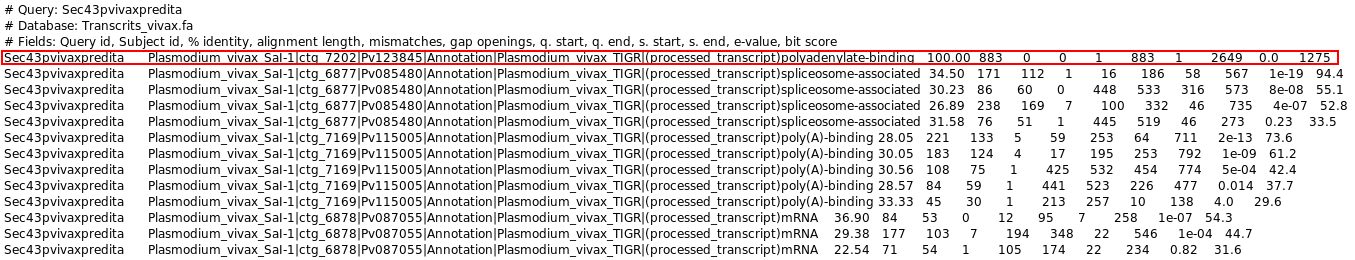
|
| Figura 2. Resultats TBLASTN transcrits |
| Descarrega't l'arxiu |
Així, veiem que tenim un hit molt bo que correspon al contig que ja havíem deduït, concretament al transcrit amb referència: Pv123845. El primer que comprovem és que el transcrit comença en aquella metionina que estava 9 aminoàcids enrere de la predicció de GeneWise, i per altra banda comprovem que el final del transcrit es troba en aquell codó STOP que havíem predit que estava tan allunyat de l'inici en comparació amb la predicció de GeneWise.
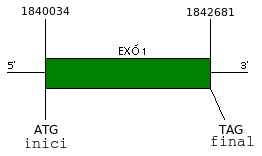
La nostra sorpresa més gran és que fent un alineament amb T-COFFEE entre el transcrit i el cDNA de la proteïna predita per nosaltres veiem que l'intró que havíem predit realment és codificant (veure l'alineament). Una possible hipòtesi que donem és que aquella regió no estigui tan conservada ja que no és de vital importància per la funcionalitat del gen (d'aquí que el GeneWise l'hagi predit com un intró), però que per altra banda sí que ho és pel correcte plegament, i per tant, per la proteïna en sí. Així doncs, gràcies aquest últim BLAST deduïm que la seqüència consta d'un únic exó de 2652 bp i una proteïna de 883 aminoàcids. Tot seguit es mostra la informació del gen en format .gff.
# seqname source feature start end score strand frame attributes # Plasmodium_vivax_SaI-1|ctg_7202|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome gene 1840034 1842682 4589 +
. gene_id 1 ; sequence Sec43pvivaxpredita ; gene_orientation . Plasmodium_vivax_SaI-1|ctg_7202|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 1840034 1842682 . +
. Plasmodium_vivax_SaI-1|ctg_7202|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 1840034 1842682 . +
. insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_7202|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome similarity 1840034 1842682 4589
+ . alignment_id 1 ; Query Sec43pvivaxpredita ; Align 1840034 1 2649
| Figura 3.Exonerate en format .gff |
Per corroborar la nostra hipòtesi procedim a estudiar els dominis que té la nostra proteïna amb InterProScan. Així, si comparem els dominis que té la SECp43 de Drosophila melanogaster veiem que té pràcticament els mateixos dominis; entre ells aquells dominis necessaris per la unió a RNA (PF00076, SM00360, PS50192).
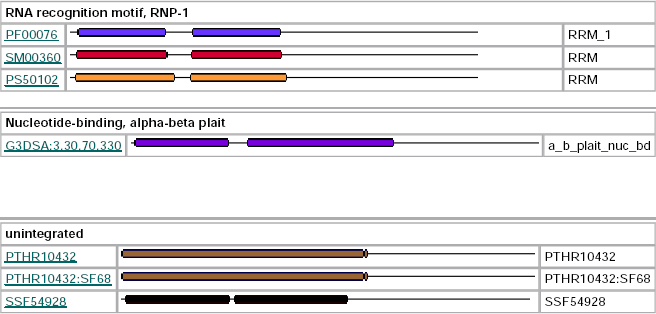  |
| Figura 4. A l'esquerre, dominis de Drosophila melanogaster. A la dreta, dominis de Plasmodium vivax |
Finalment, per altra banda procedim a fer un TBLASTN de la nostra hipotètica proteïna contra una base de dades de mRNA de Plasmodium vivax des de NCBI per veure si realment la nostra proteïna és expressada en aquest organisme, i els resultats són òptims; obtenim un e-value de 0,0.
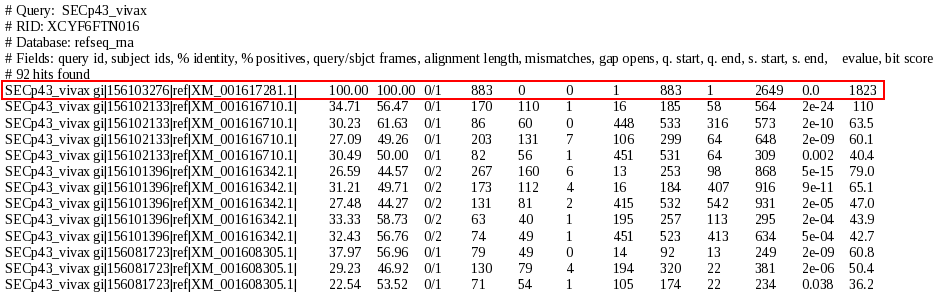
|
| Figura 5. Resultats TBLASTN mRNA |
| Descarrega't l'arxiu |
Un cop fetes les corroboracions esmentades, la nostra predicció de SECp43 per Plasmodium vivax és la segŁent:
|
| Figura 6. Esquema de SECp43 de Plasmodium vivax |