
La seqüenciació del genoma humà ha permès estudiar la variació de la seqüència en diferents poblacions, posant especial atenció en els SNPs.
Els SNPs (Single Nucleotide Polymorphisms) són canvis d'un sol nucleòtid en els dos al·lels, que apareixen amb una freqüència major del 1% en una població.
Se'ls atribueix la major part de la variació dins d'una població.
Des que es van començar a mapejar els SNPs, cada any ha augmentat notablement el seu coneixement, per exemple, fa tres anys es coneixien vora uns 300.000 SNPs en humà i en el dia d'avui se n'han mapejat més de 9 milions.
Aquest gran interès per a conèixer els SNPs és degut a que aquests petits canvis nucleotídics poden tenir implicacions biomèdiques importants:
- si apareixen en regions codificants poden provocar una alteració de la funció proteica causant l'aparició de determinades malalties
- la presència de SNPs en llocs d'splicing pot afectar al processament de mRNA, donant lloc a productes alterats
- el seu coneixement pot convertir-se en una eina de diagnòstic de les malalties que causen
- aquests canvis poden suposar des d'un augment de la susceptibilitat a determinades malalties com a una resposta diferent al tractament per determinats tipus de fàrmacs.
- poden ser útils com a marcadors biològics si es troben prop de determinades mutacions
- són els causants de la gran variabilitat entre individus i poblacions, fent que no troben dues sequències idèntiques
- l'anàlisi d'aquests polimorfismes pot ajudar a establir la història evolutiva de les poblacions
- la comparació entre espècies pot contribuir a estudis de conservació de sequències i d'evolució de les espècies
És per tot això que hi ha diverses organitzacions que es dediquen a descobrir i caracteritzar els SNPs per tal de conèixer millor la relació dels SNPs i la biomedicina, alguns exemples en són :
- SNP Consortium és fundada el 1.999 que mapeja els SNPs en el genoma i està associada a grans indústries tecnològiques del moment.
- La Universitat de Washington que detecta SNPs relacionats amb les respostes immunològiques en humans per tal de descobrir-ne les vies alterades.
- El Projecte Genoma Humà, que ha publicat el mapa del genoma humà complet i l'objectiu del qual és completar-lo localitzant els SNPs.

El coneixement de la distribució dels SNPs representa una eina molt potent per a l'anàlisi genètic i ens permetrà descobrir l'associació dels loci i els llocs específics del genoma són causa de malalties.
Mentre el nostre coneixement de la seqüència genòmica i de les seves variacions vagi augmentant, serem capaços de determinar més fenotips en comú a malalties més complexes.
De la mateixa manera, comparant les seqüències genòmiques corresponents a diferents espècies, es podran identificar regions ben conservades que reflecteixen les funcions i la regulació de gens ben conservats.
Tot això, ens permet accelerar la interpretació de la seqüència del genoma humà.
Amb aquest treball doncs, volem contribuir una mica més a conèixer les propietats d'aquests canvis tan petits que ens permeten diferenciar-nos entre nosaltres.
Els programes que hem creat tenen com a finalitat estudiar la distribució dels SNPs dins del genoma, tant en l'humà com el de ratolí, permetent la comparació entre els dos.
A més de la distribució en els cromosomes, les regions que analitzarem es mostren en el gràfic següent:
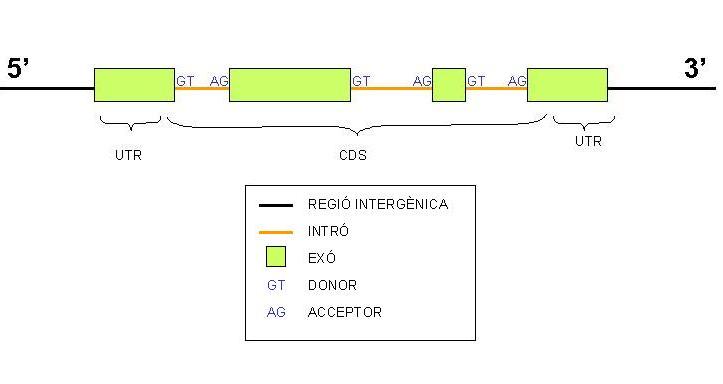
D'aquesta manera, un cop obtingudes les dades que ens proporcionarà el programa, que són el nombre de SNPs en cada cromosoma i regió gènica, podrem ser capaços d'arribar a determinar diversos aspectes dels SNPs i dels gens com ara:
- com és la distribució de SNPs en les diferents regions del genoma i dels cromosomes
- la variabilitat genètica i la conservació de determinades regions del genoma i dels cromosomes
- les diferències interespecífiques (entre humà i ratolí)

| Introducció | Materials i mètodes | Anàlisi de dades | Conclusions |
