Iron Response ElementS
![]() ESTADO DE ARTE DEL TEMA
ESTADO DE ARTE DEL TEMA
![]()
![]() ESTRUCTURE DE LOS IREs
ESTRUCTURE DE LOS IREs
Los
IREs han sido identificados en un gran
número de organismos (vertebrados,
invertebrados
y bacterias, pero no plantas)
y como se ha podido observar recientemente las proteínas
relacionadas con el metabolismo del hierro y el oxígeno no són las únicas que
cuentan con este motivo estructural en sus mRNAs. Ciertas proteínas housekeeping
para estrés térmico, proto-oncogenes,
proteínas de unión a la cllorofila
y algunas ribonucleoreductasas también contiene IREs en sus mensajeros.
Inicialmente se penso que los IREs, 5' and 3',
eran estructuralmente idénticos debido a la similitud de
estructura secundaria. Sin embargo comparaciones entre un gran número de
secuencias con IREs procedentes de diferentes mRNAs han mostrado que existen
cambions nucleotídicos específicos entre las secuencias que conferirían una
interacción ligeramente diferente con las IRPs y por tanto una regulación
individualizada. Pueden por tanto definirse varias isoformas de IREs en función
del patrón nucleotídico presentado.
-
Estructura primaria de los IREs: La
comparación de IREs animales ha mostrado que la conservación de la secuencias es
mucho más importante entre especies para el mismo mRNA que entre
diferentes mRNAs pero dentro de la misma especie (90% vs 36-85%).
A pesar de las diferencias, todos los IREs cuentan con entre
26 y 30 nucleotidos
encontrando una secuencia nucleotídica central,
CAGWUN,
y un residuo de C
(cisteina) cinco pases upstream,
totalmente conservados.
En la ferritina aparece un conjunto adicional de bases
conservadas, MGC,
que como veremos posteriormente cuentan con una función concreta en cuanto a la
vía de regulación se refiere.
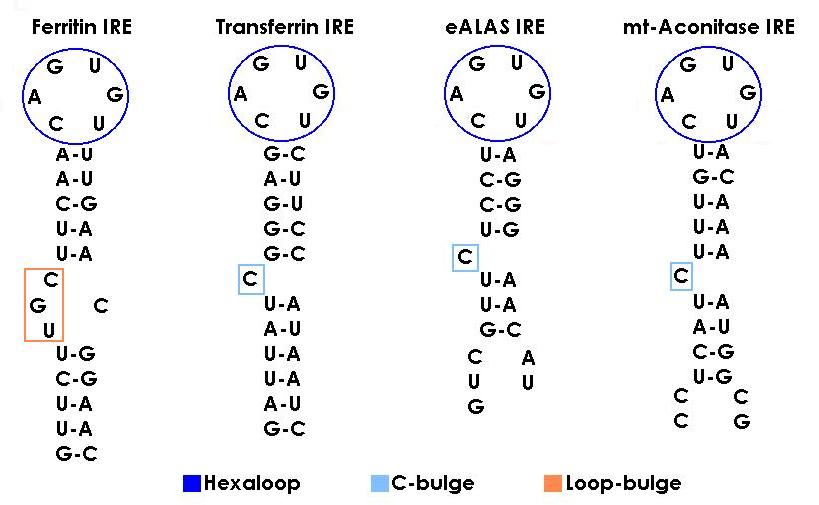
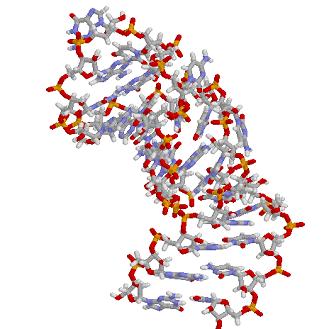
- Estructura secundaria de los IREs: La secuencia CAGWGX antes mencionada se pliega en un hexaloop terminal en el que la C1 y la G5 forman un apareamiento necesario para que se produzca la unión con las IRPs. Se ha podido comprobar que la substitución de esta C-G por A-U previene la unión tanto de IRP-1 como IRP-2. El patrón conformacional que proporciona al IRE el apareamiento C-G es probablemente crucial para la interacción RNA-proteína. Bajo el hexaloop el IRE cuenta con un tallo de 9 o 10 pares de bases apareadas que se pliegan en una a-hélice que siempre es interrumpida bien por el residuo de cisteina antes mencionado, conocido como bucle C (presente en la transferrina por parte de las proteínas del metabolismo del hierro, y en eALAS y la aconitasa mitocondrial por parte de las del metabolismo oxidativo), o por la secuencia MGC conocida también como bucle loop (solamente presente en el mRNA de la ferritina).
Experimentos recientes han determinado a través de las técnicas de mutagénesis dirigida que los residuos circundantes al bucle de C o loop también contribuyen a la interacción con las IRPs así que, no todas las substituciones nucleotídicas se encuentran permitidas.
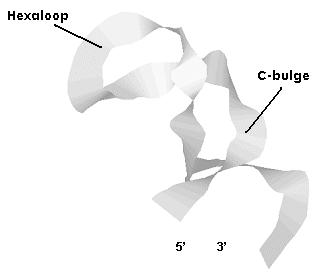
La estructura mostrada corresponde a la transferrina. Recordar que el bucle C se encontraría
substituido por el bucle de tipo loop en el resto de los mRNAs con IREs.
- Estructura terciaria de los IREs: La espectroscopía de RMN ha mostrado que los apareamientos C-G alredor del hexaloop CAGWGN hexaloop empujan a los residuos AGW hacia el solvente, lo que facilita la unión de las IRPs con el IRE. Ambos tipos de bucles, el C y el loop, potencian gracias a la distorsión introducida en la hélice, la formación de puntos de contacto entre las bases y las ribosas. Además del ésto el bucle loop de las ferritinas acoge un átomo metálico lo que selectivamente posivilita la unión a IRP-2. Ésto hace que centremos nuestra atención en lo importante que resulta la estructura primaria en el plegamiento del mensajero y como consecuencia en la conformación tridimensional que va a mediar la unión diferencial a proteínas o elementos.