INTRODUCCIÓ
Selenoproteïnes
Característiques de les selenoproteïnes
Una selenoproteïna és un pèptid que incorpora a la seva estructura una selenocisteïna ja sigui a la primera meitat de la seqüència d'aminoàcids, sent la part essencial del lloc actiu intern de la proteïna, o al final de la proteïna, servint com a cofactor en reaccions d'oxidació-reducció (redox). Aquestes proteïnes apareixen tant en eucariotes com en bactèries i arquees. La selenocisteïna es considera l'aminoàcid número 21 i està codificada pel triplet UGA, que també codifica per un codó STOP. No obstant això, si el codó UGA apareix a dins de la seqüència codificant i incorpora una seqüència anomenada SECIS (seqüència d'inserció de la selenocisteïna), la selenocisteïna acabarà incorporant-se al pèptid format [1][2][3].
Les selenoproteïnes participen en processos d'oxidació-reducció, com a defensa antioxidant, en el metabolisme d'hormones tiroidees i en la resposta immune. Aquesta és la raó per la qual s'ha establert una alta correlació entre anomalies en les selenoproteïnes i malalties humanes com la malaltia de Keshan, infecciones víriques o en la funció de les hormones tiroidees [4][5].
Les selenoproteïnes poden classificar-se atenent a la localització de la Sec (Fig. 1) . Aquelles en que la Sec està situada a prop de l'extrem C-terminal pertanyen al grup I. Algunes d'aquestes són la metionin-R-sulfoxi reductases i les tioredoxina reductases. Les selenoproteïnes del grup II, en canvi, tenen la Sec situada a l'extrem N-terminal, com les selenoproteïnes de mamífers H, M, N, T, V, W, Sel15, selenofosfatasa sintetasa 2 (SPS2), la "deionidase thyroid hormone" i algunes peroxidases de glutatió (GPxs). Tot i això, la majoria són tioredoxines amb el motiu CxxC (dues cisteïnes separades per dos residus) al lloc actiu de l'enzim [2].
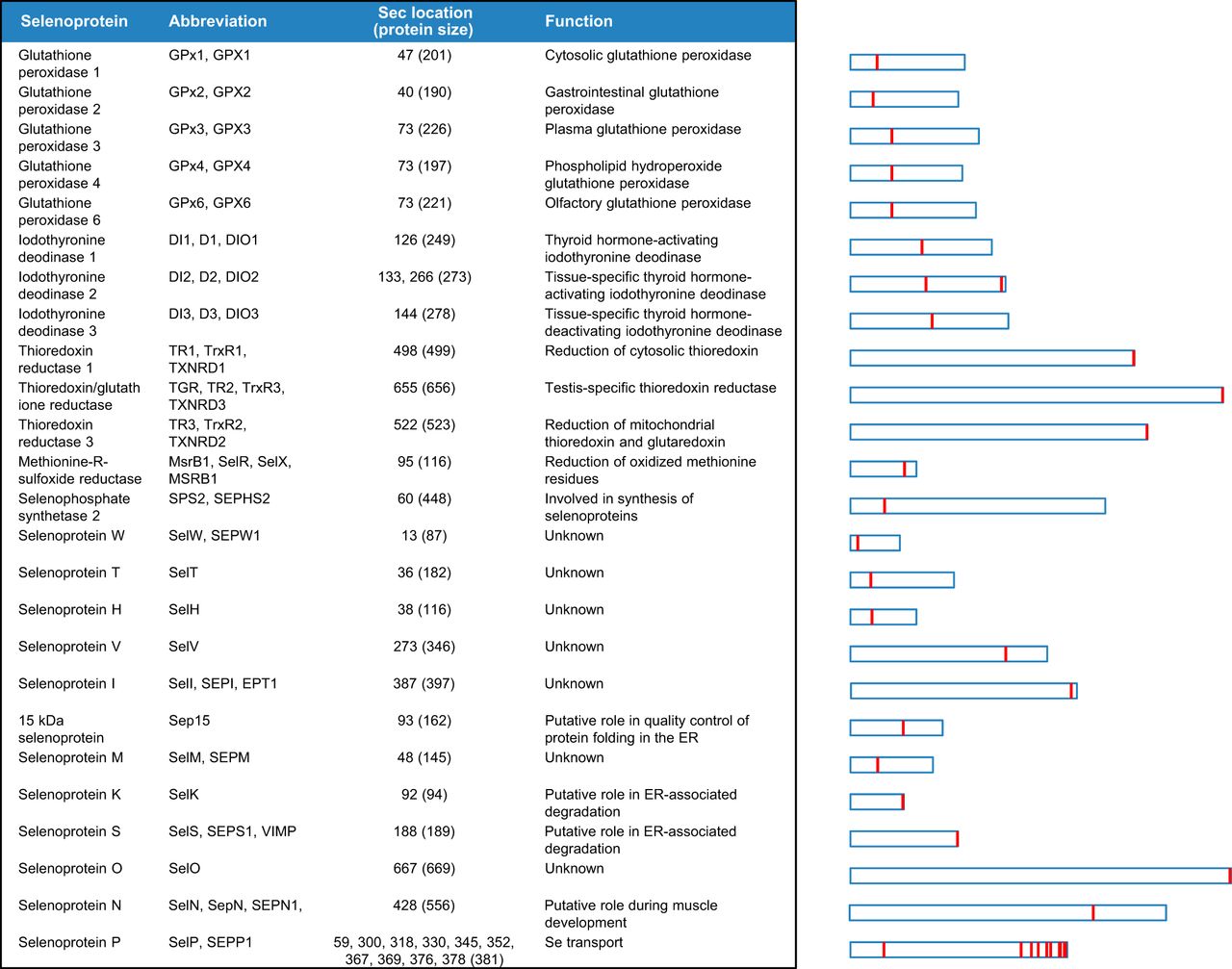
Figura 1. A l'esquerra, veiem el selenoproteoma humà. A l'esquerra l'allargada relativa de les selenoproteïnes i la localització del SEc en la seva seqüència. [2].
La major part de les selenoproteïnes d'un organisme es troben en forma de proteïnes ortòlogues que contenen cisteïna. Les selenocisteïnes són similars a les cisteïnes excepte pel fet que les primeres contenen seleni a la seva estructura en comptes de sulfur, característica que probablement els atorgui propietats úniques. Això podria explicar el fet que les selenoproteïnes s'hagin mantingut evolutivament al genoma malgrat les altes necessitats energètiques de la seva maquinària de síntesi [6][7].
Una característica distintiva de la selenocisteïna en comparació amb la cisteïna, és el seu potencial per tal de facilitar reaccions de transferència d'electrons. Una altra característica diferencial de la selenocisteïna és el seu pKa de 5.2, significativament més baix que el de la cisteïna, de 8.5, el qual facilita l'inici de les reaccions amb una taxa redox major quan el sistema no es troba en equilibri per part de la Sec. Tot i així, es creu que aquest potencial redox no es deu únicament a la presència de selenocisteïna sinó a la aparició de motius proteics amb funcions redox a les selenoproteïnes [8][9].
Diversos grups han dut a terme l'expressió de proteïnes recombinants en E.coli en les quals s'inseria Sec en comptes de la Cys al lloc actiu d'algunes tioredoxines. Malgrat que el potencial redox d'aquestes tioredoxines recombinants disminuïa, la seva activitat catalítica es veia incrementada i augmentava també la seva eficiència en l'intercanvi de grups ditiols i disulfats. A més a més, altres estudis amb glutaredoxina en d'E.coli, suggerien que l'intercanvi d'activitat no era propulsat pel menor potencial redox sinó per l'alt potencial nucleofílic de la selenocisteïna [10].
Elements SECIS
Com ja hem dit, la selenocisteïna està codificada pel mateix triplet que el codó stop (UGA). Degut a que el triplet codificant pot ser confós amb un codó stop, existeix una identificació incorrecta de les selenocisteïnes. Aquestes no estan ben anotades, motiu pel qual es dóna una pèrdua d'informació biològica i funcional molt valuosa. No obstant això, també és necessari l'element SECIS (Selenorprotein Insertion Sequence), una estructura secundària de mRNA que permet l'acció conjunta d'altres factors per tal de formar la selenoproteïna [2][3][11].
L'element SECIS és una estructura de RNA amb forma de loop que es situa a l'extrem 3' UTR. Aquest element permet la inserció de la selenocisteïna a l'interior del pèptid en el moment previ a la seva traducció. Això és necessari ja que quan la selenocisteïna es troba lliure, aquesta acaba trencant-se donant lloc a seleni i cisteïna, motiu pel qual no existeix un pool de selenocisteïna lliure a l'organisme. Una altra raó per la qual aquest pool no pot existir al medi és el risc de que la selenocisteïna s'incorpori erròniament al lloc de la cisteïna [2][3][11].
Els elements SECIS d'eucariotes i arquees tenen diferents seqüències (Fig.2), motius i estructures. En bacteris es situen a dins de la regió codificant, just a continuació del codó UGA que codifica per la Sec, mentre que els elements SECIS en eucariotes estan formats per dues hèlix separades per un loop intern, un quartet GA i un loop apical. El quartet GA es localitza a la base de l'hèlix II i està composta per quatre bases aparellades. Es tracta de l'element funcional més important del SECIS, ja que és necessari per a què aquest pugui interaccionar amb la SBP2, una proteïna de la maquinària de síntesi de la qual parlarem més endavant. A més a més, un loop intern gran afavoreix la interacció de SBP2. De la mateixa manera, els nucleòtids situats a les posicions 2 i 3 són els que influeixen en la afinitat de SBP2 per l'element SECIS. En alguns SECIS, el loop intern forma un mini-loop que s'utilitza per classificar els diferents elements SECIS en dos tipus. Els de tipus I, que contenen aquest mini-loop i els de tipus II, que no el contenen. Així mateix, al centre de l'element SECIS existeix un motiu conservat AAR essencial per tal de incorporar la Sec a la proteïna [2][3][11].
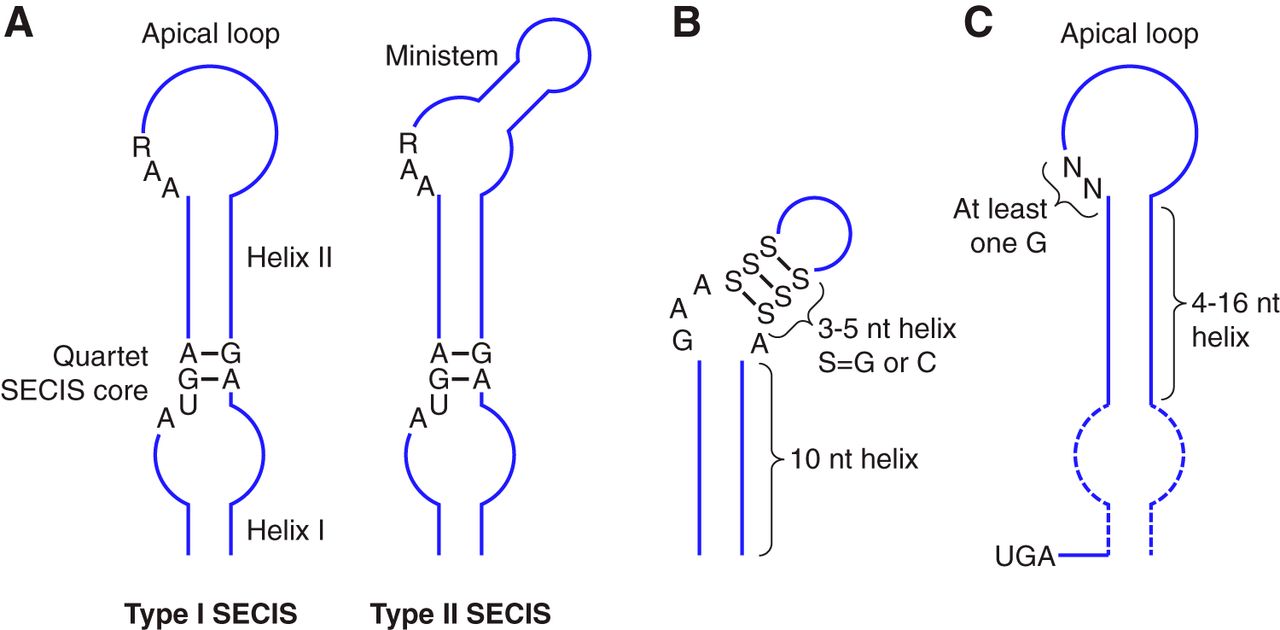
Figura 2. Estructura dels elements SECIS en (A) eucariotes, (B) arquees i (C) bacteris. Es mostren els nucleòtids conservats en la majoria de les seqüències de mRNA consens dels elements SECIS [2].
Síntesi de les selenoproteïnes
La síntesi de les selenoproteïnes (Fig.3) requereix d'altres factors a més de l'element SECIS: aquells que permeten la inserció de la selenocisteïna a l'interior del pèptid (SPS1/2, SecS, Pstk, Secp43), i els que permeten la síntesi física de la pròpia selenoproteïna (SBP2 i EFSec).
Quan una serina és carregada sobre un tRNA es forma el complex anomenat seryl-tRNA. Aquest pot ser directament convertit en una selenocysteinyl-tRNA[Ser]Sec, indirectament mitjançant un intermediari O-fosforilat en la creació del qual està involucrada una quinasa phosphoseryl-tRNA[Ser]Sec (pstk). Aquest enzim sintetitza un intermediari fosforilat denominat pSer-tRNA[Ser]Sec que serveix com a substrat de SecS [2][3][11].
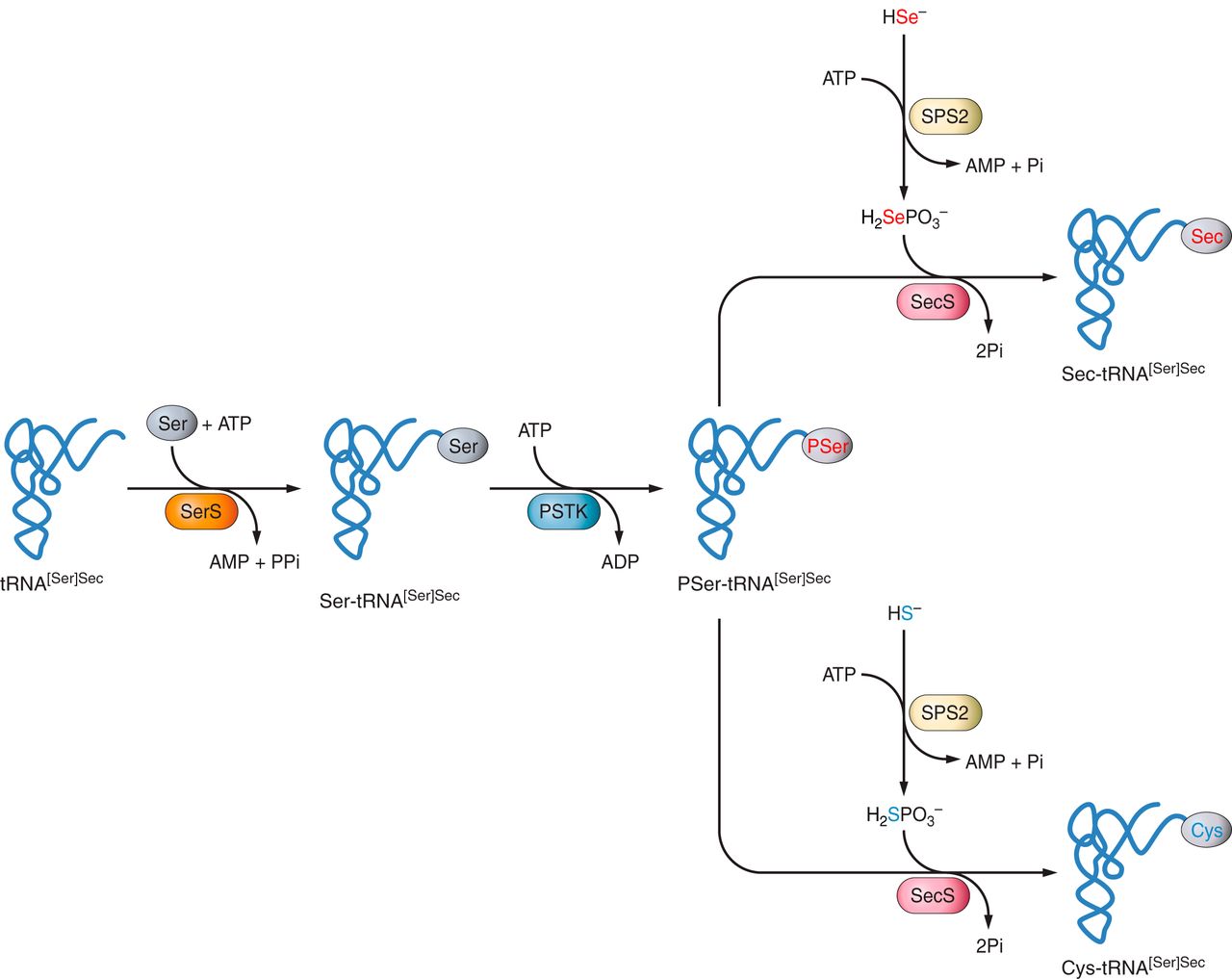
Figura 3. Mecanisme de biosíntesi de la selenocisteïna en eucariotes i via de síntesi de la cisteïna mitjançant la maquinària de formació de Sec [2].
A continuació, comença la síntesi a través de la catàlisi ocasionada per la selenofosfatasa sintetasa 2 (SBP2). El seleni és convertit en monoselenofosfat, el donant actiu del seleni que participa en la formació de la selenocisteïna. El monoselenofosfat (SPS) generat a parir del seleni i d'ATP, i és l'encarregat de donar seleni a la SecS. El Sec-tRNA[Ser]Sec es situa sobre el factor d'elongació (EFSec) i el mRNA s'uneix al ribosoma que, al seu torn, també s'uneix a SBP2. Degut a la distancia entre el UGA i l'element SECIS al 3'-UTR, qualsevol UGA que es trobi dins del marc de lectura pot ésser codificat com a selenocisteïna, ja que el factor d'elongació que roman unit al Sec-tRNA [Ser]Sec, serveix com a donador de selenocisteïna durant la traducció [2][3][11].
Un cop s'ha creat la selenocisteïna, per tal que aquesta es pugui incorporar a una selenoproteïna, es necessiten dues senyals principals (Fig. 4): un codó UGA al mRNA a dins de la seqüència codificant, i un element SECIS situat al 3' UTR en eucariotes, ja que en procariotes aquest també es troba a dins de la seqüència codificant. El complex ribosomal SBP2-SECIS, dirigit per la seqüència UGA, reconeix al conjunt format pel SectRNA[Ser]Sec i proporciona una selenocisteïna a la proteïna. A més, en eucariotes, existeixen altres factors que determinen l'eficiència d'inserció de les selenocisteïnes al pèptid, com la proteïna ribosomal L30, el factor d'iniciació eIF4a3 o la nucleolina factor d'elongació que roman unit al Sec-tRNA [Ser]Sec, serveix com a donador de selenocisteïna durant la traducció [2][3][11].
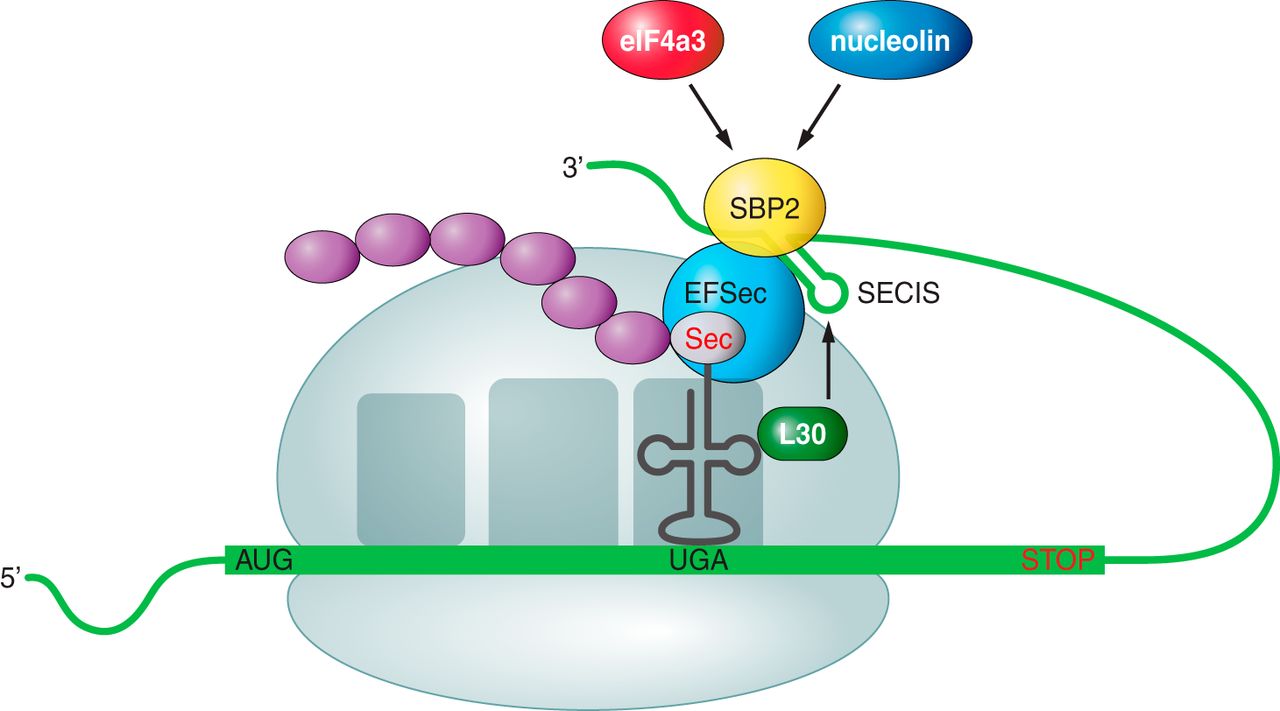
Figura 4. Representació dels factors que requerits per incorporar la selenocisteïna en resposta al codó UGA en eucariotes [2].
Mètodes d'identificació de les selenoproteïnes
En les primeres investigacions sobre les selenoproteïnes, la seva identificació es realitzava experimental a partir de l'anàlisi de proteïnes per tal de valorar l'existència de selenocisteïna mitjançant espectrometria de masses i la detecció de seleni radioactiu que s'incorporava metabòlicament a les proteïnes en forma de Sec. La primera selenoproteïna identificada va ser la Glutatió Peroxidasa 1 de mamífers (GPx1) [1].El nombre de selenoproteïnes identificades en els últims anys ha anat creixent de forma dràstica gràcies al desenvolupament de diverses eines bioinformàtiques. Tot i que la seva identificació sigui complexa degut a la doble interpretació possible del codó UGA, l'aparició dels elements SECIS, que tenen una estructura determinada en forma de loop en el seu mRNA, han estat de gran ajuda en aquesta tasca [12].
Per tal de superar aquesta limitació, s'han desenvolupat nous mètodes fonamentats en la predicció d'elements SECIS basant-se en la creació d'una puntuació que depèn de les prediccions de la estructura del RNA i que permet millorar significativament la sensibilitat. Les selenoproteïnes s'identifiquen buscant codons UGA que es troben a dins del marc de lectura de genomes que han sigut analitzats per complet. Degut a que la majoria de les selenoproteïnes contenen homòlegs en els que la selenocisteïna ha estat substituïda per cisteïna, aquesta estratègia permet la identificació de selenoproteïnes independentment dels elements SECIS.Recentment, també es va crear el programa SeBlastian (Fig. 5), una eina per la predicció i l'anàlisi dels gens de les selenoproteïnes que combina la tècnica d'identificació de les selenoproteïnes basada en elements SECIS amb la tècnica basada en els homòlegs de Sec/Cys [12].
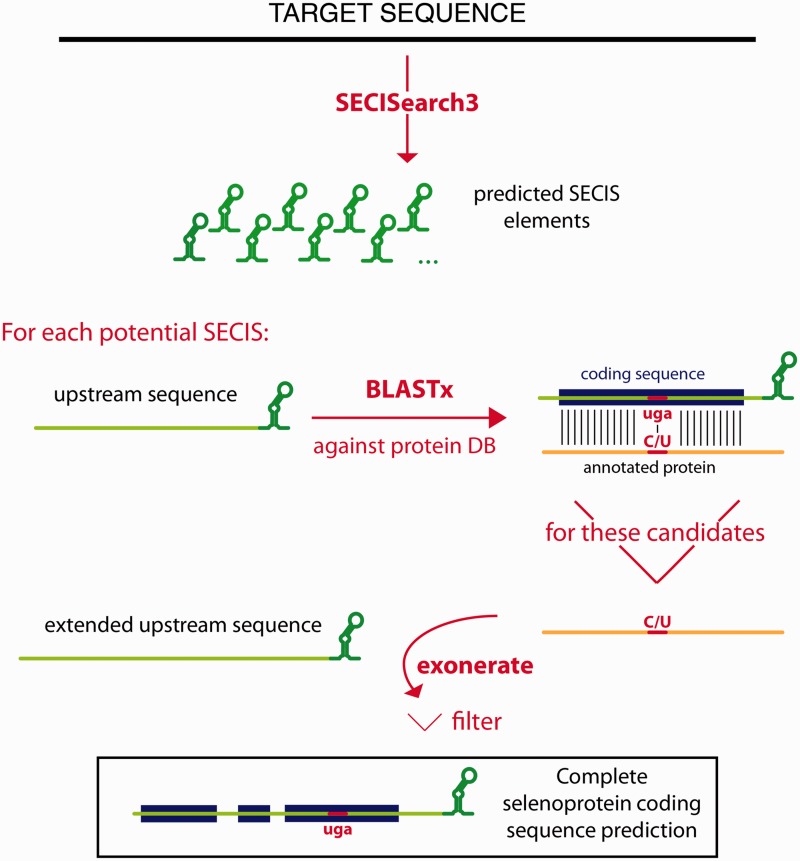
Figura 5. Seblastian com a exemple d'eina bioinformàtica per la identificació de proteïnes [12].
Evolució de les selenoproteïnes
La distribució de les selenoproteïnes varia enormement entre espècies. Malgrat que es distribueixen a tots els dominis eucariota, arquee i eubacteries, les selenoproteïnes han desaparegut totalment als fongs, les plantes superiors i algunes espècies d'animals com els escarabats, els cucs de seda i altres insectes [6][7].

Figura 6. Representació de les diferents famílies de selenoproteïnes. Les caixes agrupen aquelles selenoproteïnes que només es troben en vertebrats o en cèl·lules úniques d'eucariotes, mentre que en vermell es troben aquelles selenoproteïnes que es troben en organismes procariotes. A la dreta està representada la mida relativa de cada selenoproteïna i la localizació de la selenocisteïn en cada una d'elles [2].
La comparació d'organismes amb proteomes grans amb aquells en els quals el nombre de selenoproteïnes era força reduït, va rebel·lar que diferents grups d'organismes terrestres van reduir la utilització de selenocisteïna reemplaçant-la per cisteïna i donant lloc a homòlegs o perdent totalment aquestes selenoproteïnes. Contràriament, alguns organismes marins compten amb selenoproteomes de grans proporcions, fet que suggereix que l'ambient juga un paper important en l'evolució de les selenoproteïnes.La majoria de selenoproteïnes identificades es troben presents tant en eucariotes monocel·lulars com en vertebrats, fet que indica el seu origen ancestral. Cal destacar que els vertebrats, com altres grups, també van reduir el seu nombre de selenoproteïnes. Moltes d'aquestes van evolucionar mitjançant un procés de duplicació gènica [6][7].
El treball de Mariotti et al. (Fig. 7) va caracteritzar el selenoproteoma de diversos vertebrats. Els selenoproteomes més grans s'observaven en peixos ossis, mentre que els selenoproteomes més petits corresponien als de les granotes. De les 45 selenoproteïnes seqüenciades en vertebrat, 28 es trobaven en mamífers. Es van trobar 21 selenoproteïnes comunes a tots els vertebrats: GPx1-4, TR1, TR3, Dio1, Dio2, Dio3, SelH, SelI, SelK, SelM, SelN, SelO, SelP, MsrB1 (methionine-R-sulfoxide reductase 1), SelS, SelT1, SelW1, Sep15. Mentre que la resta únicament es trobava en alguns llinatges, subratllant el procés dinàmic pel qual algunes selenoproteïnes s'han generat per duplicació, mentre que d'altres ho han fet mitjançant el reemplaçament per cisteïna [6].
Destaca el fet que el selenoproteoma del cavall, es troba, molt relacionat filogenèticament amb el selenoproteoma de gats i de gossos, del qual hauria divergit recentment en l'evolució. Aquests selenoproteomes partirien d'un ancestre comú compartit amb els ratpenats [6].
Figura 7. El selenoproteoma ancestral dels vertebrats està indicat en vermell. Les selenoproteïnes que només es troben en vertebrats estan subratllades. Les noves selenoproteïnes creades tenen el seu nom escrit en verd, mentre que la seva pèrdua es troba en gris. Així mateix, les selenocisteïnes reemplaçades per cisteïnes apareixen en blau. A més a més, es mostra el nombre de selenoproteïnes esperades per a cada espècie [6].
Equus Przewalskii
El cavall de Przewalskii, cavall salvatge mongol o Takhi, és una subespècie de cavall salvatge, tot i que alguns autors suggereixen elevar-lo a la categoria d'espècie, Equus ferus przewalskii. És el parent vivent més proper dels cavalls domèstics (Equus caballus). Equus ferus przewalskii és un cavall que pertany al ordre dels Perissodactyla i la família dels Equidae. En el passat va quedar extint en estat salvatge però a la dècada del 2000 els esforços de conservació van permetre alliberar-ne una població salvatge d'uns 200 exemplars. La població total és d'uns 1.000 exemplars a tot el món.
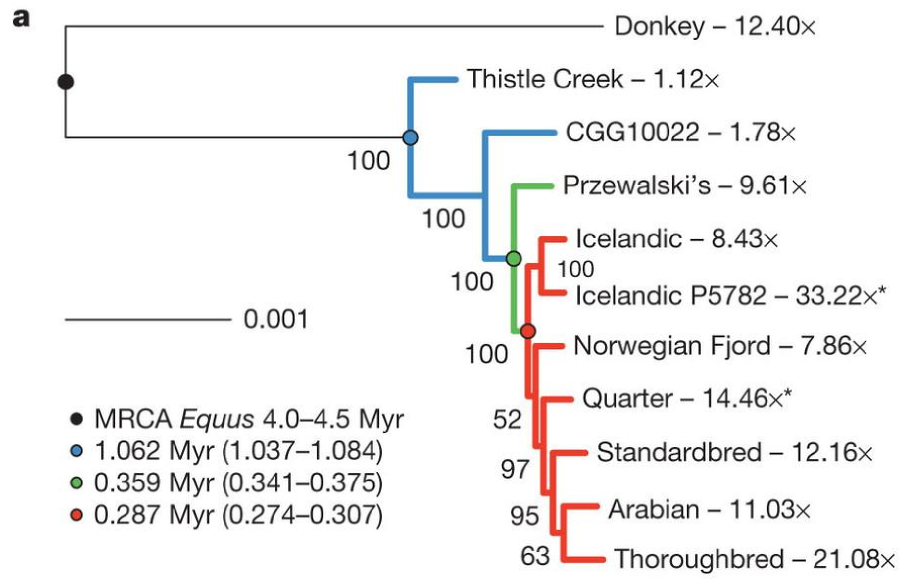
Figura 8. Filogènia del gènere Equus on es pot observar la proximitat d'Equus przewalskii amb el cavall domèstic (en vermell).
Descripció
Aquest cavall es diferencia de la major part dels seus congèneres domèstics per l'estructura del seu crani. Aquest presenta un morro convex, a diferència de la majoria de les races domèstiques, en què és còncau. És un cavall de mida petita, potes proporcionalment més curtes, estructura massissa, cua llarga i cap més gran en comparació a la resta del cos. La grandària típica d'un exemplar adult d'Equus przewalskii és de 2.1 metres de llarg i uns 350 quilos de pes.
Els cavalls de Przewalskii són animals socials que viuen en bandades de mida variable dirigides per un mascle dominant o semental, i en les quals la resta d'integrants són femelles amb cries menors de 2 anys. Quan superen aquesta edat, abandonen el grup: els mascles intentaran aconseguir la seva pròpia manada, mentre que les femelles s'integraran en altres grups, garantint així l'intercanvi genètic. Com qualsevol altre cavall, aquests animals s'alimenten essencialment d'herbes. Els seus principals depredadors són els llops.
Context genètic d'Equus przewalski
L'Equus przewalskii (EPR), és una espècie de cavall salvatge amb un nombre cromosòmic diploid 2n=66 mentre que el cavall comú E. Caballus (ECA) té un nombre cromosòmic diploid de 2n=64. Les discussions per la seva relació filogenètica i taxonòmica han portat a comparar les seves similituds morfològiques, proteiques i de DNA mitocondrial, la seva capacitat de procrear obtenint descendència híbrida fèrtil i a comparar la seva morfologia cromosòmica i de patró de bandes. Estudis anterior basant-se en les bandes GTC suggerien que els dos equins eren homòlegs i que les diferències en el nombre cromosòmic eren degudes únicament a una translocació Robertsoniana ocorreguda entre dos parells de cromosomes acrocèntrics en EPR i un parell de cromosomes metacèntrics en l'ECA. Finalment la Comissió Internacional de Nomenclatura Zoològica va considera Equus przewalskii i E. caballus com dues espècies diferents [13].
També podeu trobar tota la informació en castellà i en anglès.