DISCUSSIÓ
Molts estudis han demostrat que Equus przewalskii està estretament relacionat amb Equus caballus. Degut a aquesta proximitat filogenètica, per a la identificació de selenoproteïnes es va partir de les selenoproteïnes ja anotades de Equus caballus. En el moment en que amb Equus caballus no es generaven anotacions correctes i sorgien problemes en les prediccions, es va procedir a comparar-ho amb les proteïnes d'Homo sapiens, ja que és el genoma més ben anotat.
Selenoproteïnes
DI
Les Iodothyronine deiodinase s'expressen en tots els teixits i controlen l'activació i desactivació de les hormones tiroidees. En mamífers, aquesta família està formada per tres enzims: DI1, DI2, DI3. D2, expressada en el reticle endoplasmàtic, activa les hormones tiroidees permetent la conversió de la prohormona tiroxina (T4) a triiodotironina (T3) a nivell de teixit mitjançant l'eliminació del iode de l'anell exterior de T4. DI3, en canvi, és responsable de la seva inactivació als teixits perifèrics ja que participa en la seva conversió en triiodotironina reversa (Rt3). DI1, es localitza, com DI3, a la membrana plasmàtica i és capaç de realitzar ambdós processos a nivell plasmàtic [14][15][16][17].
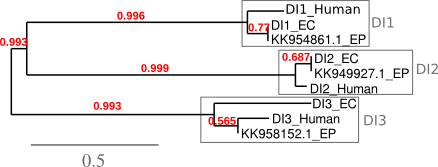 |
DI1
La seqüència predita compta amb 16246 nucleòtids (2619-18865). Està formada per 4 exons i 3 introns situats al scaffold KK954861.1. Així, aquest fragment codifica, en el sentit invers, per una proteïna idèntica a la DI1 d'Equus caballus. Aquesta presenta una selenocisteïna situada en el segon exó del gen que es troba conservada en Equus przewalskii. Això concorda amb el fet que el programa SECISearch3 trobi un element SECIS a l'extrem 3' de la cadena complementària on es situa el gen, també predit pel programa SeBlastian. Aquest SECIS, amb un grau A, es troba a una distància de 1291 nucleòtids de la selenocisteïna i de 922 de l'últim exó.
DI2
La proteïna DI2 es troba al scaffold KK949927.1. La seva seqüència és de 8560 nucleòtids (783350-791910) i està formada per 2 exons i 1 intró presents a la cadena forward. Les prediccions per Exonerate i Genewise coincideixen. La seqüència d'aminoàcids d'Equus caballus és també idèntica a la proteïna predita a partir del genoma d'Equus przewalskii. Aquesta conté una selenocisteïna present en ambdues espècies al segon i últim exó.
El programa SECISearch3 no ha trobat cap element SECIS, motiu pel qual no ha estat possible predir cap selenoproteïna mitjançant SeBlastian. Tot i així, el fet que la proteïna contingui una selenocisteïna contradiu aquesta falta de resultats.
DI3
En aquest cas, només hem aconseguit alinear el fragment final de la proteïna predita amb la proteïna DI3 d'Equus caballus. L'inici de la seqüència de nucleòtids del scaffold on suposadament es troba aquesta, no correspon a la posició de la seqüència d'aminoàcids d'Equus caballus sinó que correspon a l'inici del fragment truncat de la proteïna predita. En realitzar la comparació amb DI3 d'Homo sapiens, trobem el mateix problema. Així doncs, la impossibilitat d'obtenir un alineament complet és degut al fet que el fragment del genoma d'Equus przewalskii anotat en aquest scaffold no cobreix la proteïna completa.
GPx
Les peroxidases de glutatió són una àmplia família de proteïnes expressades en tots els teixits de mamífers que protegeixen a les cèl·lules de l'oxidació. Aquestes catalitzen la reacció de formació de disulfits de glutatió a partir de dues molècules de glutatió reduït. Com anirem veient, aquesta família inclou tant selenoproteïnes com homòlegs de cisteïna.
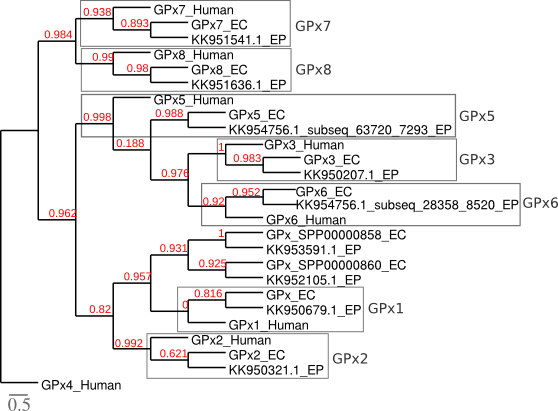
En mamífers s'acostumen a trobar 8 homòlegs de GPx, 5 dels quals són pròpiament selenoproteïnes (GPx1-4 i GPx6). Tot i això, en la base de dades, per a Equus caballus, hi ha anotades en canvi 9 proteïnes de la família GPx. A partir d'anàlisis de filogènia en mamífers s'han pogut identificar tres grups evolutius: GPx1/GPx2, GPx3/GPx5/GPx6, i GPx4/GPx7/GPx8 [18][19][20].
Degut a la complexitat d'aquesta família i la falta de correspondència d'algunes proteïnes d'Equus caballus amb les d'humans, hem realitzat la filogènia de la família per a analitzar més fàcilment els resultats. En la filogènia obtinguda veiem que l'agrupació correspon grups evolutius descrits anteriorment. Com podem veure a l'arbre a continuació, la proteïna GPx1 humana és propera a la proteïna d'Equus caballus amb id SPP00000859 i a la proteïna predita en el scaffold KK950679. Les proteïnes GPx2, 3, 5, 6, 7 i 8 d'humà corresponen a les proteïnes homònimes d'Equus caballus. GPx4 en canvi, és la proteïna humana més allunyada de les GPx en el gènere Equus, ja que no presenta semblances amb cap GPx ni d'Equus caballus ni amb les predites en Equus przewalskii. Cal posar especial èmfasi en la branca on trobem el grup GPx 3, 5 i 6 ja que es coneix que GPx5 i GPx6 són resultat d'una duplicació en tàndem de GPx3 a l'inici de la separació dels mamífers placentaris [6]. És per això que els casos de GPx5 i GPx6 s'han analitzat amb més detall.
En l'arbre també veiem dues de les proteïnes d'Equus caballus que no corresponen a cap de les GPx d'humans, però que presenten semblances amb el grup de GPx1/GPx2.
 |
GPx1
La proteïna es troba al scaffold KK950679.1. La seva seqüència és de 830 nucleòtids (321034-321864) i està formada per 2 exons i un intró presents a la cadena reversa. La seqüència d'aminoàcids d'Equus caballus és pràcticament idèntica a la proteïna predita a partir del genoma d'Equus przewalskii, excepte per un petit fragment de nucleòtids al primer exó que només estan presents en aquest últim. Aquest fet pot significar que s'ha produït una inserció en el cavall d'estudi, o bé pot ser degut a un error dels programes. La proteïna presenta una selenocisteïna situada en el primer exó del gen, que es troba conservada en Equus przewalskii. Això concorda amb el fet que el programa SECISearch3 trobi un element SECIS a l'extrem 3' de la cadena negativa on es situa el gen, també predit pel programa SeBlastian. Aquest SECIS, amb un grau A, es troba a una distància de 509 nucleòtids de la selenocisteïna i de 47 de l'últim exó.
GPx2
La Glutathione peroxidase 2 és una proteïna citosòlica depenent de seleni present als epitelis, especialment, en els epitelis pulmonar i gastrointestinal. Es troba implicada en l'activitat reductora del peròxid d'hidrogen mitjançant glutatió [18][19][20].
En Equus przewalskii, la proteïna és codificada per 2 exons separats d'un intró, situats a la cadena positiva del scaffold KK950321.1. La seqüència té una llargada de 3080 nucleòtids (7405331-743611). La proteïna resultant és idèntica a GPx2 d'Equus caballus, i, com aquesta, conté una selenocisteïna situada en el primer exó.
L'element SECIS d'aquesta selenoproteïna es troba a la mateixa cadena positiva, amb una separació respecte a la selenocisteïna de 673 nucleòtids i de 223 respecte a l'últim exó.
GPx3
La Glutathione peroxidase 3 és una proteïna plasmàtica sintetitzada predominantment als ronyons i que també es troba implicada en la detoxificació del peròxid d'hidrogen [18][19][20]. La proteïna predita és molt similar a GPx3 d'Equus caballus. En aquesta última trobem però un fragment absent a la proteïna d'Equus przewalskii. Una deleció podria explicar aquest fet. Una altra opció pot ser el fet que es tracti d'una proteïna amb splicing alternatiu. Tot i així, és probable que l'anotació de la proteïna o del genoma no siguin suficientment exactes i que, per tant, cap de les hipòtesis plantejades sigui correcta, i que els resultats obtinguts no siguin totalment fiables.
La seqüència que codifica per aquesta proteïna està formada per 2482 nucleòtids (205043-207525) presents al scaffold KK950207.1, i és llegida en sentit invers. En el primer exó trobem una selenocisteïna conservada respecta a Equus caballus. El seu element SECIS, de grau A, se situa, segons SECISearch3, a la mateixa cadena, a una distància de 1080 nucleòtids d'aquesta i a 618 de l'últim exó.
GPx5
La proteïna predita per Exonerate a partir de la GPx5 d'Equus caballus és pràcticament idèntica a aquesta malgrat que no s'ha pogut aconseguir el fragment inicial de la proteïna. El fragment predit, situat al scaffold KK954756.1 està codificat per 5299 nucleòtids (64717-70016) de la cadena forward. Aquesta seqüència nucleotídica compta amb 14 exons i 13 introns. Es tracta d'un homòleg amb cisteïna tant en els dos cavalls com en humans. El fet que el programa SECISearch3 no hagi estat capaç de trobar cap element SECIS indica que, probablement, el canvi de selenocisteïna a cisteïna ha portat a la pèrdua d'aquest element amb el pas del temps. En realitzar el procés mitjançant la query d'Homo sapiens, observem que la seqüència predita es troba a la mateixa regió genòmica. No obstant, tampoc se'n pot obtenir el fragment inicial.
GPx6
GPx6 es situa en el mateix scaffold (KK954756.1) que GPx5. Aquesta troballa no ens sorprèn ja que es tracta d'una duplicació en tàndem, i per tant, cabia la possibilitat que ambdues proteïnes es trobessin properes en un mateix scaffold. Destaca però el fet que es troben en cadenes oposades, situant-se GPx6 a la cadena reversa. De nou, la proteïna predita es troba truncada. No s'ha pogut obtenir el fragment inicial a partir de la query d'Equus caballus, ni d'Homo sapiens. La seqüència codificant del fragment alineat té una llargada de 6532 nucleòtids (29349-35881) i està formada per 4 exons i 3 introns. La selenocisteïna està conservada en ambdues espècies. Situada al primer exó del gen, presenta un element SECIS de grau A a la mateixa cadena, separat d'aquesta per 1012 nucleòtids, i a una distància de 568 nucleòtids de l'última regió codificant.
GPx7
La Glutathione peroxidase 7 també participa en la detoxificació del peròxid d'hidrogen. A diferència d'altres proteïnes de la família, aquest enzim és independent del seleni[18][19][20]. A més, no conté cap selenocisteïna; es tracta d'un homòleg amb cisteïna. Això concorda amb el fet que el programa SECISearch3 no hagi estat capaç de trobar cap element SECIS.
La proteïna predita tant per Exonerate com per Genewise presenta 3 exons i 2 introns compresos en 1791 nucleòtids (365318-367109) de la cadena forward, i es troba al scaffold KK951541.1. És pràcticament idèntica a GPx7 d'Equus caballus a excepció de dos aminoàcids.
GPx8
La Glutathione peroxidase 8, de la mateixa manera que GPx7, té el mateix efecte sobre el peròxid d'hidrogen, el seu mRNA no compta amb cap codó UGA (que codifica per selenocisteïna), i és independent de seleni [18][19][20].
Aquest homòleg de cisteïna es troba a la cadena forward del scaffold KK951636.1. La seqüència codificant està formada per 2 exons separats per un intró. La llargada del conjunt és de 3507 nucleòtids (117449-120956). La seqüència d'aminoàcids resultant és molt semblant a GPx8 d'Equus caballus a excepció d'un cert nombre d'aminoàcids.
Amb el programa SECISearch3 trobem un element SECIS de grau B, malgrat que el programa SeBlastian no prediu cap selenoproteïna. Aquest fet concorda amb la hipòtesi segons la qual GPx8 contenia una selenocïsteina que, amb el pas del temps, ha mutat a cisteïna, donant lloc a l'homòleg actual.
GPx none1
El scaffold seleccionat és el KK953591.1. La seqüència codificant es troba situat a la regió 156367-156516 del genoma. La predicció realitzada tant per Genewise com per Exonerate indica que el gen té una llargada de 149 nucleòtids, amb un sol exó. Aquest fragment codifica en el sentit invers per una proteïna pràcticament idèntica a la GPx None1 amb ID SPP00000858 d'Equus caballus. Presenta una selenocisteïna en el seu únic exó del gen, que es troba conservada en Equus przewalskii. L'element SECIS d'aquesta predit pel programa SECISearch3, de grau A, es troba a l'extrem 3'UTR de la mateixa cadena. Tot i això no ha estat possible obtenir una predicció amb el programa SeBlastian.
GPx none2
La seqüència predita tant per Exonerate com per Genewise compta amb 347 nucleòtids (226825-227172). Està formada per un sol exó situat al scaffold KK952105.1. Així, aquest fragment codifica, en el sentit forward, per una proteïna idèntica a la GPx amb ID SPP00000860 d'Equus caballus. Com Equus caballus, la proteïna d'Equus przewalskii tampoc conté selenocisteïna. Es tracta doncs de dues proteïnes homòlogues amb cisteïna. Tot i això, el programa SECISearch3 ha predit un element SECIS a la cadena positiva, encara present malgrat la pèrdua de la selenocisteïna. El programa SeBlastian, com cabria esperar, ho ha predit cap selenoproteïna.
MsrA
La methionine sulfoxide reductase A (MsrA) es tracta d'un homòleg amb cisteïna la funció del qual es relaciona amb la reparació del dany oxidatiu produït en proteïnes mitjançant la reacció de reducció del sulfòxid de metionina a metionina[21].
La proteïna MsrA es troba al scaffold KK950289.1. A partir de les prediccions d'Exonerate i Genewise s'obté una seqüència de 140255 nucleòtids (49419-189674) que consta de 4 exons i 3 introns a la cadena reversa. En aquest cas, en l'alineament veiem que la proteïna predita en Equus przewalskii és més curta que la proteïna d'Equus caballus. El programa SECISearch3 ha estat capaç de predir un element SECIS, fet que suporta la hipòtesi que MsrA era inicialment una selenoproteïna que va evolucionar canviant la selenocisteïna per la cisteïna, malgrat que encara conserva aquesta estructura associada a la selenoproteïna.
Sel15
La selenoproteïna de 15 kDa (Sel15) és una selenoproteïna amb funció redox, involucrada en el plegament correcte d'altres proteïnes en el reticle endoplasmàtic, on es localitza [22][23][24].
En el genoma de Equus przewalskii trobem una selenoproteïna homòloga a Sel15 totalment idèntica en el scaffold KK949903.1. La seqüència que codifica per aquesta proteïna és de 35907 nucleòtids (1961242-1997149) es troba en sentit invers. Conté 5 exons i 4 introns, la selenocisteïna es troba localitzada en el tercer exó.
A més, per aquesta proteïna s'ha pogut predir tant l'element SECIS, que es troba a una distància de 586 nucleòtids de l'últim exó del gen i de 793 respecte la Sec, com l'alineament amb SeBlastian.
SelH
La selenoproteïna H (SelH) és una proteïna amb un domini tioredoxina i un motiu Cys-X-X-Sec conservat. S'expressa en un gran nombre de teixits, se n'ha descrit una localització nuclear i una possible funció antioxidant [Dikiy et al.].
En el genoma d'Equus przewalskii s'ha trobat una proteïna homòloga a SelH d'Equus caballus codificada per 1587 nucleòtids (168188-169775) a la cadena reversa del scaffold KK951476.1. El gen conté 3 exons i 2 introns. La proteïna d'Equus przewalskii manté la selenocisteïna i presenta una gran homologia amb la d'Equus caballus. Tot i això, mitjançant el programa SECISearch3 no s'han pogut predir elements SECIS i per tant, tampoc s'ha pogut obtenir l'alineament amb SeBlastian.
SelI
La selenoproteïna I és una de les menys estudiades, per la qual cosa la seva funció no es coneix amb certesa. El que sí que s'ha observat és que conté un domini CDP-alcohol fosfatidiltransferasa, amb la qual cosa podria estar implicada en el metabolisme de lípids. Un tret característic d'aquesta proteïna respecte els seus homòlegs és la presència d'una extensió al C-terminal que conté la Sec [6].
La proteïna d'Equus przewalskii que presenta homologia amb SelI d'Equus caballus es troba al scaffold KK950007.1, codificada per una seqüència de 22520 nucleòtids (1077742-1100262) a la cadena en sentit invers. Tant la predicció de Genewise com d'Exonerate indiquen que aquesta seqüència conté 9 exons i 8 introns. En l'alineament s'observa que a la proteïna predita li manca una regió inicial ja que no comença amb metionina i s'alinea a partir de l'aminoàcid 18 de la proteïna d'Equus caballus. En ambdues proteïnes trobem la selenocisteïna pròxima a la regió C-terminal. No s'han pogut predir SECIS per aquesta proteïna.
SelK
La selenoproteïna K es localitza, tant a la membrana plasmàtica com al sistema de membranes del reticle endoplasmàtic. La seva expressió es dóna majoritàriament als cardiomiòcits, on és probable que, com moltes altres, actuï com a antioxidant [27].
La seqüència d'aminoàcids predita tant per Genewise com Exonerate utilitzant Sel K d'Equus caballus com a referència és només un petit fragment de la proteïna real malgrat la seva alta identitat. Per aquest motiu hem repetit l'anàlisi a partir de la proteïna, probablement millor anotada, d'Homo sapiens. La proteïna predita per Exonerate és molt similar a la Sel K humana. La seqüència obtinguda a partir de Genewise no pot ser utilitzada ja que el programa interpreta el codó TGA com a codó stop i no com a selenocisteïna, que es troba en realitat conservada en Equus przewalskii.
La seqüència de nucleòtids que codifica per Sel K és de 104 nucleòtids (203025-203129), fet que correspon amb el petit mida de la proteïna (16 kDa). Es troba localitzada en la cadena rerversa del scaffold KK951974.1. La selenocisteïna es situa al tercer exó, i el seu element SECIS, de grau B, es troba a la mateixa cadena. El programa SeBlastian no ha pogut predir cap selenoproteïna, motiu pel qual no podem conèixer a quina distància es troba la selenocisteïna d'aquesta estructura.
SelM
Sel M és una petita selenoproteïna que s'expressa al reticle endoplasmàtic cel·lular d'un gran nombre de teixits, i especialment a l'encèfal. Malgrat que la seva funció no es coneix amb exactitud, es creu que protegeix a les neurones de l'estrès oxidatiu. La seva conformació és similar al grup de les tioredoxines [28].
La seqüència d'aminoàcids predita a partir del genoma d'Equus przewalskii és més curta que la proteïna d'Equus caballus. El primer aminoàcid d'Equus przewalskii no és una metionina, i es troba alineat molt més endavant de l'inici real de la proteïna. Les seqüències alineades són, però, pràcticament idèntiques. D'altra banda, la proteïna predita per Genewise és un fragment encara més curt que l'extreta de l'Exonerate i no conté la selenocisteïna, motiu pel qual el problema no podrà ser resolt. Per tant, la seqüència de nucleòtids obtinguda no és fiable, tot i que és segur que es troba a la cadena forward.
Sel M d'Equus caballus conté una selenocisteïna conservada en Equus przewalskii, malgrat que degut al problema esmentat no és possible determinar la seva localització a nivell d'exó. Els programes SECISearch3 i SeBlastian han predit un element SECIS de grau A situat a la mateixa cadena a una distància de 34 nucleòtids de l'últim exó i a 325 de l'aminoàcid en qüestió.
SelN
La selenoproteïna N és una proteïna transmembrana situada a la membrana plasmàtica i al reticle endoplasmàtic de múscul esquelètic, cervell, pulmó i placenta. Controla l'estat redox dels canals d'alliberació de calci com el receptor de rianodina [29][30]. El splicing alternatiu del seu mRNA dóna lloc dues isoformes. La isoforma 1 està formada per la totalitat del transcrit. L'eliminació del tercer exó dóna lloc a la isoforma 2, majoritària.
La proteïna d'Equus caballus és idèntica a la predita a excepció de dos aminoàcids. Aquesta està codificada en el sentit invers per una seqüència de 13422 nucleòtids (425542-412120) formada per 11 exons i 10 introns (transcrit sencer) segons la predicció d'Exonerate. L'element SECIS resultant de SeBlastian és de grau C i es troba a 706 nucleòtids del CDS i a 1090 de la selenocisteïna conservada situada a l'exó 8 del scaffold KK951189.1.
SelO
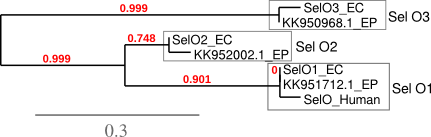
La selenoproteïna O humana és una proteïna de funció desconeguda. No obstant, conté un domini Cys-X-X-Sec que suggereix que es tracta d'una proteïna amb propietats antioxidants relacionades amb les reaccions redox. En cavall, en canvi, trobem 3 selenoproteïnes O, és a dir, una família. [31].
Com podem veure en el següent arbre filogenètic, la Sel O amb ID SPP00000875 es situa al scaffold KK950968.1, mentre que la proteïna amb ID SPP00000874 es troba al scaffold KK952002.1. Finalment, la selenoproteïna O SPP00000873 (que anomenarem Sel O1) present al scaffold KK951712.1 és la proteïna de la família més pròxima a la única Sel O humana. Els resultats ens mostren que aquesta última és la única que conté una selenocisteïna que ha estat conservada per l'evolució, tant en Equus przewalskii com en Homo sapiens.
 |
Sel O1
La seqüència predita tant per Exonerate com per Genewise compta amb 9040 nucleòtids (208374-21897). Està formada per 6 exons i 7 introns situats al scaffold KK951712.1. Així, aquest fragment codifica, en el sentit forward, per una proteïna pràcticament idèntica a la Sel O1 d'Equus caballus, en excepció de l'inici d'aquesta, que tampoc ha pogut ser obtingut a partir de la query humana. La proteïna presenta una selenocisteïna situada en el primer exó del gen, que es troba conservada en Equus przewalskii. Això concorda amb el fet que el programa SECISearch3 trobi un element SECIS a l'extrem 3' de la cadena complementària on es situa el gen, també predit pel programa SeBlastian. Aquest SECIS, amb un grau A, es troba a una distància de 100 nucleòtids de la selenocisteïna i de 94 de l'últim exó.
Sel O2
Aquesta proteïna es troba al scaffold KK952002.1. La seva seqüència és de 8966 nucleòtids (291532-300498) i està formada per 9 exons i 8 introns presents a la cadena forward. La seqüència d'aminoàcids d'Equus caballus és idèntica a la proteïna predita a partir del genoma d'Equus przewalskii, excepte per un sol aminoàcid. El programa SECISearch3 ha trobat un element SECIS que no considerarem fiable degut al seu grau C així com al fet que el programa SeBlastian no hagi predit cap element. No obstant, en tractar-se d'un homòleg en el qual la selenocisteïna ha estat substituïda per una cisteïna al llarg de l'evolució, és possible que encara conservi el seu element SECIS.
Sel O3
La seqüència codificant es troba situada a la regió 368603-368875 del genoma, al scaffold KK950968.1. La predicció de Genewise i de Exonerate indica que el gen té una allargada de 272 nucleòtids, amb un sol exó. Aquest fragment, present a la cadena forward, codifica per una proteïna idèntica a la Sel O amb ID SPP00000875 d'Equus caballus. Aquesta no presenta selenocisteïnes. Això concorda amb el fet que els programes SECISearch3 i SeBlastian no hagin predit elements SECIS ni, conseqüentment, selenoproteïna.
SelP
La selenoproteïna P és una glicoproteïna extrace·ular present al plasma participa en el transport de seleni en diferents òrgans i teixits. Presenta una funció antioxidant [32], i a diferència de la resta, conté un gran nombre de selenocisteïnes a la seva seqüència.
La proteïna és codificada per 5920 nucleòtids (575487- 581407) en el sentit invers al scaffold KK950737.1. Compta amb 4 exons i 3 introns. Les selenocisteïnes, conservades, es troben únicament en el primer i últim exó, sent aquest el més llarg i amb un major nombre de selenocisteïnes. La seqüència predita és pràcticament idèntica a la Sel P d'Equus caballus. Algunes selenocisteïnes presents al final d'aquesta corresponen a aminoàcids que no han pogut ser correctament predits per cap dels dos programes. Genewise, per exemple, talla la proteïna abans d'arribar a l'extrem C-terminal d'aquesta ja que reconeix un dels múltiples codons TGA com a codon stop. Destaquem que el programa T-Coffee alinea una de les selenocisteïnes d'Equus caballus amb una cisteïna. És molt probable però, que aquest alineament no sigui correcte ja que es troba alineat amb un motiu U-C-U. Així doncs, no podem saber amb certesa si la cisteïna correspon en realitat a la cisteïna del motiu o si la selenocisteïna inicial ha mutat convertint-se en cisteïna en Equus przewalskii.
L'element SECIS d'aquest conjunt de selenocisteïnes es troba a l'extrem 3' de la cadena reversa, a una distància de 690 nucleòtids del CDS i de 699 de la última selenocisteïna de la proteïna.
SelR
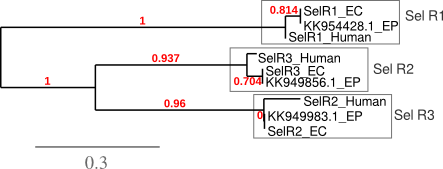
Les selenoproteïnes R formen part de la família de proteïnes metionina sulfòxid reductasa (Msr), que també inclou la proteïna MsrA. Els enzims d'aquesta família són enzims de reparació que redueixen els residus de metionina oxidats. Les proteïnes SelR es poden localitzar tant al nucli com al citosol i s'expressen en una gran varietat de teixits [33]. En aquesta família només Sel R1 conté selenocisteïna a la seva seqüència. Sel R2 i R3 són homòlegs amb cisteïna.
A la figura següent es mostra la filogènia de la família de SelR en les espècies Equus caballus i en les prediccions d'Equus przewalskii:
 |
Sel R1
El millor hit per aquesta proteïna es localitza al scaffold KK954428.1. Segons le prediccions d'Exonerate i Genewise la seqüència que codifica per aquesta proteïna consta de 2240 nucleòtids (46889-49129), es troba en la cadena reversa i conté 3 exon i 2 introns. L'alineament mostra que ambdues proteïnes presenten una gran homologia. En Equus przewalskii es manté la selenocisteïna, que es troba a l'últim exó. Malgrat tot, no s'ha pogut predir element SECIS.
Sel R2
Com hem dit, es tracta d'un homòleg amb cisteïna, per al qual trobem un hit al scaffold KK949983.1 que codifica per una seqüència de 12341 nucleòtids (11838-1196187) a la cadena forward, tal com podem extreure de les prediccions d'Exonerate i Genewise. La seqüència codifica per una proteïna de 4 exons i 3 introns sense selenocisteïna a la seva seqüència, tal com podíem esperar. Tot i això, en l'alineament amb T-Coffee s'observa que manca la regió inicial de la proteïna. L'anàlisi prenent com a query la Sel R2 d'humans resulta també en un alineament en el qual la proteïna predita en Equus przewalskii comença uns quants aminoàcids més enllà del primer aminoàcid de la proteïna humana. Per tant, malgrat que la proteïna predita és un homòleg amb cisteïna que presenta bona homologia amb Sel R2 en ambdues espècies amb les què s'ha comparat, pot ser que la proteïna que hem obtingut no sigui completa.
Pel què fa a la cerca amb SECISearch3 s'han trobat quatre elements SECIS, però amb un grau B que el considerem m´s aviat baix. Malgrat això, no ha estat possible predir cap selenoproteïna mitjançant SeBlastian.
Sel R3
En aquest cas també es tracta d'un homòleg amb cisteïna. A partir de la predicció amb Exonerate obtenim una seqüència de 106778 nucleòtids (3133307-3240085) de 5 exons i 4 introns que codifica per una proteïna que presenta un bon grau d'homologia amb Sel R3 d'Equus caballus. Igual que en el cas falta la regió inicial que tampoc es pot arribar a obtenir fent l'anàlisi partint de Sel R3 d'humà. Així doncs, ens trobem novament en un cas en que obtenim una proteïna amb bona homologia però incompleta.
Igual que en l'anterior, s'obtenen dos elements SECIS però de grau B i no es pot obtenir alineament amb SeBlastian.
SelS
La selenoproteïna S és una proteïna que es pot trobar tant a la membrana plasmàtica com al reticle endoplasmàtic i està implicada en diversos processos patològics com la resposta inflamatòria, resistència a insulina i processos d'estrés oxidatiu [34].
En Equus przewalskii, tant la predicció amb Genewise com amb Exonerate a partir de les dues espècies amb les que s'ha comparat (Equus caballus i humà), s'obté una proteïna incompleta que en l'alineament amb T-Coffee veiem que coincideix amb el fragment central de les proteïnes query. Així doncs, a la selenoproteïna predita li manca l'extrem inicial que inclou la metionina i el segment final on en les altres espècies es troba la selenocisteïna. Per aquesta proteïna no s'ha pogut predir ni SECIS ni alineament amb SeBlastian. Per tant, no disposem dels resultats necessaris per confirmar que aquest fragment obtingut pertanyi a la selenoproteïna S.
SelT
La selenoproteïna T pertany a la superfamília de les tioredoxines, i, juntament amb Sel H i Sel W, presenta un domini tioredoxina i un motiu Cys-X-X-Sec conservat [Dikiy et al, 2008]. Es troba a l'aparell de Golgi i al reticle endoplasmàtic i la seva funció està relacionada amb la regulació dels estats redox. A més, juga un paper central en els processos d'adhesió cel·lular [35]. La seqüència codificant es troba a la regió 563457-568486 del genoma. El gen, situat a la cadena forward del scaffold KK949882.1. Té, segons Exonerate i Genewise, una llargada de 5029 nucleòtids, amb 4 exons i 3 introns. El fragment predit codifica per una proteïna idèntica a la SelT d'Equus caballus. Aquesta presenta una selenocisteïna situada en el primer exó del gen que es troba conservada en Equus przewalskii.
Això concorda amb el fet que el programa SECISearch3 trobi un element SECIS a l'extrem 3' de la cadena complementària, també predit pel programa SeBlastian. Segons aquest, el SECIS, de grau A, es troba situat a una distància de 998 nucleòtids de la selenocisteïna i de 560 de l'últim exó.
SelU
Actualment, encara es desconeix la funció de la família de selenoproteïnes U tot i que també forma part de la superfamília de les tioredoxines. En humans, aquesta família està formada per 3 proteïnes, mentre que només se'n troben dues en cavall. La Sel U1 s'expressa al teixit ossi, al cervell, al fetge i als ronyons [26] [36].
Sel U1
La seqüència predita tant per Exonerate com per Genewise compta amb 9040 nucleòtids (73547-82587). Està formada per 5 exons i 4 introns situats al scaffold KK954222.1. Així, aquest fragment codifica, en el sentit forward, per una proteïna pràcticament idèntica a la SelU1 d'Equus caballus. Pel que fa a l'anàlisi del SeBlastian veiem que no ha estat possible predir cap selenoproteïna malgrat que la proteïna presenta un element SECIS de grau B segons el programa SECISearch3. Els nostres resultats, doncs, demostren que es tracta d'un homòleg amb cisteïna; malgrat que la proteïna hagi perdut la selenocisteïna, substituïda per una cisteïna, aquesta encara conserva el seu element SECIS.
Sel U2
La proteïna Sel U2 es troba al scaffold KK952962.1. La seva seqüència és de 7608 nucleòtids (203909-211517) i està formada per 6 exons i 5 introns presents a la cadena reversa. La seqüència d'aminoàcids d'Equus caballus és pràcticament idèntica a la proteïna predita a partir del genoma d'Equus przewalskii, a excepció d'un petit nombre d'aminoàcids. Com la Sel U1, es tracta d'un homòleg amb cisteïna que conserva l'element SECIS. Aquest ha estat predit pel programa SECISearch3 amb un grau B. No s'han obtingut resultats amb el programa SeBlastian degut a l'absència de selenocisteïna.
SelW
La família de Sel W en humans està formada per únicament dues selenoproteïnes involucrades en processos d'oxidació-reducció com la resta de membres de la superfamília de les tioredoxines (Sel H i Sel T) . La Sel W1 s'expressa principalment al múscul esquelètic i al cor, mentre que la Sel W2 és més ubiqua [37].
Pel que fa a la família SelW en Equus caballus observant la filogènia, veiem que una de les proteïnes (ID SPP00000884) en la filogènia es relaciona estretament amb SelW1 humana. L'altra proteïna que trobem en cavall (ID SPP0000085) malgrat no relacionar-se directament amb cap de les proteïnes humanes és més propera a SelW1 que a SelW2.
 |
La selenoproteïna amb ID SPP0000084 no ha pogut ser predita a partir de la query d'Equus caballus ja que l'E-value (2e-08) és superior a la restricció establerta en el script. Amb la Sel W1 humana, la predicció del programa TBLASTN també és exclosa degut a aquesta restricció (E-value de 6e-08).
La seqüència codificant predita a partir de Sel W2 humana, que és un homòleg amb cisteïna, està formada per 4 exons i es troba a la cadena reversa del scaffold KK950318. Té una llargada de 993 nucleòtids (84190-85183). L'absència de selenocisteïna concorda amb el fet que no es trobin elements SECIS. En comparar amb la selenoproteïna SPP0000085 d'Equus caballus obtenim dos hits, un localitzat al scaffold KK951606 i un al scaffold KK954477. La seqüència predita al scaffold KK951606 té una llargada total de 3108 nucleòtids (309544-312652) a la cadena reversa. En aquest cas, mentre que en cavall hi ha presència de selenocisteïna, en Equus przewalskii no en trobem. En canvi, el programa SECISearch3 és capaç de trobar un element SECIS de grau A amb la proteïna predita a partir d'Equus caballus. Aquest es situa a la cadena reversa, a 935 nucleòtids del codó UGA present al segon exó dels quatre que codifiquen per la proteïna. Pel que fa a la seqüència del scaffold KK954477, degut al seu baix E-value i poca longitud podem pensar que malgrat que s'alineï amb la proteïna SPP0000085 de cavall podria ser fruit de l'atzar, més que tractar-se d'un possible pseudogen.
TR
La família de les tioredoxin reductases està involucrada en la formació de ponts disulfur mitjançant la qual realitzen la seva funció antioxidant. Utilitzant NADPH com a catalitzador, les proteïnes d'aquesta redueixen la tioredoxina, captant els seus electrons per tal de cedir-los a les proteïnes i convertir els seus disulfurs oxidats en la seva forma reduïda (SH2)[38][39].
 |
Sel TR1
La tioredoxina reductasa 1 és una proteïna citosòlica que redueix la forma oxidada de la tioredoxina citosòlica. Presenta com a mínim 6 isoformes que difereixen en la seva seqüència N-terminal [38][39].
La proteïna predita a partir de la TR1 d'Equus caballus és molt semblant a aquesta a excepció d'un fragment present únicament en Equus przewalskii. Pot tractar-se d'una inserció o bé d'un error dels programes en interpretar la seqüència codificant. D'altra banda, cap de les dues proteïnes de cavall comença per metionina. Destaca també el fet que la selenocisteïna present a la TR1 del cavall domèstic no pot ser trobada en el cavall salvatge ja que tant Genewise com Exonerate tallen la seqüència predita abans del final de la proteïna. Per aquest motiu no és possible saber si la Sec de la tioredoxina reductasa 1 està conservada en Equus przewalskii. El fet que el programa SECISearch3 no trobi cap element SECIS podria indicar que la TR1 d'aquest últim no conté selenocisteïna. Tot i així és possible que aquesta manca de resultats es degui a una mala anotació ja sigui de la query utilitzada o del genoma d'Equus przewalskii.
En alinear la proteïna amb la humana, observem un alineament relativament bo, motiu pel qual podem pensar que les seqüències d'ambdós cavalls són incompletes i que els manca el fragment inicial. A més, tant Exonerate com Genewise segueixen donant una proteïna truncada en la qual falta el fragment final de l'últim exó on hauria de trobar-se la selenocisteïna.
La seqüència codificant del fragment predit de la tioredoxina reductasa 1 compta amb 15 exons i 14 introns compresos en 77569 nucleòtids (531244-608813) a la cadena reversa del scaffold KK950772.1.
Sel TR2
La tioredoxina reductasa 2 també s'expressa al citosol, especialment de les espermàtides, i presenta un domini glutaredoxina, que catalitza una gran varietat de reaccions específiques pels sistemes de glutaredoxines [38]
En agafar la proteïna d'Equus caballus com a referència, la predicció d'Exonerate resultant no presenta un bon alineament amb la primera. El fragment inicial és pràcticament idèntic malgrat que cap comenci per metionina. La resta de la proteïna és força diferent: alguns fragments presents a Equus caballus no es troben a Equus przewalskii i viceversa. La predicció de Genewise tampoc pot ser utilitzada ja que el programa reconeix el codó TGA com a codó stop.
En utilitzar la proteïna humana, Genewise torna a tallar a proteïna en la posició prèvia a la selenocisteïna. Per aquest motiu, hem seleccionat la predicció d'Exonerate. Cal destacar però que aquesta no comença per metionina i que no presenta el fragment inicial de la proteïna. Tot i així, les proteïnes són força similars. La seqüència predita està codificada per 38392 nucleòtids (99330-137722) en sentit invers, compta amb 14 exons i 13 introns i es troba al scaffold KK953564.1. La selenocisteïna està conservada i es troba a l'últim exó de la proteïna d'ambdós cavalls. Amb programa SECISearch3 s'obté un element SECIS de grau B present a l'extrem 3'UTR de la cadena complementària. El programa SeBlastian, en canvi, no és capaç de predir cap selenoproteïna.
Sel TR3
La tioredoxina reductasa 3 es troba als mitocondris i redueix la forma oxidada de la tioredoxina i de la glutaredoxina 2 [39].
La proteïna predita per Exonerate és idèntica a la TR3 d'Equus caballus a excepció d'un petit nombre d'aminoàcids absents en aquesta última. Podria tractar-se d'una inserció present únicament a la proteïna d'Equus przewalskii tot i que una altra possibilitat és que es tracti d'un error dels programes emprats. Destaca el fet que malgrat que el seu alineament sigui perfecte, cap de les dues comença per metionina. La seqüència codificant de la proteïna es troba a la cadena forward del scaffold KK953348.1. Formada per 9 exons i 8 introns, la seva llargada és de 42531 nucleòtids (120576-163107). La selenocisteïna està conservada i, de la mateixa manera que les altres proteïnes de la família, es troba a l'últim exó. El seu element SECIS es troba la mateixa cadena, a una distància de 176 nucleòtids d'aquesta i, degut a que es tracta del penúltim aminoàcid de la proteïna, a una distància de 173 nucleòtids de l'últim exó.
Maquinària
eEFsec
El factor d'elongació eucariota (eEFsec, també conegut com EFSec) està implicat tant en la biosíntesi de selenocisteïnes com en la seva incorporació a la proteïna i sembla que, a més, interacciona amb la resta de components de la maquinària de síntesi d'aquestes [40]. Aquesta proteïna, a diferència de les anteriors, no presenta selenocisteïna ni es tracta d'un homòleg amb cisteïna ja que únicament forma part de la seva maquinària de síntesi.
El scaffold seleccionat és el KK951551.1. La seqüència predita es troba situada a la regió 188854-359627 del genoma d'Equus przewalskii. La predicció realitzada per Exonerate indica que el gen té una llargada de 170773 nucleòtids, amb 6 exons i 5 introns. Aquest fragment codifica en el sentit forward. En analitzar l'alineament dut a terme pel programa T-Coffee, veiem que és una proteïna idèntica a la d'Equus caballus, probablement degut a la gran importància que té la seva conservació. La maquinària codificada pel gen eEFSec està molt conservada entre espècies del mateix gènere donat a que és essencial per la síntesi de selenoproteïnes. Destaca el fet que s'han trobat tres elements SECIS a partir del programa SECISearch3, que no han estat considerats ja que presenten un grau molt baix.
SBP2
La SECIS binding protein 2 és un component fonamental en la síntesi de selenoproteïnes i forma part d'un complex multiproteic juntament amb eEFSec, SECp43 i SPS1, entre d'altres. S'uneix a l'element SECIS, a l'extrem 3'UTR del mRNA de la selenoproteïna i interactua amb el factor d'elongació de la traducció (eEFsec) i la subunitat 60S del ribosoma per tal d'incorporar la selenocisteïna a la selenoproteïna [41].Podem observar que aquesta proteïna tampoc presenta selenocisteïna i que per tant forma part de la maquinària de síntesi de selenoproteïnes.
Cal destacar que mentre el gènere Equus presenta dues SBP2, només podem trobar-ne una en Homo sapiens. L'arbre filogenètic que trobem a continuació ens ha permès determinar quina de les dues proteïnes de cavall presenta més similituds amb la humana. Com podem veure, la més pròxima a la SBP2 d'Homo sapiens és la proteïna amb ID SPP00000863, que anomenarem SBP2 1.
 |
SBP2 1
La seqüència predita tant per Exonerate com per Genewise compta amb 98500 nucleòtids (356332-454832). Està formada per 18 exons i 17 introns situats al scaffold KK950576.1. Així, aquest fragment codifica, en el sentit invers, per una proteïna idèntica a la SBP2 1 d'Equus caballus, probablement degut a la gran conservació de la maquinària. La manca de resultats per part del programa SECISearch3 concorda amb el fet que es tracti d'una proteïna implicada en la síntesi de selenoproteïnes però que no forma part d'aquestes.
SBP2 2
La proteïna SBP2 None1 es troba al scaffold KK950489.1. La seva seqüència és de 36019 nucleòtids (485273-521292) i està formada per 15 exons i 14 introns presents a la cadena forward. Les prediccions d'Exonerate i Genewise coincideixen. La seqüència d'aminoàcids d'Equus caballus és també idèntica a la proteïna predita a partir del genoma d'Equus przewalskii, probablement degut a la gran conservació d'aquesta a conseqüència de la importància de la seva funció. Com la major part dels membres de la maquinària, no presenta elements SECIS.
SECp43
El tRNA Sec 1 associat a proteïna 1 és tRNA nuclear amb un paper en la biosíntesi i regulació de selenoproteïnes mitjançant la interacció Sec-tRNA, i juntament amb la SecS, participa en l'últim pas de la síntesi de la selenoproteïna[3][42].
Com en el cas anterior, el gènere Equus presenta dues SECp43. En humans, en canvi, només trobem una proteïna. En el següent arbre filogenètic veiem que la SECp43 més similar a la d'Homo sapiens és la proteïna amb ID SPP00000892, que anomenarem SECp43 1.
 |
SECp43 1
El scaffold seleccionat és el KK951401.1. La seqüència codificant es troba situada a la regió 245855-247294 del genoma. La predicció de Genewise indica que el gen té una llargada de 1439 nucleòtids, amb 3 exons i 2 introns. Aquest fragment codifica en el sentit invers. Es tracta d'una proteïna pràcticament idèntica a la d'Equus caballus, que només es diferencien per dos aminoàcids. En no comptar amb cap selenocisteïna, aquesta no presenta cap element SECIS.
SECp43 2
El tRNA SECp43 amb ID SPP00000892 es troba al scaffold KK950396.1. La seva seqüència és de 14857 nucleòtids (550977-565834) i està formada per 7 exons i 6 introns presents a la cadena reversa. La seqüència d'aminoàcids d'Equus caballus és també idèntica a la proteïna predita a partir del genoma d'Equus przewalskii. Com la resta d'elements de la maquinària vistos fins al moment, és molt probable que aquesta similitud es degui a la importància de la seva conservació. Com el tRNA amb ID SPP00000891, no es tracta d'una selenocisteïna ni d'un homòleg amb cisteïna, motiu pel qual no s'ha pogut trobar cap element SECIS.
SecS
Sec S és una selenocisteïna sintasa que transforma el O-phospho-l-seryl-tRNA[Ser]Sec en selenocisteyl-tRNA[Ser]Sec utilitzant el selenofosfat com a compost donador de seleni obtingut gràcies a SBP2 en un pas previ de la síntesi. d'aquesta manera, converteix la serina fosforilada del Ser-tRNASec en una selenocisteïna [3][43].
La seqüència predita tant per Exonerate com per Genewise compta amb 32679 nucleòtids (438703-471382). Està formada per 11 exons i 10 introns situats al scaffold KK951080.1. Així, aquest fragment codifica, en el sentit invers, per un enzim pràcticament idèntic a la Sec S d'Equus caballus, a excepció d'uns quants aminoàcids del fragment inicial de la proteïna. De nou, no conté selenocisteïna, ni elements SECIS.
pstk
La fosfoseril-tRNA quinasa és un enzim encarregat de fosforilar la serina del Ser-tRNASec, produint O-fosfoseril-tRNA(Sec) per tal d'incorporar posteriorment el seleni i convertir-la en una selenocisteïna [3][44].
La seqüència predita tant per Exonerate com per Genewise compta amb 6019 nucleòtids (27464-33483). Està formada per 5 exons i 4 introns situats al scaffold KK951858.1. Així, aquest fragment codifica, en el sentit invers, per una proteïna pràcticament idèntica a la SelU1 d'Equus caballus. El programa SECISearch3 no ha trobat cap SECI, motiu pel qual no ha estat possible predir cap selenoproteïna mitjançant SeBlastian.
SPS
La selenofosfat sintetasa és una proteïna citosòlica [3]. La seva funció és generar compostos donadors de selenofosfats, necessaris per a la biosíntesi de selenocisteïnes [6].
SPS 1
El scaffold seleccionat és el KK953938.1. La seqüència que codifica per la SPS1 d'Equus przewalskii té una llargada de 24210 nucleòtids (65958-90168), està formada per 8 exons, i es troba a la cadena reversa del scaffold KK953938.1. Els resultats del programa T-Coffee indiquen que és una proteïna idèntica a la d'Equus caballus. En aquest cas, és possible que es tracti d'un homòleg amb cisteïna que encara conserva el seu element SECIS, que ha pogut ser trobat pel programa SECISearch3. En canvi, el programa SeBlastian no ha pogut dur a terme cap predicció.
SPS 2
Aquesta proteïna no ha pogut ser analitzada degut a un error en l'execució del TBLASTN. No ha estat possible obtenir resultats amb cap de les querys utilitzades (Equus caballus i Homo sapiens). Tot i així, el fet que SPS1 presenti un element SECIS concorda amb la selenocisteïna observada en la proteïna de cavall domèstic ja que es tracta de dues proteïnes de la mateixa família.