Discussió
L'objectiu del nostre treball és identificar les selenoproteïnes i proteïnes de la maquinària en el genoma de Pelodiscus sinensis. El protocol que hem seguit el podeu trobar aquí.Selenoproteïnes i filogènia dels vertebrats
P. sinensis és un rèptil de l'ordre Testudines i, per tant, els grups més propers filogenèticament són els Archosauria (ocells, cocodrils i caimans) seguits de prop per l'ordre Lepidosauria (llangardaixos).
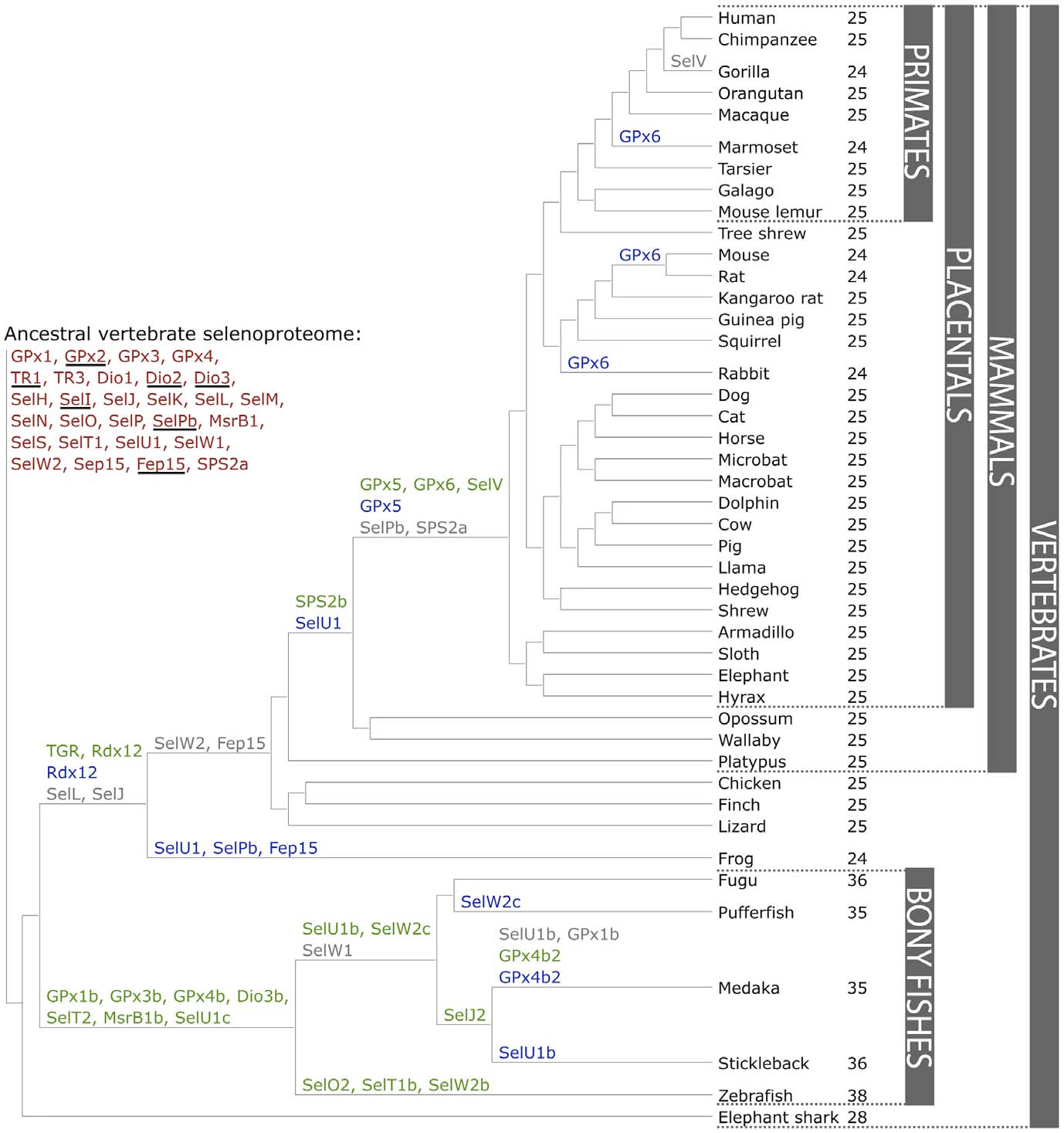
Segons un estudi dels selenoproteomes dels vertebrats, d'on prové el següent arbre filogenètic, el genoma de P. sinensis hauria de tenir aproximadament les 25 selenoproteïnes que tenen el gall (chicken), el pinsà (finch) i el llangardaix (lizard). Aquestes selenoproteïnes són les següents: GPx1, GPx2, GPx3, GPx4, TR1, TR3, Dio1, Dio2, Dio3, SelH, SelI, SelK, SelM, SelN, SelO, SelP, SelPb, MsrB1, SelS, SelT1, SelU1, SelW1, Sep15, SPS2a i TGR.[5],[6]
Comentari espècies
Les selenoproteïnes de referència o queries utilitzades per anotar els gens d'interès del nostre genoma provenen d'espècies diferents.[7] En els casos en què el Selenoprofiles ha proporcionat un bon alineament entre seqüències i el nostre genoma, s'han observat valors d'score molt elevats, així doncs s'ha mantingut l'espècie de la query utilitzada per fer la revisió manual. Aquesta situació s'ha donat en SelI, Dio 1, 2 i 3, Gpx 2 i 3, SelK, Sep15, SelN, SelR 1 i 3, SelW.2, TR1, 2 i 3, MrsA i SelS.
En tres selenoproteïnes, SelP, SelO i SelW.1, el Selenoprofiles alinea la seqüència proteica d'aquestes amb una Blast Query Master, és a dir, una predicció fruit de la barreja de diferents selenoproteïnes conegudes amb l'objectiu d'aconseguir un alineament òptim amb la seqüència d'interès. En aquest cas, la query utilitzada per l'alineament s'ha trobat manualment, triant l'espècie que presenti millor score.
En les situacions en què les queries que utilitza el Selenoprofiles no proporcionen bons alineaments, es tria manualment l'espècie que convingui més en cada cas, com ha passat amb Gpx1 i 4, SelT i SelU. Si a la base de dades només hi ha la selenoproteïna descrita d'una espècie o de diverses espècies igual de distants filogenèticament a la tortuga, s'ha realitzat l'alineament de manera manual obtenint uns valors de score subòptims. S'ha realitzat aquest procediment en SelP, SelPb, MrsB, SelH, TGR, Rdx12.
Per altra banda, el Selenoprofiles no cerca les proteïnes implicades en la síntesi de selenoproteïnes. Davant d'aquesta situació, les hem cercat manualment triant les espècies que proporcionin millors alineaments. Aquestes són SecS, SBP2, Efsec, Pstk1 i SEC43p. Aquesta última, però, presenta un pitjor alineament.
Selenoproteïnes no trobades
Hem trobat a la literatura que per filogènia, les selenoproteïnes Fep15, SelJ i SelL no s'haurien de trobar en el genoma de Pelodiscus sinensis . Per comprovar-ho, les hem cercat manualment i hem confirmat que no hi són, ja sigui perquè el BLAST no ha trobat cap hit (SelL i SelJ) o perquè l'E-value és massa elevat (2,2 en Fep15).
Sorpreses
Comparant els nostres resultats amb els coneixements previs sobre el selenoproteoma dels vertebrats ens hem trobat amb unes quantes sorpreses. No hem trobat SelM i, en canvi, hem trobat TR2, SelW2, SelRB1, SelRB3 i SelU1 (aquestes dues últimes amb cisteïna).
Comentari individual per cada proteïna
Selenoproteïnes
SelT
Inicialment, el Selenoprofiles va utilitzar la selenoproteïna de referència de l'espècie Ostreococcus taurus per realitzar l'alineament de seqüències. El hit resultant del Selenoprofiles es situava en un scaffold diferent (JH211924,1) al que troba el BLAST (JH206810.1). L'E-value per tots dos hits és bastant dolent, de l'ordre de 0,1 i l'alineament obtingut manualment no és gens bo. Per aquests motius, es va buscar la SelT en una espècie mès propera a la nostra, com és Gallus gallus.
Al analitzar aquesta selenoproteïna manualment s'obtè un score alt (93) i una selenocisteïna alineada. Com que l'alineament no comença per metionina, hem intentat allargar la seqüència del nostre genoma però al empitjorar el Score hem deduït que el fragment que s'agafava no era exònic. Així doncs, ens hem quedat amb l'alineament obtingut amb el t_coffee sense modificar la llargada de la seqüència.
DiO1
El valor del coverage que dóna el Selenoprofiles en aquesta selenoproteïna és molt elevat, 1. A més a més, l'alineament també és molt bo i s'alineen les selenocisteïna d'ambdues seqüències. Igualment, a mode de comprovació, hem realitzat l'estudi manual en el qual apareix un score màxim i l'element SECIS és strict. Això ens demostra l'alta homologia entre la DiO1 de referència i la nostra seqüència.
DiO2
En el cas de la Dio2, l'anotació generada pel Selenoprofiles semblava òptima però utilitzava com a query una versió antiga de la Dio2 de Gallus gallus en la qual es considerava com a codó STOP un TGA que en la versió actualitzada es mostra que codifica per a una selenocisteïna. Utilitzant aquesta nova versió es va aconseguir un alineament en el qual les dues selenocisteïnes de la proteïna estaven alineades i la metionina inicial també. Tot i que el coverage disminuia respecte a l'anotació generada pel selenoprofiles (d'1 a 0,96) es va considerar que l'anotació generada manualment era més acurada.
DiO3
La selenoproteïna Dio3 es troba al genoma de Pelodiscus sinensis. Aquesta proteïna està formada per un sol exó i té una alta homologia en l'alineament. En un inici, el programa Selenoprofiles ja va alinear la selenoproteïna Dio3 coneguda de Gallus gallus amb el genoma de la nostra tortuga, donant uns resultats molt bons. Per comprovar els dades obtingudes automàticament hem realitzat l'alineament de manera manual i els resultats concorden amb el que ja teníem: l'score és molt alt (99), el coverage és total (1) i les selenocisteïnes d'ambdues seqüències s'alineen perfectament.
MsrA
El resultat d'aquesta selenoproteïna en el Selenoprofiles no és concloent i, per aquest motiu, hem repetit la cerca manualment. Amb els resultats de l'alineament realitzat podem establir que la MsrA es troba en el genoma de Pelodiscus sinensis, tot i que la homologia de l'alineament està per sota del 50% (score de 37). La baixa homologia pot ser donada per la manca d'informació del scaffold del genoma de de la tortuga en aquesta regió. El coverage d'aquesta proteïna és bastant alt (0,87), fet que dóna consistència a l'alineament. Finalment, pel que fa als elements SECIS, MsrA té un de qualitat intermèdia (Default).
MsrB1
La metionina sulfòxid reductasa B1 no es va trobar amb el Selenoprofiles. Per tant, es va cercar manualment. Es va provar d'alinear el genoma de la nostra tortuga amb seqüències proteiques conegudes de dues espècies diferents: Homo sapiens i Bos taurus. El BLAST va trobar el hit al mateix scaffold per ambdues espécies peró finalment vam escollir alinear la de Bos taurus perquè presentava un millor E-value, score i coverage que Homo sapiens. Atenent als resultats obtinguts, podem considerar que MsrB1 és una selenoproteïna de Pelodiscus sinensis .
SelH
La selenoproteïna H no es va trobar mitjançant el Selenoprofiles. No obstant això, com que al mirar a la literatura vam veure que per filogènia sí hauria d'estar, es va buscar manualment. Així doncs, de les tres espècies candidates on està anotada la SelH, Homo sapiens, Sus scrofa, i Bos taurus, vam escollir aquesta última perquè era la única on s'alineava una selenocisteïna. Cal dir que hem observat que la nostra seqüència contenia altres selenocisteïnes que no estan alineades.
Pel que fa als valors del coverage i el score predits són molt baixos, 0,16 i 15, respectivament. Per altra banda, no s'ha trobat cap element SECIS. Davant d'aquesta situació, no podem assegurar que SelH estigui en el genoma de la nostra tortuga a diferència del que afirma la literatura.
SelK
La selenoproteïna K s'obté del Selenoprofiles agafant com a model la seqüència de Gallus gallus. Aquesta mostra un coverage excepcional (1) i l'alineament és molt bo i comença per metionina. Al fer la revisió manual es confirmen els resultats obtinguts. A més a més, la qualitat de l'element SECIS és strict. Així doncs, podríem assegurar que SelK es troba en la seqüència del nostra genoma.
Selw1
La selenoproteïna Selw1 no ha estat trobada pel Selenoprofiles però sí de manera manual. En un inici es van fer alineaments amb seqüències conegudes de Selw1 de diverses espècies (Danio rerio, Homo sapiens, Xenopus laevis), seleccionant finalment la query procedent de Gallus gallus, ja que és el hit que donava millors resultats al tBLASTn.
Aquest alineament consta d'una alta homologia, presentant uns valors d'score (89) i coverage (0,91) elevats. A més a més, les dues selenocisteïnes de les seqüències comparades s'alineen perfectament i la qualitat de l'element SECIS és excel·lent (strict).
SelW2
Aquesta selenoproteïna s'ha obtingut del Selenoprofiles i és una duplicació de SelW1, encara que els hits obtinguts per cada una apareixen en scaffolds diferents i no continus. Per tal de comprovar que realment s'alinea amb la nostra seqüència hem fet l'estudi manual, obtenint uns valors de coverage i score mediocres. La selenocisteïna sí està alineada i s'ha trobat un element SECIS de qualitat intermitja o default. Tenint en compte tots els resultats, podem dir que ens trobem davant d'una Selenoproteïna que hipotèticament no hauríem d'haver trobat però que sí presenta certa homologia.SelI
El Selenoprofiles va utilitzar l'etanolamina fosfotransferasa 1 de Gallus gallus com a query per realitzar l'alineament entre seqüències, obtenint resultats satifactoris. L'alineament obtingut és quasi perfecte, ja que els valors de coverage i score són de 0,97 i 97 respectivament. No s'ha pogut trobar la metionina a l'inici de la seqüència ni amb el Selenoprofiles ni manualment, però les selenocisteïnes d'ambdues selenoproteïnes es troben alineades.SelS
La selenoproteïna S ha estat predita tant pel Selenoprofiles com manualment a partir de la seqüència de Taeniopygia guttata. Com que el valor del coverage no és gaire elevat (0.63), no s'ha obtingut un score gaire bo (58). Endemés, en aquest cas no hi ha hagut alineament entre les selenocisteïnes, ja que la seqüència de la query acaba just un aminoàcid abans que aparegui la selenocisteïna a la selenoproteïna de la nostra tortuga.
Es van revisar totes les selenoproteïnes S enregistrades a la base de dades NCBI per veure si acabaven de la mateixa manera i poder descriure aquest fenomen com a aïllat, però es va observar que el patró era igual en tots els casos. No obstant això, com que a la seqüència de la proteïna de Pelodiscus sinensis s'observa un aminoàcid codificat per un codó stop (U) que no implica el final de la traducció, hem considerat la SelS1 com a selenoproteïna.
SelR1
En aquest cas, la selenocisteïna de la nostra proteïna diana s'alinea amb una cisteïna de la query de ratolí utilitzada, ja que aquesta última presenta una mutació en l'aminoàcid 95 (sec95cys).
SelO
En aquest cas, el Selenoprofiles mostrava l'alineament de la seqüéncia de la proteïna de la nostra tortuga amb una blast query master, tal i com s'explica a materials i mètodes. Per tant, es van establir alineaments manuals amb selenoproteïnes de diferents espècies, escollint la de Gallus gallus perquè proporcionava els millors resultats. Cal remarcar que el Selenoprofiles predia una proteïna amb cisteïna i manualment s'ha obtingut una selenoproteïna, coincidint amb el que es descriu a la literatura. El coverage és de 0,76 i l'score és de 75. Pel que fa referència a l'element SECIS, s'ha trobat un de baixa qualitat (loosest) que es troba molta distància: 20266 posicions.
TR1
La thioredoxin reductasa 1 predita pel Selenoprofiles s'obté a partir de l'alineament amb una query modificada d' Homo sapiens. Per ratificar els resultats obtinguts automàticament, vam fer l'estudi manualment a partir de la TR1 de Gallus gallus perquè no hi ha cap base de dades que contingui la proteïna modificada que utilitza el programa. El valor d'score (65) indica una qualitat de l'alinemanent moderada entre les seqüències. De fet, es pot observar com aquest és nul a l'inici i molt bo a partir de la meitat de la proteïna. Això podria ser degut a una manca d'informació del scaffold del genoma de Pelodiscus sinensis.TR2
Si ens guiem per les selenoproteïnes descrites i distribuïdes a l'arbre filogenètic, la thioredoxin reductasa 2 no s'hauria de trobar al genoma de Pelodiscus sinensis, només s'haurien de trobar les thioredoxin reductases 1 i 3. No obstant això, a més de trobar aquestes dues últimes, hem trobat la TR2 tant amb el Selenoprofiles com manualment. El gen que la codifica conté 16 exons, 1 més que les altres reductases. Per considerar aquesta selenoproteïna com a una més dins el genoma de la tortuga, ens recolzem en el bon alineament obtingut (score de 98), en la qualitat del SECIS trobat (strict) i en el fet que la proteïna es trobi en un scaffold diferent a les altres dues, descartant així un mapeig sobre la mateixa regió genòmica.TR3
La TR3 presenta un bon alineament amb la proteïna de referència, ja que els valors de coverage i score són de 0,93 i 93 respectivament. Pel que fa a la qualitat de l'alineament i del SECIS, s'han obtingut els mateixos resultats a través del Selenoprofiles i de manera manual. Aprofitant la qualitat de l'alineament s'ha provat de trobar la metionina de la seqüència de manera manual, però els valors empitjoraven.TGR
El Selenoprofiles no va trobar aquesta proteïna però, d'acord amb el que descriuen els estudis filogenètics, la vam trobar manualment alineant el genoma de la tortuga amb una query de Fasciola hepatica. L'alineament de les seqüències és bo encara que la distància filogenètica entre les dues espècies comparades sigui alta, ja que el Score resultant és de 91. Endemés, les selenocisteïnes d'ambdues proteïnes estan alineades.
SelM
Si ens guiem per la distribució de selenoproteïnes conegudes i descrites a l'arbre filogenètic, hauríem de trobar la selenoproteïna M al genoma de Pelodiscus sinensis. No obstant això, no s'ha trobat cap hit significatiu dins del genoma al alinear amb queries de tres espècies diferents: Homo sapiens, Mus musculus i Sus scrofa (tots els valors de les e-values obtingudes eren superiors a 4.0).
SelPb
El Selenoprofiles prediu la seqüència de SelPb a partir de l'alineament amb una query modificada de Takifugu. A l'hora de fer l'alineament manual, en canvi, s'utilitza una query de Danio rerio perquè és el que presenta una homologia més alta i també és un peix. Tot i que hi ha selenocisteïnes alineades entre les seqüències, la selenoproteïna de la nostra tortuga en presenta una més.
SelP
En aquest cas el Selenoprofiles intentava l'anotació utilitzant una blast query master. Com que el coverage era força baix (0,56) i no podíem replicar els resultats manualment (no podíem obtenir la seqüència completa de la blast query master) es va intentar una anotació manual utilitzant la SelP de Gallus gallus. Es va aconseguir un alineament força bo (score=88; coverage=0,99; metionina i múltiples selenocisteïnes alineades) en el mateix scaffold que indicava el Selenoprofiles. Ens vam quedar amb l'alineament manual perquè el coverage i l'score eren més grans. Ambdós alineaments són conseqüents i confirmen la presència de SelP en el selenoproteoma de Pelodiscus sinensis .
SelP15
El Selenoprofiles va trobar aquesta proteïna amb un alineament força bo (score=84 i la selenocisteïna estava alineada), però no va trobar la metionina inicial. Tampoc vam aconseguir trobar la metionina manualment. Tenint en compte les evidències recollides manualment i amb el Selenoprofiles considerem que aquesta selenoproteïna es troba codificada en el genoma de Pelodiscus sinensis però falta per trobar el fragment inicial de la proteïna (coverage=0,85), que inclou la metionina d'inici. Segurament no s'ha aconseguit trobar l'inici de la proteïna perquè està codificat en un altre exó que no estem detectant. Potser aquest exó coincideix amb un tros de la seqüència genòmica que no està ben descrit, és a dir, reomplert amb nucleòtids indeterminats (Ns) o queda excepcionalment lluny de la resta d'exons.
SelN
El Selenoprofiles va trobar aquesta proteïna amb un alineament molt bo (score=92 i la selenocisteïna estava alineada). Tot i així, no es trobava alineada la proteïna completa (coverage=0,93) i no va trobar la metionina inicial. Utilitzant l'exonerate amb l'opció exhaustive vam aconseguir trobar un exó més que el Selenoprofiles, però no vam poder trobar la metionina inicial. O bé falta anotar un fragment del primer exó o falta trobar l'exó inicial que hauria de contenir la metioinina. Malgrat tot, considerem que s'han recollit prou evidències per afirmar que Pelodiscus sinensis té aquesta selenoproteïna.
GPx1
El Selenoprofiles no va trobar aquesta proteïna al genoma de Pelodiscus sinensis i, per tant, es va haver d'anotar manualment per complet. Com a queries es van utilitzar la GPx1 de Gallus gallus (anotada parcialment) i la d'Homo sapiens (completa), però amb cap de les dues queries no es van aconseguir alinear ni la selenocisteïna ni la metionina inicial. Els resultats presentats a la taula corresponen als obtinguts utilitzant la GPx1 de Gallus gallus, ja que, com era d'esperar, tenia una major homologia de seqüència. No aconseguim alinear la selenocisteïna i la metionina perquè es troben a l'inici de la proteïna (observat mirant l'anotació en Homo sapiens , que està completa) i tant en l'anotació a Gallus gallus com la que hem realitzat en Pelodiscus sinensis no es troba l'inici de la proteïna. Això vol dir que, un altre cop, manca trobar un tros del primer exó o, més probablement, un exó sencer a l'inici de la proteïna.
GPx2
Es tracta d'una proteïna altament conservada entre els llinatges de Mus musculus i Pelodiscus sinensis , ja que l'alineament té un coverage d'1 i un score de 100. El selenoprofiles generava una anotació subòptima (coverage=0,99) i utilitzant l'exonerate amb l'opció exhaustive es va aconseguir millorar. Manualment (opció loosest) vam trobar un element SECIS, però força lluny (10000nt) del final de la regió codificant. Possiblement l'element SECIS real no és aquest, sinó un altre més pròxim i que no vam trobar.
GPx3
El selenoprofiles troba aquesta proteïna utilitzant com a query la GPx6 de Sus scrofa (porc senglar, un mamífer placentari). La GPx6 es va originar per duplicació del gen de la GPx3 en la branca de mamífers placentaris [4], per aquesta raó té una elevada homologia amb la GPx3. Es va comprovar l'anotació manualment utilitzant la GPx3 d'Homo sapiens . Manualment es va poder trobar la metionina inicial i millorar l'score fins a 97. Tot i que té selenocisteïna i aquesta es troba alineada amb la query (GPx6 d'Sus scrofa ) no es va poder identificar cap element SECIS a la regió 3' propera ni amb el selenoprofiles ni manualment amb el SECISearch. Tot i així, creiem que les evidències són suficients per a poder confirmar la presència de la GPx3 en el selenoproteoma de Pelodiscus sinensis . Podria ser que l'element SECIS es trobès en una regió de seqüència encara per determinar del genoma d'interès.
GPx4
El selenoprofiles va anotar aquesta selenoproteïna utilitzant com a query la Glutatió peroxidasa de Burkholderia graminis (una proteobacteria). Per fer una anotació més acurada vam utilitzar la GPx4 de Melearis gallopavo (el gall d'indi), ja que les aus són filogenèticament molt properes als rèptils. L'alineament resultant era força bo (score=96; U alineada; coverage=0,99), però no es va aconseguir trobar la metionina inicial. Cal remarcar, però, que en l'anotació de GPx4 en Melearis gallopavo tampoc hi ha la metionina inicial.
Selenoproteïnes no trobades
Hem trobat a la literatura que per filogènia, les selenoproteïnes Fep15, SelJ, SelL, SPS2 no s'haurien de trobar en el genoma de Pelodiscus sinensis. Per comprovar-ho, les hem cercat manualment i hem confirmat que no hi són, ja sigui perquè el BLAST no ha trobat cap hit (SelL, SelJ i SPS2) o perquè l'E-value és massa elevat (2,2 en Fep15).
Proteïnes amb cisteïna
SelR3
La metionina sulfòxid reductasa B3, a diferència de la reductasa B1, és una proteïna amb cisteïna enlloc de selenocisteïna. El Selenoprofiles alinea una selenocisteïna de la query amb una cisteïna de la seqüència predita, mentre que s'alineen dues cisteïnes de manera manual. En aquest cas, el primer programa ha realitzat una modificació a la query descrivint-la com a similar a la proteïna original. El gen que la codifica conté 5 exons que es troben separats per introns molt llargs. Per aquest motiu, no s'ha pogut agafar tota la seqüència genòmica a l'hora de realitzar l'alineament i s'ha obtingut un valor d'score de 50 encara que el fragment extret estigués ben alineat.
Per altra banda, encara que no presenti selenocisteïnes, hem trobat un element SECIS loosest. Hi ha dues possibles raons que expliquin aquesta troballa. Per una banda, la cerca del SECISearch de vegades és prou inexacte com per trobar SECIS en proteïnes que en realitat no en tenen. Per l'altra, potser anteriorment aquesta era una selenoproteïna que, per falta de pressió per la seva conservació, ha patit canvis en la seva seqüència però encara manté el SECIS.
SelU1
Es va generar una anotació força acurada d'aquesta proteïna mitjançant la cerca per homologia amb la SelU1 humana, l'espècie amb SelU1 anotada més propera a Pelodiscus sinensis . El Selenoprofiles trobava aquesta proteïna a tres scaffolds diferents i utilitzava tres queries, cap de les quals ens va semblar adequada (d'una família amb similaritat de seqüència o prediccions i cap anotada a les bases de dades com a SelU1). L'alineament obtingut manualment era molt bo (score=98; coverage=1; metionina inicial alineada) i es conservava completament la caixa redox (CFLC). No es va trobar cap selenocisteïna, cosa que contradiu la informació prèvia sobre el selenoproteoma dels vertebrats (representat en l'arbre filogenètic). Tampoc es va trobar cap element SECIS. Aquestes evidències suggereixen que la substitució de selenocisteïna a cisteïna a la SelU1 es va donar anteriorment al que es creia, segurament en l'avantpassat comú entre els mamífers i els sauròpsids (rèptils i ocells) i que els sauròpsids estudiats per a construir l'arbre (lizard, finch i chicken) han revertit la substitució. Aquesta reversió es podria haver produït en l'avantpassat comú d'aquestes tres espècies un cop separat del llinatge de les tortugues, és a dir, en la separació entre Archosauria i Lepidosauria d'una banda i Anapsida de l'altra. Una altra explicació possible és que en la separació entre el llinatge de les tortugues i el dels altres rèptils representats en l'arbre s'hagués produït la substitució. La primera opció és més parsimoniosa perquè assumeix una substitució menys (2 enlloc de 3), per tant constitueix una explicació més probable de la història evolutiva de la SelU1.Rdx12
Es va aconseguir un alineament força bo amb la Rdx12 de Mus musculus (coverage=1, score=99), però no es va aconseguir trobar la metionina inicial (s'alinea una Leucina amb la primera metionina de la seqüència de Mus musculus). En l'alineament es pot observar la conservació del motiu caixa redox ([UC]XX[UC]), un element freqüent en les selenoproteïnes. En aquest cas tant en la seqüència de ratolí com en la de Pelodiscus sinensis s'ha donat la substitució de selenocisteïna a cisteïna. El millor element SECIS trobat era de categoria loosest i estava localitzat força lluny de l'últim exó (2500nt). Aquest fet és conseqüent amb la idea que els SECIS es perden (s'allunyen del consens) quan es produeix la substitució, ja que desapareix la pressió selectiva per a mantenir-los funcionals.
Els elements SECIS i les cisteïnes
En el cas de les selenoproteïnes en les que la selenocisteïna està substituida per cisteïna el fet de no trobar l'element SECIS no és tant important, ja que la seva funció és permetre la incorporació de selenocisteïna. Per aquesta raó el SECIS no s'hauria de conservar i això es reflecteix en que de les 3 substitucions de Sec per Cys el SECISearch troba 1 SECIS de categoria loose, 1 de categoria loosest i en 1 cas no en troba. Si comparem aquests resultats amb els obtinguts en les selenoproteïnes en les quals no s'ha donat la substitució es pot observar una clara diferència pel que fa a la qualitat del SECIS.
Maquinària de síntesi
SecS
Aquesta proteïna forma part de la maquinària necessària per la síntesi i producció de selenoproteïnes. Com que el Selenoprofiles no té proteïnes de maquinària a la seva base de dades, l'hem cercat manualment. SecS presenta molta homologia amb el nostre genoma, presentant uns valors de coverage i score de 0,92 i 93, respectivament. Un altre punt positiu és que si ens fixem en l'alineament, totes dues seqüències comencen amb metionina. Pel que fa a l'element SECIS, sí que s'ha trobat però és de la qualitat més baixa (loosest). No hi ha selenocisteïnes alineades en cap de les dues seqïències però sí que hi veiem més d'una cisteïna alineada.
Efsec
S'ha trobat el factor d'elongació específic de selenocisteïnes al genoma de Pelodiscus sinensis de manera manual, ja que el Selenoprofiles no troba els factors que pertanyen a la maquinària de síntesi de selenoproteïnes. Després de realitzar alineaments amb queries d'Homo sapiens, Mus musculus i Gallus gallus, hem triat l'últim perquè presentava millor homologia de seqüència. El valor d'score obtingut és molt baix, 43, però concorda amb el valor del coverage, 0,45.
Aquesta proteïna no presenta cap selenocisteïna ni cap SECIS, però no ens sorprèn perquè els factors que pertanyen a la maquinària de síntesi de selenoproteïnes no han de ser necessàriament selenoproteïnes.
SBP2
Després de realitzar alineaments manuals amb seqüències de SBP2 conegudes d' Homo sapiens i Gallus gallus , el millor score obtingut és de 51 i el de coverage és de 0,55 amb l'última espècie citada. D'acord amb l'explicació realitzada a l'anterior proteïna, aquesta no presenta ni selenocisteïna ni element SECIS.
SPS 1
La selenofosfat sintetasa 1 forma part de la maquinària de síntesi de les selenoproteïnes. Aquesta proteïna no s'ha obtingut pel Selenoprofiles.
L' alineament obtingut és molt bo, ja que els valors d'score (81) i del coverage (0,82) són elevats. La SPS1 no conté a la seva seqüència cap selenocisteïna, perquè al ser una proteïna de la maquinària de síntesi no té perquè estar formada per una selenocisteïna. Finalment, la falta de SECIS es pot justificar perquè al ser una proteïna de maquinària i no tenir selenocisteïna no necessita elements SECIS per poder sintetitzar-se.
SPS2
L'anotació generada per aquesta proteïna no és gaire bona (score=78; metionina inicial i selenocisteïna no alineades). Per a trobar-la es va utilitzar la SPS2 humana, ja que estava ben anotada i Homo sapiens era una de les espècies més properes amb SPS2 anotada. Es tracta d'una proteïna de la maquinària de síntesi de selenoproteïnes i en humans té selenocisteïna, però en l'anotació generada per a Pelodiscus sinensis no es va trobar cap selenocisteïna. En l'alineament amb la SPS2 humana s'aprecia com falta un fragment a l'inici (amb la metionina inicial) per anotar la proteïna completa.
SEC p43
La proteïna associada a tRNAs de selenocisteïnes o SEC p43 no s'ha obtingut del Selenoprofiles, així que la hem fet manualment.
De la mateixa manera que les anteriors proteïnes, forma part de la maquinària de síntesi de selenoproteïnes, no conté cap selenocisteïna en la seva seqüència ni cap element SECIS. No presenta un alineament gaire bo, ja que els valors d'score (46) i el coverage (0,56) no són gaire elevats. Aquests resultats s'expliquen si tenim en compte que s'han alineat seqüències proteiques procedents d'espècies allunyades filogenèticament: Pelodiscus sinensis i Homo sapiens.
Pstk1
La Pstk1, al igual que les anteriors, forma part de la maquinària de síntesi de selenoproteïnes, no conté cap selenocisteïna en la seva seqüència ni cap element SECIS. Té un alineament molt bo, ja que els valors d'score i coverage són molt elevats, éssent 92 i 0.96 respectivament. Com que ha donat uns bons resultats al comparar dues seqüències procedents d'espècies filogenèticament allunyades (Pelodiscus sinensis i Mus musculus) podem dir que aquesta proteïna està força conservada.
Comentari Selenoprofiles
En alguns casos sorprèn força la query escollida pel Selenoprofiles. Dóna la sensació que o bé la base de dades que utilitza no està actualitzada/completa o que l'algorisme de decisió utilitzat per escollir la query òptima per a fer l'alineament comet algun error.
