
SELENOPROTEÍNAS DE Meleagris gallopavo
MATERIALES Y MÉTODOS: MÓDULO II
MAPA CONCEPTUAL
|
|||
|
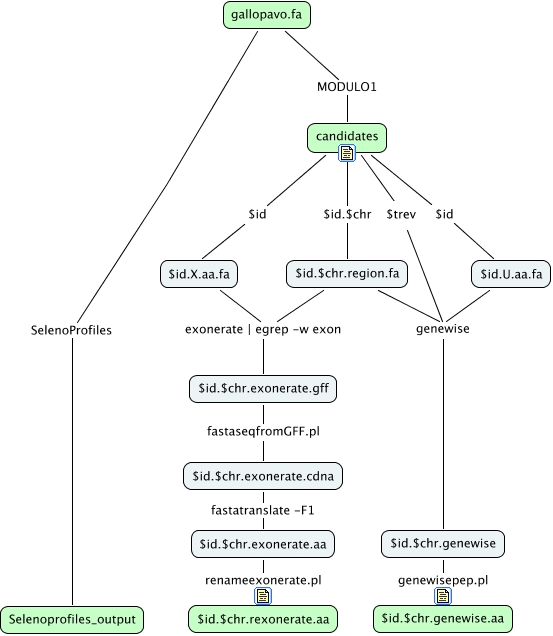
En este módulo partimos de las regiones candidatas a contener selenoproteínas y usamos las selenoproteínas de selenodb como query para predecir la secuencia de las posibles selenoproteínas con exonerate y genewise. Para exonerate usamos las selenoproteínas con X en vez de U y con genewise usamos las selenoproteínas que tienen U. Usamos la variable $trev que informa sobre la orientación del hit de tblastn para hacer la predicción en la orientación pertinente. Cambiamos el nombre del output de exonerate y de genewise para que contengan el nombre del programa y el id de la proteína de selenodb que ha usado como query junto con el cromosoma de la región. También ejecutamos SelenoProfiles a partir del genoma.
BASH script
############################################################################################### ##################################### - MÓDULO 2 - ################################### ############################################################################################### ########################### ANÁLISIS: Predicción proteínas ########################### ############################################################################################### ############################################################################################### #!/bin/bash #$ -o our.stdout #$ -e our.stderr #$ -q llicen.q #$ -N OurJob #$ -cwd export PATH=/cursos/BI/soft/exonerate/i386/bin:$PATH export PATH=/cursos/BI/bin:$PATH export WISECONFIGDIR=/cursos/BI/soft/wise-2.2.0/wisecfg ######################################### EXONERATE ########################################## n=$(wc -l ~/perlscripts/scripts_output/candidates | cut -d ' ' -f1); for line in $(seq 1 $n); do chr=$(head -$line ~/perlscripts/scripts_output/candidates | tail -1 | cut -f2 | tr -d ' '); id=$(head -$line ~/perlscripts/scripts_output/candidates | tail -1 | cut -f1 | tr -d ' '); exonerate -m p2g --showtargetgff --exhaustive yes -q ~/gallopavodb/selenoprots/Xselenoprots/$id.X.aa.fa -t ~/gallopavodb/regions/$id.$chr.region.fa | egrep -w exon > ~/our_results/exonerate/$id.$chr.exonerate.gff; fastaseqfromGFF.pl ~/gallopavodb/regions/$id.$chr.region.fa ~/our_results/exonerate/$id.$chr.exonerate.gff > ~/our_results/exonerate/$id.$chr.exonerate.cdna; fastatranslate -F 1 -f ~/our_results/exonerate/$id.$chr.exonerate.cdna > ~/our_results/exonerate/aa/$id.$chr.exonerate.aa; #renameexonerate.pl > Cambiamos el nombre del output de exonerate y de genewise para que contengan el nombre del programa y el id de la proteína de selenodb que ha usado como query junto con el cromosoma de la región en la que se ha hecho la predicción. ~/perlscripts/renameexonerate.pl < ~/our_results/exonerate/aa/$id.$chr.exonerate.aa $id $chr > ~/our_results/exonerate/aa/$id.$chr.rexonerate.aa; done; ######################################### GENEWISE ############################################ n=$(wc -l ~/perlscripts/scripts_output/candidates | cut -d ' ' -f1); for line in $(seq 1 $n); do chr=$(head -$line ~/perlscripts/scripts_output/candidates | tail -1 | cut -f2 | tr -d ' '); id=$(head -$line ~/perlscripts/scripts_output/candidates | tail -1 | cut -f1 | tr -d ' '); trev=$(head -$line ~/perlscripts/scripts_output/candidates | tail -1 | cut -f7 | tr -d ' '); #la variable $trev que informa sobre la orientación del hit de tblastn para hacer la predicción en la #orientación pertinente. genewise -pep -pretty -cdna -gff $trev ~/gallopavodb/selenoprots/Uselenoprots/$id.U.aa.fa ~/gallopavodb/regions/$id.$chr.region.fa > ~/our_results/genewise/$id.$chr.genewise; #genewisepep.pl > extrae la secuencia peptídica que está comprendida entre las líneas que contienen #las expresiones /pep/ y /\//. ~/perlscripts/genewisepep.pl < ~/our_results/genewise/$id.$chr.genewise $id $chr > ~/our_results/genewise/aa/$id.$chr.genewise.aa; done; ########################################## SELENOPROFILES ################################## #debido a problemas en la instalación al principio tubimos problemas. es muy importante no olvidar el export WISECONFIGDIR=/cursos/BI/soft/wise-2.2.0/wisecfg Selenoprofiles results_folder -t /homes/users/U63748/gallopavo.fa -s "Meleagris_gallopavo" -p eukaryotic
......................................................................................................................................................................................................................................................

