INTRODUCCIÓ
OBJECTIUS DE L'ESTUDI
L'objectiu d'aquest estudi és la cerca de selenoproteïnes en el genoma de Cricetulus griseus, conegut amb el nom de hàmster xinès, ja que el seu genoma ha estat seqüenciat recentment [1] i encara no ha estat analitzat per la cerca de Selenoproteïnes.
LES SELENOPROTEÏNES
El seleni és un micronutrient essencial per a moltes formes de vida ja que intervé en el desenvolupament i contribueix a tenir una vida llarga i saludable. El seu dèficit pot donar lloc a diverses malalties, com per exemple, la malaltia de Keshan. No obstant, un excés de seleni pot arribar a ser tòxic, per aquest motiu els éssers vius l’incorporen a les cèl·lules en forma de selenoproteïnes, mantenint-lo així com a element essencial [2].
Aquestes proteïnes, com ara les glutatió peroxidases, tiorredoxina reductases, i iodotironina desiodinases, tenen funcions antioxidants ja que estan implicades en moltes reaccions redox, essent la selenocisteïna un residu del centre actiu essencial per a l'activitat catalítica de la proteïna. Aquestes també participen en el metabolisme de l'hormona tiroïdal, i en les respostes del sistema immune.
Les selenoproteïnes incorporen a la seva seqüència l’aminoàcid selenocisteïna, codificat per un triplet TGA al DNA; el qual correspon a l’aminoàcid 21 i s’anomena amb la lletra U [3].
Els aminoàcids selenocisteïna i cisteïna es diferencien en un radical: el primer ha substituït el sofre (SH) del radical per un seleni (SeH).
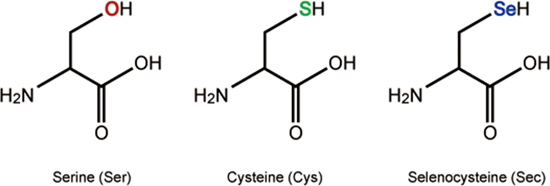
Aquests aminoàcids presenten propietats químiques similars, tot i que el seleni resulta ser un element més reactiu que el sofre. Aquest fet explica que quan es busquen els gens ortòlegs de selenoproteïnes en altres organismes, en ocasions es trobin Cys enlloc de SeCys, ja que en ambdós casos les proteïnes tenen funcions similars. Fins i tot podem arribar a trobar, dins una mateixa espècie, un gen que codifica per una Selenocisteïna i una còpia del mateix gen que conté una cisteïna.
Actualment es coneixen unes 25 famílies de selenoproteïnes en eucariotes, tot i que segurament n’existeixen més. No obstant, el nombre de famílies de selenoproteïnes varia depenent de l’espècie. Per exemple, es coneixen 3 en Drosophila melanogaster i 1 en Caenorhabditis elegans.
Fins fa poc es pensava que les selenoproteïnes eren essencials per la vida animal, donat que tots els animals coneguts tenien selenoproteïnes. Tanmateix, després de l’obtenció de la seqüència del genoma de dotze espècies de Drosophila, es va descobrir, mitjançant anàlisis computacionals, que D.willistoni no té selenoproteïnes i ha perdut la capacitat de codificar-ne. L’anàlisi d’un nombre de genomes d’insectes seqüenciats fa un parell d'anys ha permès comprovar que les selenoproteïnes s’han extingit en moltes altres espècies d’insectes, tot i que n’hi ha d’altres que encara les mantenen. A més a més, altres organismes com les plantes tampoc presenten aquestes proteïnes. Tot això demostra que les selenoproteïnes no són essencials per a la vida i fa que calgui replantejar conceptes que es donaven gairebé per establerts [4].
SÍNTESI DE SELENOPROTEÏNES
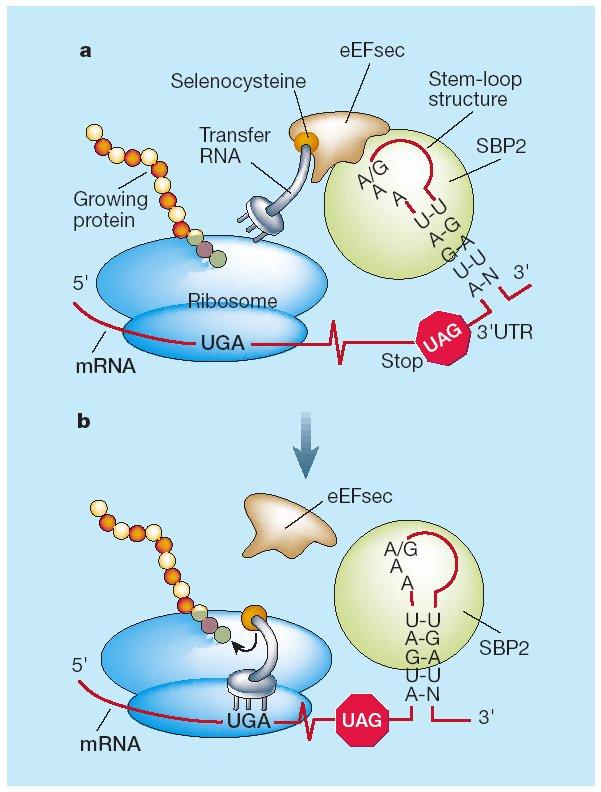
Com ja hem comentat, el codó que codifica per la Selenocisteïna és el UGA, el qual normalment codifica per a un codó Stop, però en determinades ocasions ho fa per a la Sec. És aquesta la raó que explica que moltes selenoproteïnes hagin estat mal anotades.
El motiu que fa que aquest codó no sigui reconegut com STOP és l’existència d’una estructura tridmensional a la regió 3’ UTR del gen (en eucariotes) i en la posició dowstream del codó UGA (en bacteris). Aquesta estructura rep el nom de SelenoCystein Insertion Sequence (SECIS Element). Aquest element recluta una proteïna anomenada SECIS Binding Protein (SBP2), la qual s’uneix a un factor d’elongació específic de selenoproteïnes (Efsec). Efsec aproxima el tRNA de la selenocisteïna al codó UGA del mRNA de les selenoproteïnes i en determina la seva traducció a selenocisteïna. La traducció del mRNA continua fins que el ribosoma reconeix un nou codó de terminació.
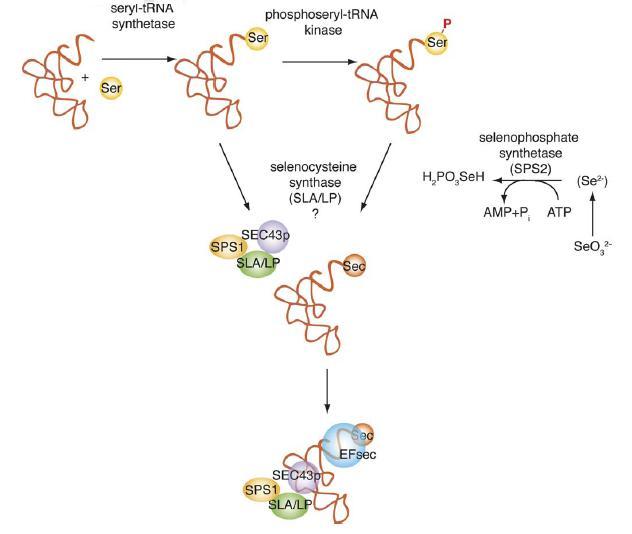
El tRNA se sintetitza a partir de tRNA Ser (serina). Altres proteïnes participen específicament en la síntesi de selenocisteïna. Les quals són la selenofosfat sintetasa (SPS1 i SPS2), la selenocisteïna sintasa (SLA/LP), la fosfoseril tRNA quinasa (PTSK) i la proteïna associada a selenocisteïna (SECP43) [5].
Anotació i predicció
És complicat identificar les selenocisteÏnes, degut al fet que estan codificades per un codó STOP. Per a predir Selenoproteïnes en un genoma que encara no ha estat analitzat, el més còmode és buscar seqüències de Selenoproteïnes ja anotades en el genoma nou, és a dir, buscar proteïnes homòlogues. A més, per a poder anotar els gens de les selenoproteïnes serà necessari tenir en compte la presència d'elements SECIS en la regió 3’UTR. Així doncs, considerarem com a gens candidats aquells que compleixen aquests dos requisits.
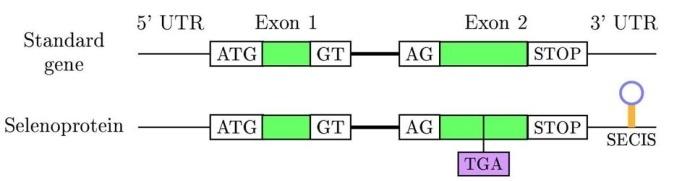
El principal inconvenient és que no hi ha cap conservació a nivell de seqüència entre els SECIS. No obstant, s’ha vist que sí que comparteixen l’estructura tridimensional (loop-helix-loop). Actualment existeixen programes informàtics que determinen l’estructura 3D a partir d’una seqüència donada; així doncs definint un patró estructural dels elements SECIS podrem predir la seva presència en les seqüències que estiguem analitzant.
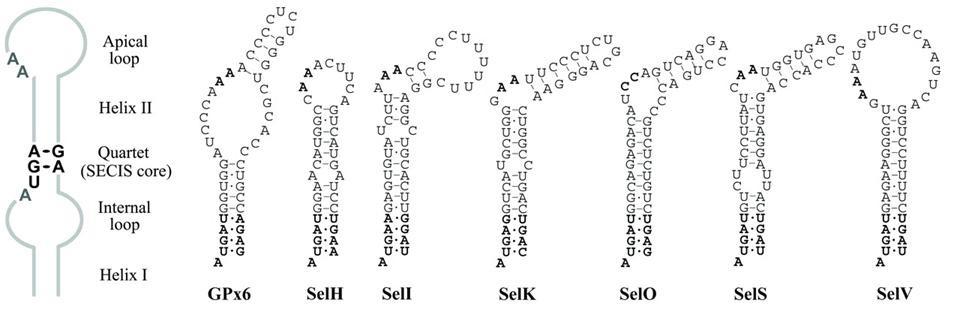
SECIsearch és un dels programes que poden fer aquesta cerca mitjançant la predicció aproximada de l'estructura d'un RNA petit. Se li dóna el patró estructural i un conjunt de seqüències, i el programa mostra quina/es d’aquestes pot adoptar aquesta estructura. Per fer una predicció eficient de la localització de selenoproteïnes se sobreposa la informació d'elements SECIS amb la localització d'exons del gen que estem estudiant [6].
CRICETULUS GRISEUS
Els hàmsters són rosegadors de la subfamília Cricetinae. Aquesta subfamília conté unes divuit espècies, classificades en sis o set gèneres. La majoria són originàries de l'Orient Mitjà i del sud-est dels Estats Units. Com que són fàcils de criar en captivitat, els hàmsters són utilitzats sovint com a animals de laboratori i mantinguts com a animals de companyia en els països econòmicament més desenvolupats.
El hàmster xinès, el nom científic del qual és Cricetulus griseus, procedeix de les estepes i regions semidesèrtiques xineses i mongoles, pertany a l’ordre dels rosegadors i a la classe dels mamífers. A la següent imatge s'observa la seva filogènia.
Cricetulus griseus és un animal interessant en el camp de la recerca ja que les cèl·lules del seu ovari (CHO) són una eina freqüent en la investigació biològica i estan entre les línies cel·lulars més àmpliament utilitzades per a la producció de proteïnes recombinants terapèutiques.
Torna a dalt