Selenoproteïna 1
Cerca en Protistes
El primer pas per la cerca de selenoproteïna 1 en els genomes dels protistes què han estat seqüenciats el darrer any ha estat fer un alineament d’ambdues seqüències amb el TblastN. Hem comprovat tots els E-values i no hem trobat cap hit significatiu. Considerem un hit significatiu aquell que té un valor menor de 10-14. A la següent taula representem els resultats.
| Organisme | E-value |
|---|---|
| Thalassiosira pseudonana | 4,3 |
| Phytophthora sojae | 1,4 |
| Phytophthora ramorum | 0,64 |
| Theileria annulata | 0,24 |
| Theileria parva | 0,007 |
| Babesia bovis | 1,5 |
| Entamoeba histolytica | 0,23 |
| Entamoeba terrapinae | 0,069 |
| Trypanosoma cruzi | 1,00 |
| Monosiga brevicollis | 1,1 |
| Giardia intestinalis | 2,5 |
Llista dels protistes amb els valors de E-value després de l'alineament del genoma corresponent amb la seqüència proteica de Sel1 per mitjà del TblastN.
Tot i no trobar cap hit significatiu vam voler comprobar-ho. Vam agafar el valor més baix, que correspon a Theileria parva (0,007), i vam analitzar el hit obtingut seguint el procés estipulat fins obtenir un resultat negatiu, el qual ens confirma l'absència de la selenoproteïna1 en aquest organisme. L'exonerate ens ha creat un fitxer out en blanc, interpretant d'aquesta manera, que no ha trobat coincidències. En aquest moment, podem afirmar que no es troba selenoproteïna1 en cap dels protistes seqüènciats el darrer any.
Cerca en Plasmodium
Durant la recerca bibliogràfica de Sel1 (veure referència), vam veure que aquesta selenoproteïna ha estat descrita en algunes espècies del gènere Plasmodium. Per aquesta raó vam repetir el protocol en aquests protistes tot i no ser inclosos en la llista dels seqüenciats el darrer any. Veiem els resultats en la següent taula.
| Organisme | E-value |
|---|---|
| P.vivax | 1·10-15 |
| P.knowlesi | 8·10-14 |
| P.Berghei | 6·10-11 |
| P.chabaudi | 5·10-11 |
| P.yoelii | 3·10-12 |
| P.falciparum | 3·10-28 |
| P.gallinaceum | 0,016 |
Valors de E-value de 7 espècies diferents del gènere plasmodium del alineament per TblastN dels seus genomes amb la seqüència de Sel1.
Observem que la Sel1 es troba present en la majoria de les espècies del gènere estudiat, tal com esperàvem. Inesperadament, P.gallinaceum és l’únic Plasmodium que presenta un E-value no significatiu. Els E-values de 10-11 i 10-12 es consideren significatius, tot i no entrar en el rang establert, ja que anteriorment ja s'ha confirmat la presència de Selenoproteïna 1 en aquests protistes. És per aquest motiu que vam decidir seguir el procés amb P.falciparum i P.gallinaceum paral·lelament per comprovar la presència o absència d’aquesta selenoproteïna. El motiu pel qual hem escollit l’espècie falciparum és perquè és la que presenta un E-value més significatiu, per tant, més fiable.
Hem seguit els mateixos passos amb les dues seqüències, però ens trobem que l’exonerate de P.gallinaceum no ens dóna cap resultat. Podem afirmar que, excepcionalment, P.gallinaceum, a diferència de les altres espècies del gènere plasmodium, no presenta Sel1.
Cerca en P.falciparum
Arribats a aquest punt seguim l’anàlisi amb la Selenoproteïna 1 de P.falciparum. El resultat de l'exonerate el podem veure en el següent link: P.falciparum.exonerate
Extraiem el fitxer.gff amb el fastaseqfrom i ens dóna el resultat del següent enllaç que correspon al cDNA de la selenoproteïna 1 predita: P.falciparum.cDNA
Per veure la seqüència proteica fem córrer el fitxer amb el cDNA al fastatranslate de manera que obtenim la seqüència proteica següent: P.falciparum.prot
A continuació, amb la seqüència de la proteïna predita i el programa T-COFFEE fem l'alineament per trobar les regions conservades. El resultat és el següent: P.falciparum.tcoffee
Per assegurar-nos de que realment la seqüència que hem trobat es correspon amb la selenoproteïna fem ús del blastp. Alhora, gràcies a aquest programa podem fer la cerca d'homòlegs.
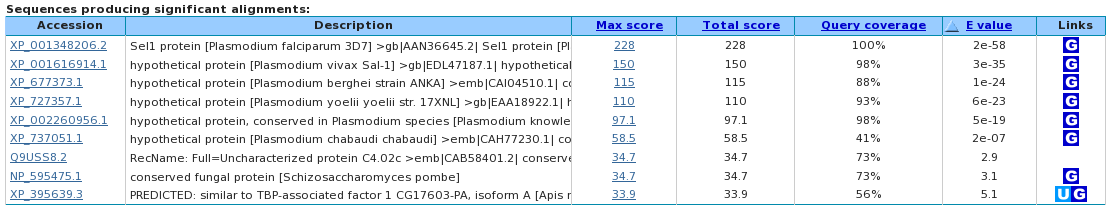
Si introduim la seqüència de selenoproteïna predita de P.falciparum al blastp obtenim com a resultat varis alineaments, entre ells, amb un 100% d'identitat la Selenoproteïna Sel1 de P.falciparum. Entre els homòlegs trobem les possibles selenoproteïnes de les altres espècies de Plasmodium ( P.vivax, P.berghei. P.yoelii, P.knowlesi, P.chabaudi). Amb aquest assaig corroborem els nostres resultats anteriors (obtinguts amb TblastN i Exonerate), determinant que la Selenoproteïna 1 queda restringida al gènere Plasmodium a excepció de l'espècie P.gallinaceum.
Per acabar d'afinar l’anàlisi podem buscar elements que són imprescindibles per la síntesi d'una selenoproteïna. Un d'aquests elements és el SECIS. Tota selenoproteïna disposa d’un element SECIS que té una estructura 3D determinada. SECISearch es basa en aquest caràcter i en la seqüència consens, podem trobar seqüències que es comporten com element SECIS. És evident que si trobem, en la següència de selenoproteïna, un element SECIS podem dir que es tracta d’una cerca positiva, de la selenoproteïna1.
Si utilitzem el programa de cerca de SECIS els resultats que obtenim per P.falciparum són positius. Com era d'esperar, trobem un element SECIS al cromosoma 14:
1 Pfa3D7|chr14|2002.10.03|GENOMIC|TIGR:subseq(131554 10000): 1737 1840 UAUAUAUAUU UUAAAUG AAUAUACUCAUAA AUGAU UUGCUAAUUUGAA AA UACAGAAAAAUAAAUG UAUAAAUUAGUAA UGAG AAAU GAUGUUA UAUUCAUUUA