Discussió
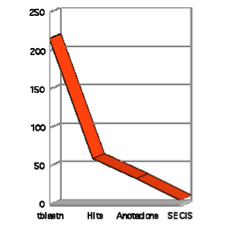
Després d'haver fet tots els passos descrits al protocol, vam pensar que seria interessant incloure un gràfic que resumís tots els resultats obtinguts.
En aquest gràfic veiem com partim de 216 tblastn, mitjançant els quals trobem només 60 hits. Per a fer el gràfic, hem considerat com a hits significatius aquells alineaments predits pel tblastn que presentaven un E-value menor o igual a 10-4.
Cal remarcar però, que l'anàlisi realitzat amb els genomes dels organismes del 2010 el vam fer considerant com a hits els alineaments amb E-value menor que 1.
Durant el treball hem observat diversos aspectes que ens han fet pensar que les selenoproteïnes i els seus homòlegs han patit fortes modificacions al llarg de l'evolució. Mentre realitzàvem la cerca ens vam trobar amb una sèrie de modificacions que ens van cridar l’atenció i que mostrem a continuació:
DI
En fer el t-blastn hem observat que les proteïnes DI1, DI2 i DI3 humanes han sigut alineades, gairebé sempre, en la mateixa regió del genoma en els protistes que les presentaven. Aquest fet, podria explicar-se per una triplicació en humans del gen que codifica per a la proteïna DI.
SelJ
Les espècies L.brazilensis i L.major presenten la proteïna SelJ, i és molt sorprenent que l'Exonerate ens hagi predit que en ambdós genomes hi ha 94 i 95 aminoàcids centrals de la proteïna SelJ inicial que no s'alineen amb la proteïna trobada. A més, aquests 94 i 95 aminoàcids es troben en el mateix lloc en les dues espècies.
En totes les espècies analitzades (excepte en E.huxleyi), les selenoproteïnes i els homòlegs trobats són més curts que la SelJ inicial. En tots els casos (excepte en L.infantum que comença a la posició 127), les proteïnes trobades comencen aproximadament a la posició 110 de la proteïna inicial. Aquest fet podria ser conseqüència d'una deleció a les proteïnes trobades, però el més probable és que hi hagi hagut un problema de seqüenciació o de predicció d'exons ja que és justament al final d’aquesta regió delecionada on s’hauria de trobar la selenocisteïna.
D’altra banda, per a entendre millor les modificacions de les selenoproteïnes trobades respecte les inicials, hem fet alineaments múltiples. La primera proteïna que apareix sempre a l’alineament és la utilitzada per a fer la cerca i la resta són les proteïnes que hem trobat ordenades filogenèticament. Comentarem cadascun dels alineaments:
DI1:

La selenoproteïna DI1 que hem trobat és molt més curta que la humana, però malgrat això, des del seu inici fins al final està molt conservada amb la humana.
DI2:

Igual que passava amb la DI1 trobada, les dues DI2 que hem trobat són molt més curtes que la DI2 humana.
DI3:

Les dues proteïnes que hem trobat al cercar DI3 són diferents. La proteïna DI3 present en D.purpureum presenta un perfil similar al de les altres proteïnes trobades quan vam cercar DI1 i DI2, és a dir, és molt més curta que la humana.
No obstant, la proteïna trobada a E.Huxleyi és molt diferent a les altres proteïnes que hem trobat al cercar DI1 i DI2, ja que aparentment és molt més llarga degut a que conté molts gaps. Per tant, se'ns plantegen dues possibles hipòtesis:
1) Possiblement aquesta proteïna a l’inici era una selenoproteïna, però podria ser que hagués mutat un domini essencial que no hauria de ser necessàriament el domini que contingui la selenocisteïna, ja que aquesta pot haver-se perdut posteriorment com a conseqüència del gran cost que suposa la producció de selenoproteïnes.
2) No ha sigut una selenoproteïna mai.
Com que hem vist que les proteïnes obtingudes amb les diferents DI humanes són molt semblants, hem decidit fer un alineament múltiple de totes elles.

Si ens fixem primer només en les tres DI humanes veiem que les tres són de la mateixa mida. A més, els residus que estan més conservats en totes elles corresponen amb els residus de les proteïnes trobades, exceptuant la DI de E.huxleyi que es troba a l’scaffold 1614, que a més no conté selenocisteïna i per tant no és una selenoproteïna. Per tant, això pot indicar que la regió important per a la proteïna és la que està conservada en tots els organismes i que la part de la proteïna no conservada s’ha perdut en els protistes.
Observant l’alineament veiem que les tres selenoproteïnes humanes són molt semblants entre si i això ens apunta a que provenen d’una triplicació. A més a més, com que comparteixen un alt grau d’identitat de seqüència amb les proteïnes trobades, quan hem cercat qualsevol de les tres DI, hem obtingut hits amb E-values semblants en els mateixos llocs del genoma.
Sel4:
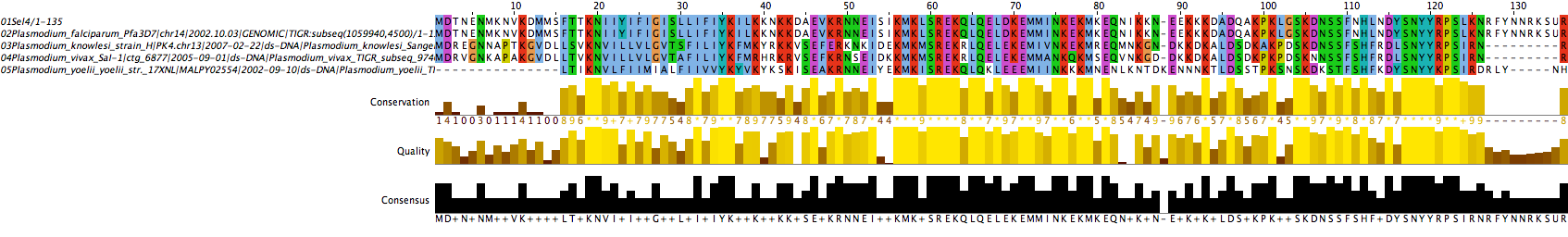
La selenoproteïna 4 inicial es va identificar en P.falciparum; com veiem a l'alineament, la seqüència de la selenoproteïna obtinguda en P.falciparum és idèntica a la inicial.
Altres organismes que presenten Sel4 són P.vivax, P.knowlesi i P.yoellii. Si ens fixem en l'arbre filogenètic que presentem a la plana principal de l'apartat Resultats, veurem que P.vivax i P.knowlesi són dues espècies molt més relacionades filogenèticament que qualsevol d'elles amb P.yoellii. Aquesta és, probablement, la raó per la qual les Sel4 que hem trobat a P.vivax i P.knowlesi són molt més semblants entre si que la Sel4 trobada a P.yoellii, tot i que totes les seqüències estan força conservades.
SelTryp:
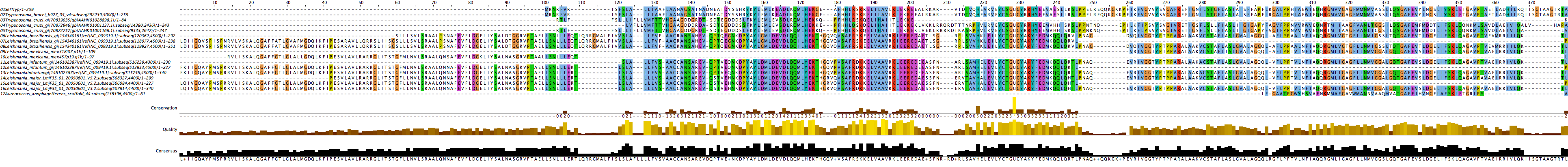
La proteïna que vam utilitzar per a localitzar SelTryp en altres organismes és la descrita a T.brucei. Com que nosaltres també hem cercat aquesta proteïna en T.brucei hem trobat la mateixa seqüència.
Si ens fixem amb l'alineament múltiple, podem observar dos tipus de proteïnes, unes que són més curtes i unes altres més llargues. Les més curtes corresponen a les proteïnes SelTryp que apareixen al gènere Trypanosoma. Les selenoproteïnes més llargues es troben a les espècies del gènere Leishmania.
Per altra banda, hem trobat a A.anophagefferens una proteïna molt més curta que la resta de selenoproteïnes comentades anteriorment. Com que és la que filogenèticament està més allunyada pensem que probablement, en un inici, tots els organismes tenien aquesta selenoproteïna, però que actualment només es conserva com a selenoproteïna en els gèneres Trypanosoma i Leishmania. Aquestes selenoproteïnes contenen la caixa CXXU.
Ens ha sorprès que a totes les espècies hem trobat tres SelTryp, excepte a A.anophagefferens i T.brucei que només n’hem trobat una i a L.mexicana que n'hem trobat dues. D'aquestes dues proteïnes que hem trobat a L.mexicana hem vist que semblen dos fragments diferents de la mateixa proteïna. Aquest fet ens fa pensar que potser en seqüenciar el genoma, els diferents fragments de la proteïna han quedat a contigs diferents, i que per això hem trobat hits “complementaris” a diferents contigs. Una altra opció és que cada part de la proteïna s'hagi especialitzat en una funció determinada i que, per tant, ja no calgui l'altra part de la proteïna i es perdi.
SelJ:
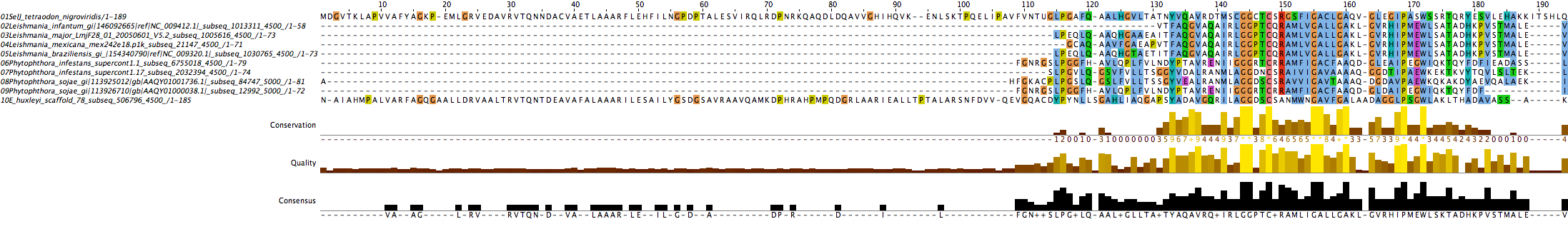
L'alineament que ens ha predit l'Exonerate per a les proteïnes SelJ a L.brazilensis i L.major no coincideix amb l'alineament que ens ha fet el T-coffee. No obstant, pensem que és més fiable el resultat que ens ha donat el T-coffee perquè gairebé totes les altres SelJ que hem trobat s'alineen de la forma que ens ha predit el T-coffee.
E.huxleyi és l’única espècie en la que hem obtingut una proteïna de la mateixa longitud aproximadament que la SelJ inicial, però si comparem les seqüències d’ambdues proteïnes (SelJ original i SelJ a E.huxleyi) ens adonem que els residus no estan gaire conservats.
A més, cal destacar que en totes les proteïnes SelJ identificades observem que la conservació dels residus comença en els aminoàcids 115.
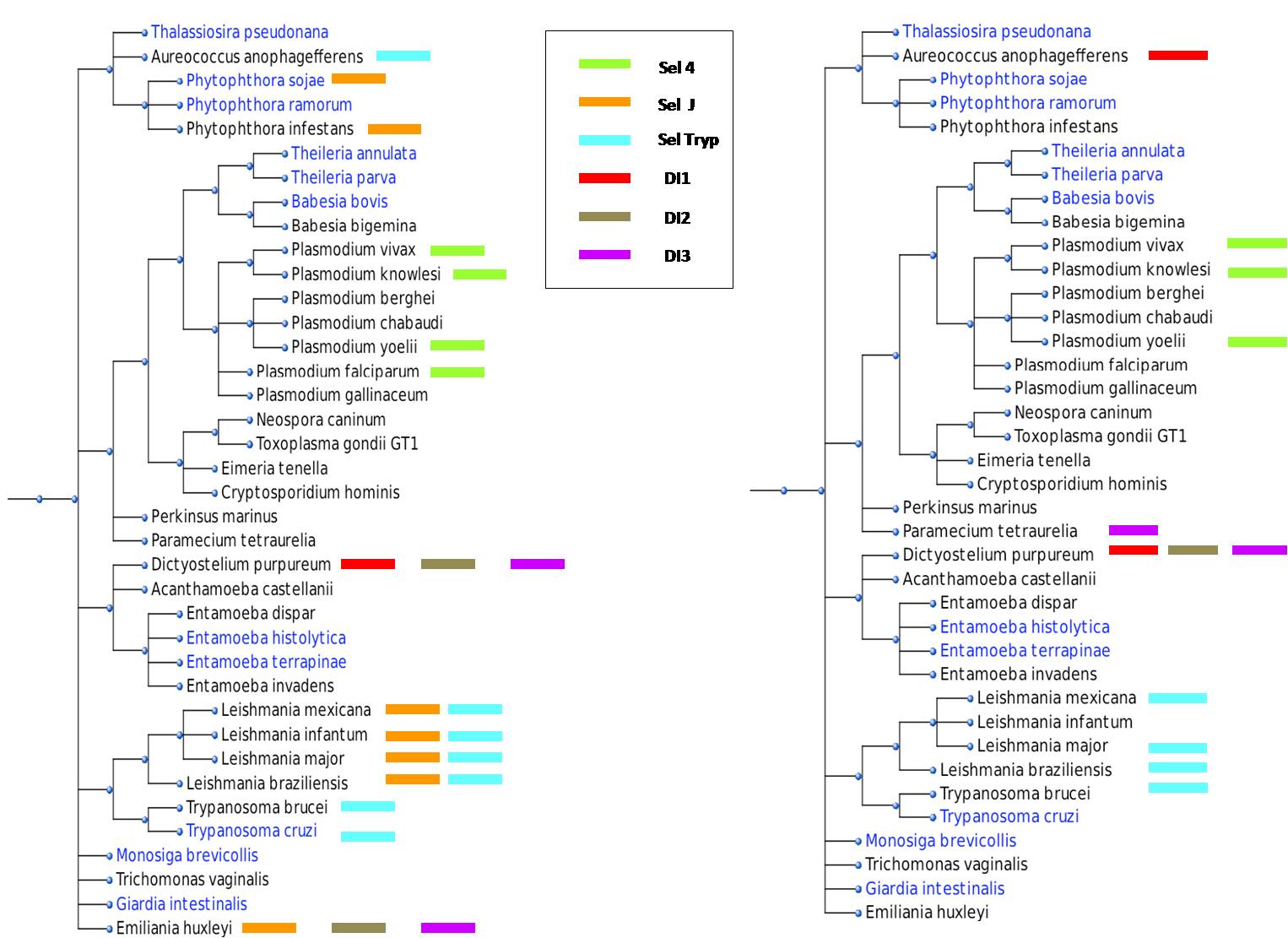
Per tal d'analitzar la distribució de les selenoproteïnes trobades, hem pensat utilitzar l'arbre que presentàvem a la plana principal de Resultats (arbre de l'esquerra), així com un altre (arbre de la dreta) en el que veiem les selenoproteïnes que han trobat els autors de projectes d’altres anys en les mateixes espècies (excepte les seqüenciades aquest darrer any que apareixen en blau). Aquesta comparació ens serveix per a verificar els nostres resultats, ja que la major part de selenoproteïnes que hem trobat en els organismes coincideixen amb les que van trobar els diferents autors de projectes d’altres anys.
Tant nosaltres com els autors dels projectes d’altres anys, hem trobat Sel4 en diferents espècies del gènere Plasmodium.
A més, hem localitzat i per tant, verificat amb els resultats d’anys anteriors, les proteïnes DI1, DI2 i DI3 a Dictyostelium purpureum. En el cas de l’èspecie Paramecium tetraurelia, altres autors han trobat DI3 i nosaltres no hem trobat cap selenoproteïna. Mirant l’E-value que vàrem obtenir, ens adonem que malgrat que és un E-value baix (3x10-5), l’Exonerate no ens ha trobat l’anotació.
És important comentar que la proteïna SelJ hem observat que està conservada en espècies del gènere Leishmania i del gènere Phytophthora. Aquest resultat no l’hem pogut comparar amb els resultats d’altres anys ja que en projectes anteriors no es va cercar la proteïna SelJ.
En el cas de les selenoproteïnes Tryp, les hem trobat a espècies del gènere Leishmania i Trypanosoma, obtenint el mateix resultat que en anys anteriors. En el cas de l’espècie Aureococcus anophagefferens, nosaltres hem localitzat la proteïna SelTryp, mentre que altres autors han localitzat la DI1. Això pot ser degut a que nosaltres hem utilitzat les proteïnes DI humanes per a la cerca, mentre que altres autors van partir de les DI de Danio rerio.
Per últim hem trobat SelJ, DI2 i DI3 a Emiliania huxleyi, mentre que els autors dels projectes d’altres anys no han trobat cap selenoproteïna en aquesta espècie.
Hem trobat un element SECIS de baixa energia lliure (-28,06) a E.huxleyi en la regió 3’ del gen de la proteïna DI3 localitzada a l’scaffold 1614. Ens ha sorprès molt trobar aquest SECIS ja que la proteïna DI3 hem vist abans (a l’alineament de les 3 DI) que no conté cap selenocisteïna i per tant no hauria de contenir elements SECIS.
Això podria ser degut a que realment aquesta proteïna és una selenoproteïna però hi ha hagut problemes en la seqüenciació i s’ha perdut la selenocisteïna (U).
L’altre opció és que aquesta proteïna hagués sigut anteriorment una selenoproteïna i hagués perdut la selenocisteïna mantenint, però, l’element SECIS.
Amb la cerca de proteïnes no redundants amb l’NCBI, hem vist que les selenoproteïnes no només estan conservades entre els protistes, sinó que també estan conservades en animals, plantes i fongs.