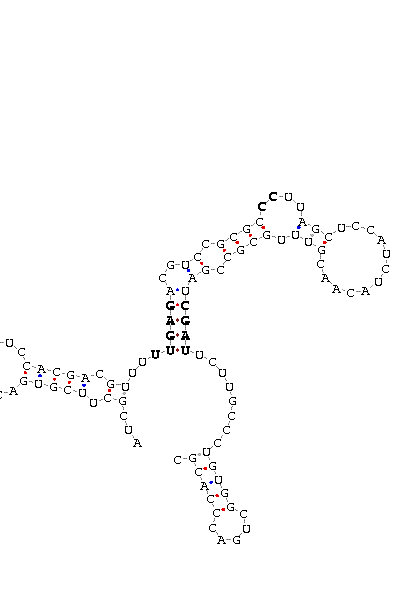RESULTATS TR (supercontig 62)
Seqüència de la selenoproteïna homòloga a TR3:

Seqüència de nucleòtids on es troba la proteïna:

Discussió de la proteïna:
Aquesta proteïna és una selenoproteïna homòloga a la proteïna TR3 humana i conté 2 exons i 1 intró. En realitat, aquesta proteïna és homòloga a les tres selenoproteïnes de TR conegudes però, a partir del Blast i del posterior ClustalW, hem pogut comprovar que té més similituds a la proteïna TR3 i a la TR1. Després de realitzar el tBlastN, hem vist que l'Score i l'E-Value són molt significatius i similars a la proteïna TR1 i TR3 humanes (tot i que lleugerament més semblant a aquesta última). Per tant, hem pogut arribar a la hipòtesi de que ens trobàvem a una regió del nostre organisme molt semblant a la de TR3 humana. Quan ens hem fixat en l'alineament, hem vist que, on la proteïna humana tenia una selenocisteïna, la nostra proteïna contenia un stop (en aquest cas UGA), per tant, hem pogut començar a hipotetizar que, amb molta probabilitat, estem davant d'una selenoproteïna.
TR1: Score = 486, Expect(3) = e-136
TR2: Score = 464, Expect(3) = e-130
TR3: Score = 489, Expect(3) = e-137
Tot seguit hem realitzat el genewise,que ens ha mostrat un intró en una zona que amb el Blast nosaltres havíem vist que estava bastant conservada. és per això que hem realitzat l'Exonerate. Els resultats obtinguts a partir d'aquest programa tampoc els hem trobat fiables del tot, ja que ens marcava un intró en un lloc diferent al que ens marcava el genewise i que tampoc hem vist que era gaire coherent.
Per poder tenir una altra informació sobre els possibles introns que conté la nostra proteïna, hem mirat a la base de dades que té el Broad Institute que ens dóna informació sobre els transcrits. Hem blastejat la proteïna TR3 humana amb els transcrits de la base de dades (basats en EST i en prediccions del Geneid) , i hem obtingut un e-value molt significant amb un transcrit del mateix supercontig que estàvem buscant (el 62). Hem comparat les seqüències d'aminoàcids, i hem vist que és gairebé idèntica a la nostra. Tot i així, la figura que mostren a la base de dades creiem que està equivocada:

Està equivocada perquè consideren l'UGA de la nostra selenoproteïna com si fós un stop, i l'integren dins d'un suposat intró. Després consideren que hi ha un altre exó que nosaltres considerem inexistent, ja que després de la selenocisteïna, trobem una glutamina i finalment un codó stop.
Tot i així, la seqüència aminoacídica del transcript ens ha servit per predir el primer intró que no sabíem ben bé on col·locar.
D'aquesta manera, hem obtingut dues possibles seqüències aminoacídiques, una basada a partir de l'informació del genewise, i l'altre a partir de l'informació obtinguda del transcript.
Resultat GFF
Seqüència a partir del Genewise
Seqüència a partir del Transcript
Per saber quina de les dues seqüències és la correcta i per confirmar a quina proteïna humana s'assemblen més, les hem posat al ClustalW juntament amb les seqüències de les proteïnes humanes.
El resultat que hem obtingut del ClustalW, no coincideix amb el resultat del Blast, ja que a partir del ClustalW hem obtingut un score major en comparar la TR1 i la nostra selenoproteïna. Això és degut a que el Blast compara només una part de la proteïna, i en canvi, el ClustalW agafa tota la proteïna sencera.
Finalment, per comprovar que realment es tracta d'una selenoproteïna, hem buscat possibles elements SECIS que ens poguessin fer concloure l'existència real d'aquesta. Els resultats obtinguts, però no han estat satisfactoris, ja que tots els Secis predits presenten un COVE score de 0, quan el mínim recomanat és de 15.
De tota manera, creiem que l'score no és l'únic factor a tenir en compte a l'hora de determinar els Secis, ja que s'ha de mirar la posició en la que aquests es troben a la seqüència genòmica i si són viables respecte a la regió codificant de la selenoproteïna. Tot i que tots els Secis predits tenen un score de 0, creiem que els següents són possibles candidats per tractar-se del Secis de la proteïna estudiada.
P. infestans supercont1.62 of Phytophthora infestans [DNA: _394018-399730_- 1123 upstemen: -6.70 fullstructen: -14.80 UCUCGCUUGC GUAGCUG AUACUUGCGGAUA UUGAC ACACCUAGAAU GG UGGAAUUUUCGAG CUCAGGGUUGU CGAU CCAGA CGAUGGC GUACUCCCAG COVE score: 0 (Recommended threshold for COVE: 15)
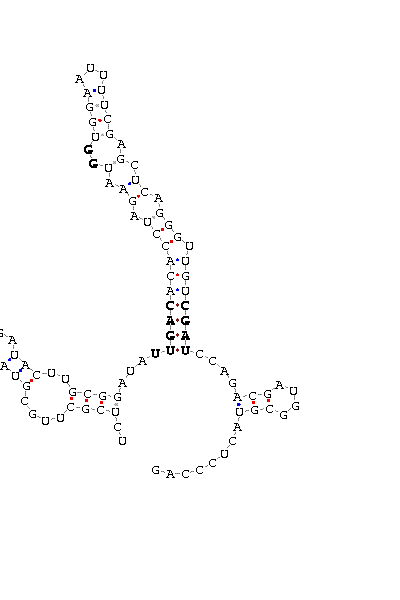
P. infestans supercont1.62 of Phytophthora infestans [DNA: _391018-403730_- 10850 upstemen: -8.40 fullstructen: -18.20 ACUAUCGAUA CCUUAGC UAACGUGCAGUG UUGAA UGUAAUUAGC GA GUUGCGACCUGCAUUACAUUGCAAA GCCAGCACA UGAG GCUGAU GUUCGAG CAAUUCCCGG COVE score: 0 (Recommended threshold for COVE: 15)
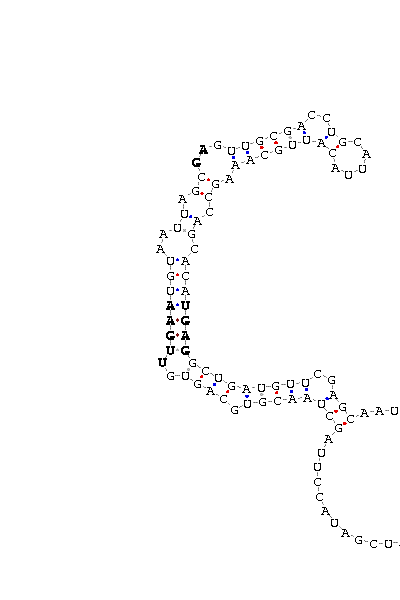
P. infestans supercont1.62 of Phytophthora infestans [DNA: _391018-403730_- 11385 upstemen: -7.73 fullstructen: -14.73 CGGGUGACGA UGUGGAU GAUUGUGGCGACU GUGAG UUUGGUCAAGCU GA UAUUGAGCAUACCGAAGAGAGCUU GCUUGCCGUGGG CGAU CAU UGCAGUA AGACUAGGAC COVE score: 0 (Recommended threshold for COVE: 15)
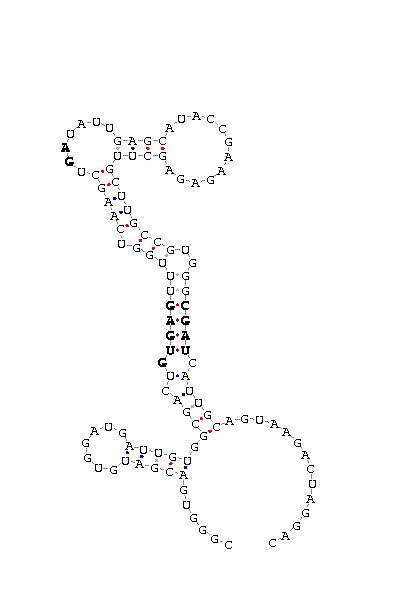
P. infestans supercont1.62 of Phytophthora infestans [DNA: _391018-403730_- 9843 upstemen: -5.39 fullstructen: -16.79 AUCGCUUCGU GACCCUG AUCCACGACGUUU UUGAG ACGUCCGCGC CC UUAGCUCCAUCUACAACGU UUGCGCCGAU CGAU UCUUGCC CUGUGGC UGACCCACGC COVE score: 0 (Recommended threshold for COVE: 15)