
RESULTATS TR (supercontig 28)
Seqüència de la proteïna homòloga a TR2:

Seqüència de nucleòtids on es troba la proteïna:
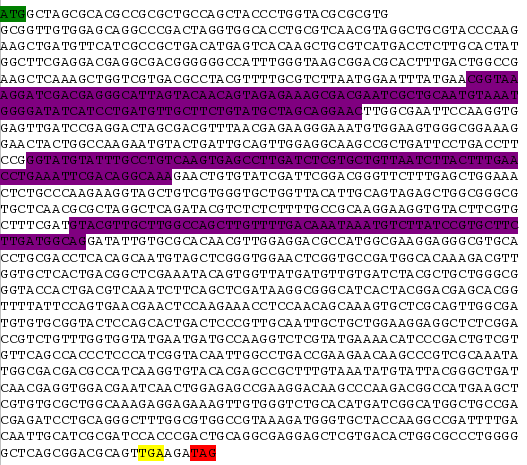
Discussió de la proteïna:
Aquesta proteïna és una selenoproteïna homòloga a la proteïna TR2 humana i conté 3 introns i 4 exons.
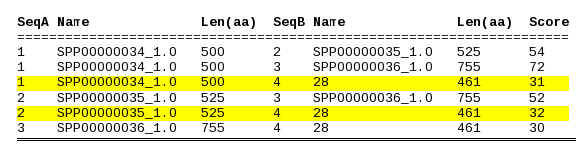
Quan hem realitzat el tBlastN, hem vist que els valors obtinguts del supercontig 28 eren molt semblants en TR1 i TR2, tot i que en aquest últim l'score és una mica més elevat, i l'e-value més baix. És per això que podem dir que en aquest cas la TR2 humana és homòloga a la nostra proteïna. A més, ens hem hagut de fixar en els 3 hits obtinguts en el Blast corresponents al supercontig 28 per poder deduir que la nostra selenoproteïna conté 3 introns, ja que el Blast en trobar-los, ha considerat que l'score havia baixat massa per considerar els 3 hits com a la mateixa proteïna.
TR1: Score = 137, Expect(3) = 7e-41
TR2: Score = 140, Expect(3) = 2e-49
TR3: Score = 125, Expect(3) = 7e-28
A partir del Genewise hem pogut deduir que la proteïna conté 3 introns. Això ens ha lligat amb el fet que a partir del tBlastN havíem obtingut els 3 hits en el mateix supercontig que corresponen a la mateixa proteïna, però dividida pels introns.
Obtenir GFF
Per poder confirmar que la nostra selenoproteïna realment és homòloga a TR2, hem realitzat un ClustalW comparant-la amb totes les homòlogues humanes.
Finalment, per comprovar que realment es tracta d'una selenoproteïna, hem buscat possibles elements SECIS que ens poguessin fer concloure l'existència real d'aquesta. Els resultats obtinguts, però no han estat satisfactoris, ja que tots els Secis predits presenten un COVE score de 0, quan el mínim recomanat és de 15. Tot i així mirant l'estructura del SECIS, hem pensat que els possibles candidats podrien ser:
P. infestans supercont1.28 of Phytophthora infestans [DNA: _1895880-1902700_+ 100 upstemen: -7.51 fullstructen: -17.01 GCUUGGCGCC AUGCUUC UGAUUGAGUUG CUGAA CUAACUGGUGU CA UGGUCAUCUCGCAUUUUAACAGG CCGCCGGAUAG CGAC AUGAUU GGAUUCU CUAUUGACGA COVE score: 0 (Recommended threshold for COVE: 15)
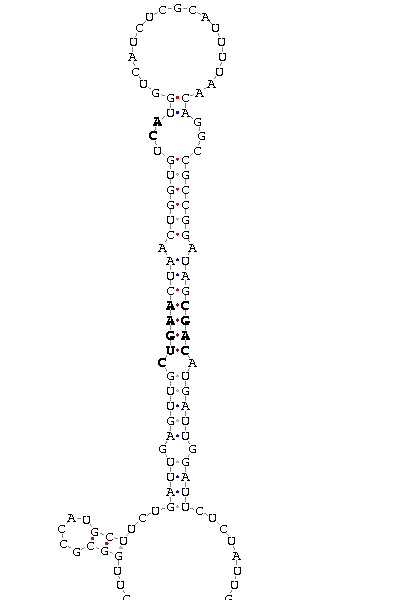
P. infestans supercont1.28 of Phytophthora infestans [DNA: _1893880-1904700_+ 9055 upstemen: -5.20 fullstructen: -14.73 CGCCUUGUCC GCAUCUG UAGCAGG GUGAA UGACAGUCUUCG CA CCGUUGGGGUCGUUGGUA UUGCGGAUUUCA AGAG GCACCUUGCC CCACUGC GCUGGAAUGA COVE score: 0 (Recommended threshold for COVE: 15)
