RESULTATS Sel I (Supercontig 3)
Seqüència de la proteïna homòloga a Sel I:

Seqüència de nucleòtids on es troba la proteïna:

Discussió de la proteïna:
Després de realitzar el tBlastN , hem vist que l'Score i l'E-Value són molt significatius en dues regions del supercontig 3, aquestes dues regions són molt properes i les estudiarem conjuntament per la caracterització de la proteïna, ja que formen part de la mateixa.
Tot seguit hem realitzat el genewise per determinar els introns i exons del gen. Al realitzar el genewise en les dues regions hem pogut comprovar que: el segon intró trobat en la primera regió correspon amb l'intró trobat en la segona regió. En el link es mostren els dos resultats obtinguts en el genewise Els resultats indiquen que és una proteïna homòloga a SelI que conté 3 exons i 2 introns. A partir d'aquí hem seguit amb una seqüència de nucleòtids comuna per ambdues regions. A l'inici de la pàgina es mostra en groc la regió homòloga, troaba en l'alineament del BLAST, del primer hit i en blau la del segon hit.
Per poder tenir una altra informació sobre els possibles introns que conté la nostra proteïna, hem mirat a la base de dades que té el Broad Institute que ens dóna informació sobre els transcripts. Hem blastejat la proteïna humana amb els transcripts de la base de dades (basats en EST i en prediccions del Geneid) per obtenir l'estructura del transcrit que correspon a la proteïna caracteritzada.
Els resultats del BLAST amb el transcrit mostren un E-value molt significatiu amb un transcrit de Phytophthora infestans. Hem mirat la seqüència d'aminoàcids i hem vist que és gairebé idèntica a la nostra. A més a més veiem que la figura i la informació del transcrits de la seva estructura gènica coincideix amb la predita per nosaltres.
Al trobar la seqüència final de la nostra proteïna ens hem adonat que el codó que trobàvem com a STOP corresponia a un TGA, i que pocs nuclòtids després hi havia un altre codó STOP. Ni el BLAST inicial ni el genewise ens han arribat a alinear la U amb un aminoàcid, per tant de moment no sabem si realment és significatiu haver trobat un codó TGA en aquesta posició.
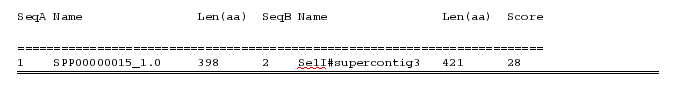
Seqüència de la proteïna considerant que conté la selenocisteïna: Com que no és possible poder alinear la U amb els mètodes utilitzats prèviament (BLAST i genewise), hem realitzat un ClustalW de la seqüència protèica, considerant el codó TGA com una selenocisteïna, amb la selenoproteïna SelI humana.
Els resultats no són gaire bons, trobem un alineament amb un score de només 28, i a més a més no s'alinea la U de la selenoproteïna humana amb la potencial selenocisteïna de la nostra proteïna. Tot i que la ditància entre les dues selenocisteïnes no és molt gran observem que els aminoàcids posteriors no estan conservats.
A partir d'aquests resultats determinariem que la nostra proteïna és homòloga a la SelI humana, però que no es tracta d'una selenoproteïna.
Per fer una altra comprovació hem realitzat una cerca d'elements SECIS . Amb aquesta cerca hem obtingut resultats, però cap de prou significatiu (donant com a significatius SECIS amb un score de més de 15) com per considerar els anteriors resultats com a determinants perquè la proteïna a caracteritzada sigui una selenoproteína. En el link mostrem el SECIS més probable , però que no l'hem considerat, ja que té un score de 0.
Amb aquesta decisió considerem que la proteïna, el transcrit, predit pel Broad Institute és correcte, ja que s'aproxima a la caracterització de la nostra proteïna considerant el TGA com a codó STOP; i per tant la proteïna caracteritzada es tracta d'una proteïna homòloga a la SelI humana, però que no conté selenocisteïna en la seva estructura.
Resultat GFF