
RESULTATS GPx (Supercontig 36)
Seqüència de la proteïna homòloga a GPx8

Seqüència de nucleòtids on es troba la proteïna:

Discussió de la proteïna:
En el supercontig 36 trobem, mitjançant el tBLASTn, tres regions en el genoma homòlogues a proteïnes humanes de la família GPx. Els alineaments mostren E-values significatius i scores alts. A continuació es mostren els resultats d'e-value i score obtinguts per cada alineament de la proteïna humana:
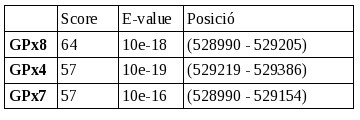
Tots els hits pertanyen a la mateixa regió del genoma, per tant, podem pensar que els alineaments de les diferents GPx humanes són tots amb la seqüència genòmica d'una sola proteïna. Aquests resultats es dónen perquè totes les GPx humanes són molt semblants.
Per determinar a quina de les proteïnes humanes és més homòloga aquesta regió del genoma, en el supercontig 36, ens basarem en els valors d'E-value i d'score mostrats en els resultats del BLAST. En aquest cas hem determinat que aquesta regió presenta més homologia amb la GPx8 humana, ja que presenta un score més alt i E-value més baix, és a dir els valors més significatius. A partir d'aquí ens centrarem en l'estudi d'aquesta regió utilitzant com a base el resultat del BLAST de la GPx8.
Hem de tenir en compte que la GPx8 no es una selenoproteïna, i tampoc ho és la homòloga que hem trobat. En ambdós casos les dues contenen cisteïna en la seva seqüència en la posició on d'altres GPx contenen una selenociteïna.
Aquesta proteïna homòloga a la GPx8 humana conté dos introns i tres exons, tal i com ens indiquen els resultats del genewise i els transcrits mostrats en la pàgina web del Broad Institute.
Resultat GFF

Tot i que el nombre d'introns i exons és coincidideix, és a dir trobem la mateixa estructura gènica, al final no obtenim la mateixa seqüència d'aminoàcids després de realitzar el genewise i buscar la metionina i l'STOP, que la dels transcrits. Com podem veure a continuació una és de les seqüències és més llarga que l'altra.
Seqüència genewise

Seqüència Transcript

En aquest cas per determinar quina de les dues seqüències d'aminoàcids possibles és la més probable de ser la correcte, i quina de les dues és la que presenta més homologia amb la seqüència humana, hem realitzat un ClustalW, els resultats del qual es mostren a continuació:

A partir del ClustalW veiem que hi ha una gran similitud entre la seqüència que hem caracteritzat i la GPx8 humana. Observem que hi ha un fragment comú entre aquestes dues seqüències que no apareix en la seqüència del transcrit. Aquest resultat ens portaria a deduir que és més probable que la seqüència correcta sigui la caracteritzada per nosaltres, ja que presenta més homologia. En aquest cas no hem tingut en compte l'score dels diferents alineaments ja que no era significatiu per poder prendre la decisió.