
RESULTATS Gpx (supercontig 67)
Seqüència de la selenoproteïna homòloga a GPx8:

Seqüència de nucleòtids on es troba la proteïna:

Discussió de la proteïna:
En el supercontig 67 el BLAST ens ha mostrat 7 hits significatius per proteïnes humanes de la família GPx. En aquesta taula es mostren els resultats dels valors d'E-value i score pels diferents alineaments:
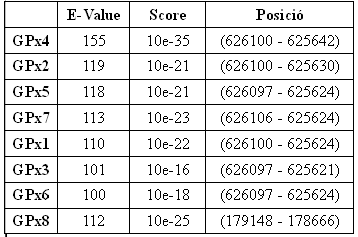
Tots els hits, excepte el de Gpx8, s'han alineat a la mateixa zona. és a dir, que tots estan alineats amb la mateixa potencial proteïna de Phythophthora infestans, perquè. Aquest resultat és degut a que totes les GPx humanes són molt semblants entre elles. Escollirem d'entre elles, per analitzar-ne l'alineament, la que tingui un E-value i score més significatius, en aquest cas determinem que la regió és homòloga a GPx4.
Al buscar la seqüència de la proteïna, trobem que és idèntica a la que ja havíem trobat al supercontig 56. En un principi, podem pensar que és tracta d'una duplicació, però si tenim en compte que Phythophthora infestans és un protista i que, a més, el seu genoma no està encara ben ensamblat, sembla que el més probable és que tornem a veure exactament la mateixa proteïna.
Veure resultats GPx4 al supercontig 56
L'altre hit significatiu i que correspon a una altra regió del supercontig 8, és l'homòloga a GPx8, que en el humans no es tracta d'una selenoproteïna. Comprovant els resultats del tBLASTn podem veure que tampoc ho és l'homòloga de GPx8 en Phythophthora infestans.
Els resultats obtinguts en realitzar el genewise no indiquen la presència de cap intró. Per comprovar aquests resultats, busquem els transcrits en la pàgina del Braod Institute i , tot i que no hi ha cap intró, observació que coincideix amb la nostra predicció, allarga la seqüència uns quants aminoàcids més per davant de la primera metionina respecte la proteïna predita per nosaltres.
Resultats GFF

Per comprovar quina de les dues seqüències és més probable que sigui la correcte, realitzem un Clustalw amb les dues seqüències (la obtinguda a partir del genewise i l'ExPASy, i la del transcrit) i la proteïna GPx8 humana.
Veure input
Els SCORES no ens donen gaire informació, però la representació JalView ens ajuda a prendre una decisió.
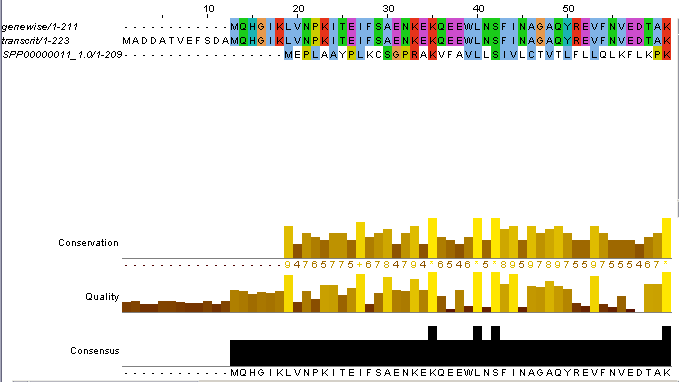
En aquesta representació veiem com els aminoàcids que ha allargat el transcrit no estan alineats amb la proteïna humana, per tant el més adient és escollir com a correcta la que hem trobat nosaltres a partir del procediment explicat a materials i mètodes.