La cerca de selenoproteïnes a un genoma representa un repte ja que els programes de predicció de genes habituals no dónen bons resultats a causa de les particularitats d'aquestes proteïnes. Aquest protocol informa del passos bàsics per predir selenoproteïnes a un genoma, i és el que hem seguit per a fer prediccions a Leishmania major.
1. Obtenció del genoma de Leishmania major
El genoma de Leishmania major es pot trobar a dues bases de dades:
- NCBI, cercant pel nom de l'organisme a Genomes. Es pot trobar el genoma complet seqüenciat i ordenat per cromosomes.
- GeneDB, una base de dades que conté genomes de diverses espècies i que permet fer cerques de BLAST contra el genoma complet de l’espècie seleccionada.
- Genoma aportat pel professorat al directori d'estudiants.
2.Obtenció de la seqüència de selenoproteïnes
Les seqüències de les selenoproteïnes conegudes s'obtenen de la base de dades SelenoDB, descarregant-les individualment en format FASTA.
Donat que a priori no coneixíem quines selenoprote&ines eren presents al genoma del nostre organisme, vam cercar les seqüències de totes les selenoproteïnes contingudes en aquesta base de dades.
3. Cerca de similaritat: BLAST
En primer lloc, cal remarcar que en tot moment hem treballat amb dades en format FASTA.
Donada la facilitat d'utilizació del GeneDB, vam utilitzar aquesta base de dades per fer un garbellament inicial que permetés tenir una primera visió del selenoproteoma de Leishmania i poder descartar aquelles selenoproteïnes que clarament no donessin resultats significatius.
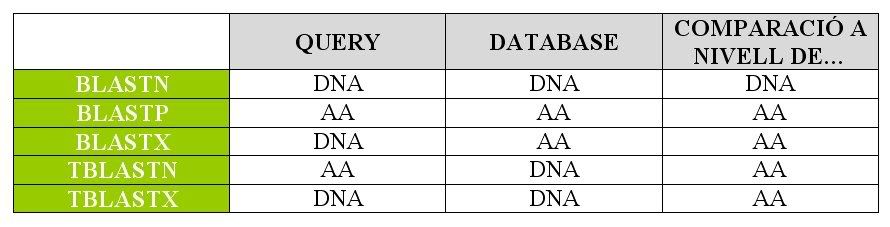
Per fer aixó cal blastejar les seqüències de les selenoproteïnes obtingudes de SelenoDB contra el genoma complet de Leishmania major . La opció que vam escollir és TBLASTN,que alinea DNA contra proteïnes tal i com s'explica al quadre. El criteri de selecció per a considerar un alineament com a significatiu va ser un E-value inferior a 10-10.
Així vam obtenir un conjunt de possibles selenoproteïnes de Leishmania major , les quals vam passar a analitzar més detalladament.
Vam utilitzar el genoma aportat pel professorat al directori d'estudiants per tal de facilitar la manipulació de les dades. Tots els passos mencionats a continuació van ser realitzats a la finestra terminal. Els passos seguits van ser:
- 1.Formatejar la base de dades amb la comanda
formatdb -i genome.fa -p F
on genome.fa és la base de dades que conté el genoma de Leishmania.
- 2.Fer la cerca de similaritat amb TBLASTN
blastall -p TBLASTN -i (query) -d genome.fa | more
on la query és la proteïna a blastejar. Amb aquest pas aliniem les proteïnes amb el genoma. La opció more permet observar els aliniaments obtinguts un a un a la finestra terminal per fer una primera avaluació de la seva qualitat.
- 3.Selecció dels aliniaments més significatius
blastall -p tblastn -i hsap.fa -d dmel_genomic.fa -m 9
Amb la opció -m 9 podem veure els aliniaments ordenats i tabulats mostrant informació referent a cadascun.
Així vam poder veure els aliniaments ordenats segons el seu E-value i descartarem tots aquells que tenien un E-value superior a 10-10. Dels restants, vam triar aquell que tenia l' E-value més petit.
4. Obtenció de la seqüència genòmica
La comanda anterior també aporta informació referent a la regió cromosòmica on es troba cadascun dels hits obtinguts. Una vegada triat l'aliniament òptim assumim que les coordenades d'aquest hit corresponen a la seqüència dins el genoma de Leishmania de la selenoproteïna que s'està estudiant.
Per poder seguir treballant, vam tallar aquesta seqüència mitjançant la comanda:
fastasubseq -f genome.fa -s (inici) -l (llargada) > subseq.fa
on subseq.fa és l'arxiu on redireccionem el fragment tallat.
Si la seqüència a tallar es troba en sentit revers són necessaris una sèrie de passos per procedir a tallar.
- Fer un índex del genoma en format multifasta amb la comanda fastaindex genome.fa genome.index
- Extreure la seqüència amb fastafetch genome.fa genome.index (fragmenta a tallar) > (arxiu on es redireccionarà)
- Extreure el fragment d'interès fastasubseq -f (arxiu redireccionat) -s (inici) -l (llargada) > (nom d'arxiu per redireccionar)
- Canviar el sentit el fragment extret amb la comanda fastarevcomp (nom de l'arxiu) > (no d'arxiu per redireccionar)
Cal fixar una llargada superior a la que indica el resultat de l'aliniament per tal d'incloure l'element SECIS a la seqüéncia.
5. Predicció de l'estructura exònica i la seqüència proteica
Un cop vam tenir en un fitxer la seqüència genòmica, el que volíem era predir, per una part, l'estructura exònica del gen i, per l'altra, la seqüéncia de la proteïna resultant.
El programa que vam utilitzar per aquests dos objectius va ser Genewise. Per fer-lo servir cal introduir:
- Seqüéncia proteica de SelenoDB
- Seqüéncia genòmica extreta de l'aliniament
Vam marcar les opcions translation, cDNA i GFF output. L'aliniament va ser global, ja que així evitàvem que regions de baixa homologia afectássin un aliniament bo per la resta de la seqüència
Genewise compara les dues seqüències i tradueix la seqüència genòmica tenint en compta els introns i les diferents pautes de lectura. Amb aquest programa vam obtenir per una banda un aliniament de la seqüència genòmica amb la proteïna indicant la localització dels introns i per l'altra la seqüència proteïca predita.
A partir d'aquest punt vam analitzar per separat ambdues seqüències.
6. Anàlisi de la seqüència proteica
El programa T-Coffee fa aliniaments múltiples entre seqüències, tant genòmiques com proteïques.
En aquest cas ens interessava comparar la seqüència de la proteïna predita amb Genewise amb les seqüències de les selenoproteïnes homòloques conegudes. El resultat ens va permetre determinar el nivell d'homologia entre la proteïna predita i les conegudes, important ja que si aquesta és elevada reforça la idea de que es tracta d'un ortòleg.
Un pas posterior va incloure l'estudi dels diferents dominis presents a la proteïna predita anb Interpro, per tal de determinar si presentaven dominis característics de selenoproteïnes conegudes o elements de la seva maquinària de síntesi.
7. Anàlisi de la seqüència genòmica
Una selenoproteïna es caracteritza per tenir:
- Element SECIS
- Codó TGA a la pauta de lectura
Per tant, la confirmació de que la seqüéncia obtinguda és una selenoproteïna passa per buscar en primer lloc un element SECIS en posició 3'. Per fer-ho utilitzem el programa SECIsearch, el qual prediu elements SECIS en base a la seva estructura tridimensional característica.
El codó TGA codificant per selenocisteïna es pot buscar manualment o per mitjà d'un programa Perl, nosaltres vam triar la primera opció.
La presència d'aquests dos elements, juntament amb un bon aliniament i una bona estructura exònica ens van indicar que la proteïnes trobades eren selenoproteïnes.
Les seqüències de les selenoproteïnes conegudes s'obtenen de la base de dades SelenoDB, descarregant-les individualment en format FASTA.
Donat que a priori no coneixíem quines selenoprote&ines eren presents al genoma del nostre organisme, vam cercar les seqüències de totes les selenoproteïnes contingudes en aquesta base de dades.
En primer lloc, cal remarcar que en tot moment hem treballat amb dades en format FASTA.
Donada la facilitat d'utilizació del GeneDB, vam utilitzar aquesta base de dades per fer un garbellament inicial que permetés tenir una primera visió del selenoproteoma de Leishmania i poder descartar aquelles selenoproteïnes que clarament no donessin resultats significatius.
Per fer aixó cal blastejar les seqüències de les selenoproteïnes obtingudes de SelenoDB contra el genoma complet de Leishmania major . La opció que vam escollir és TBLASTN,que alinea DNA contra proteïnes tal i com s'explica al quadre. El criteri de selecció per a considerar un alineament com a significatiu va ser un E-value inferior a 10-10.
Així vam obtenir un conjunt de possibles selenoproteïnes de Leishmania major , les quals vam passar a analitzar més detalladament.
Vam utilitzar el genoma aportat pel professorat al directori d'estudiants per tal de facilitar la manipulació de les dades. Tots els passos mencionats a continuació van ser realitzats a la finestra terminal. Els passos seguits van ser:
- 1.Formatejar la base de dades amb la comanda
formatdb -i genome.fa -p F
on genome.fa és la base de dades que conté el genoma de Leishmania.
- 2.Fer la cerca de similaritat amb TBLASTN
blastall -p TBLASTN -i (query) -d genome.fa | more
on la query és la proteïna a blastejar. Amb aquest pas aliniem les proteïnes amb el genoma. La opció more permet observar els aliniaments obtinguts un a un a la finestra terminal per fer una primera avaluació de la seva qualitat.
- 3.Selecció dels aliniaments més significatius
blastall -p tblastn -i hsap.fa -d dmel_genomic.fa -m 9
Amb la opció -m 9 podem veure els aliniaments ordenats i tabulats mostrant informació referent a cadascun.
Així vam poder veure els aliniaments ordenats segons el seu E-value i descartarem tots aquells que tenien un E-value superior a 10-10. Dels restants, vam triar aquell que tenia l' E-value més petit.
4. Obtenció de la seqüència genòmica
La comanda anterior també aporta informació referent a la regió cromosòmica on es troba cadascun dels hits obtinguts. Una vegada triat l'aliniament òptim assumim que les coordenades d'aquest hit corresponen a la seqüència dins el genoma de Leishmania de la selenoproteïna que s'està estudiant.
Per poder seguir treballant, vam tallar aquesta seqüència mitjançant la comanda:
fastasubseq -f genome.fa -s (inici) -l (llargada) > subseq.fa
on subseq.fa és l'arxiu on redireccionem el fragment tallat.
Si la seqüència a tallar es troba en sentit revers són necessaris una sèrie de passos per procedir a tallar.
- Fer un índex del genoma en format multifasta amb la comanda fastaindex genome.fa genome.index
- Extreure la seqüència amb fastafetch genome.fa genome.index (fragmenta a tallar) > (arxiu on es redireccionarà)
- Extreure el fragment d'interès fastasubseq -f (arxiu redireccionat) -s (inici) -l (llargada) > (nom d'arxiu per redireccionar)
- Canviar el sentit el fragment extret amb la comanda fastarevcomp (nom de l'arxiu) > (no d'arxiu per redireccionar)
Cal fixar una llargada superior a la que indica el resultat de l'aliniament per tal d'incloure l'element SECIS a la seqüéncia.
5. Predicció de l'estructura exònica i la seqüència proteica
Un cop vam tenir en un fitxer la seqüència genòmica, el que volíem era predir, per una part, l'estructura exònica del gen i, per l'altra, la seqüéncia de la proteïna resultant.
El programa que vam utilitzar per aquests dos objectius va ser Genewise. Per fer-lo servir cal introduir:
- Seqüéncia proteica de SelenoDB
- Seqüéncia genòmica extreta de l'aliniament
Vam marcar les opcions translation, cDNA i GFF output. L'aliniament va ser global, ja que així evitàvem que regions de baixa homologia afectássin un aliniament bo per la resta de la seqüència
Genewise compara les dues seqüències i tradueix la seqüència genòmica tenint en compta els introns i les diferents pautes de lectura. Amb aquest programa vam obtenir per una banda un aliniament de la seqüència genòmica amb la proteïna indicant la localització dels introns i per l'altra la seqüència proteïca predita.
A partir d'aquest punt vam analitzar per separat ambdues seqüències.
6. Anàlisi de la seqüència proteica
El programa T-Coffee fa aliniaments múltiples entre seqüències, tant genòmiques com proteïques.
En aquest cas ens interessava comparar la seqüència de la proteïna predita amb Genewise amb les seqüències de les selenoproteïnes homòloques conegudes. El resultat ens va permetre determinar el nivell d'homologia entre la proteïna predita i les conegudes, important ja que si aquesta és elevada reforça la idea de que es tracta d'un ortòleg.
Un pas posterior va incloure l'estudi dels diferents dominis presents a la proteïna predita anb Interpro, per tal de determinar si presentaven dominis característics de selenoproteïnes conegudes o elements de la seva maquinària de síntesi.
7. Anàlisi de la seqüència genòmica
Una selenoproteïna es caracteritza per tenir:
- Element SECIS
- Codó TGA a la pauta de lectura
Per tant, la confirmació de que la seqüéncia obtinguda és una selenoproteïna passa per buscar en primer lloc un element SECIS en posició 3'. Per fer-ho utilitzem el programa SECIsearch, el qual prediu elements SECIS en base a la seva estructura tridimensional característica.
El codó TGA codificant per selenocisteïna es pot buscar manualment o per mitjà d'un programa Perl, nosaltres vam triar la primera opció.
La presència d'aquests dos elements, juntament amb un bon aliniament i una bona estructura exònica ens van indicar que la proteïnes trobades eren selenoproteïnes.
La comanda anterior també aporta informació referent a la regió cromosòmica on es troba cadascun dels hits obtinguts. Una vegada triat l'aliniament òptim assumim que les coordenades d'aquest hit corresponen a la seqüència dins el genoma de Leishmania de la selenoproteïna que s'està estudiant.
Per poder seguir treballant, vam tallar aquesta seqüència mitjançant la comanda:
fastasubseq -f genome.fa -s (inici) -l (llargada) > subseq.fa
on subseq.fa és l'arxiu on redireccionem el fragment tallat.
Si la seqüència a tallar es troba en sentit revers són necessaris una sèrie de passos per procedir a tallar.
Cal fixar una llargada superior a la que indica el resultat de l'aliniament per tal d'incloure l'element SECIS a la seqüéncia.
Un cop vam tenir en un fitxer la seqüència genòmica, el que volíem era predir, per una part, l'estructura exònica del gen i, per l'altra, la seqüéncia de la proteïna resultant.
El programa que vam utilitzar per aquests dos objectius va ser Genewise. Per fer-lo servir cal introduir:
Vam marcar les opcions translation, cDNA i GFF output. L'aliniament va ser global, ja que així evitàvem que regions de baixa homologia afectássin un aliniament bo per la resta de la seqüència
Genewise compara les dues seqüències i tradueix la seqüència genòmica tenint en compta els introns i les diferents pautes de lectura. Amb aquest programa vam obtenir per una banda un aliniament de la seqüència genòmica amb la proteïna indicant la localització dels introns i per l'altra la seqüència proteïca predita.
A partir d'aquest punt vam analitzar per separat ambdues seqüències.
El programa T-Coffee fa aliniaments múltiples entre seqüències, tant genòmiques com proteïques. En aquest cas ens interessava comparar la seqüència de la proteïna predita amb Genewise amb les seqüències de les selenoproteïnes homòloques conegudes. El resultat ens va permetre determinar el nivell d'homologia entre la proteïna predita i les conegudes, important ja que si aquesta és elevada reforça la idea de que es tracta d'un ortòleg.
Un pas posterior va incloure l'estudi dels diferents dominis presents a la proteïna predita anb Interpro, per tal de determinar si presentaven dominis característics de selenoproteïnes conegudes o elements de la seva maquinària de síntesi.
Una selenoproteïna es caracteritza per tenir:
- Element SECIS
- Codó TGA a la pauta de lectura