2.1 Introducció2.3 Conservació en altres espècies
2.4 Caracterització de l'expressió del gen
Sandra Roca Aumatell (sandra.roca01@campus.upf.edu)
Cira Rubies Espinalt (cira.rubies01@campus.upf.edu)
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
1. Resum
2.1 Introducció2.3 Conservació en altres espècies
2.4 Caracterització de l'expressió del gen
En aquest treball presentem la caracterització de l'estructura exònica, homologia, funció i expressió del gen humà FIP1L1-PDGFRA. Aquest gen codifica per una proteïna de fusió que es dóna per una delecció que uneix FIP1L1 (factor involucrat en el processament del mRNA) amb l'exó 12 de PDGFRA (receptor alfa de factors de creixement derivats de plaquetes) en el cromosoma 4q12. S'ha vist que el gen FIP1L1-PDGFRA codifica per una tirosina quinasa que transforma les cèl·lules hematopoiètiques donant lloc a una malaltia coneguda com a síndrome hipereosinofílic . Aquest síndrome es tracta d'un desordre poc freqüent que es caracteritza per una sobreproducció d'eosinòfils en la medul·la òssia, eosinofília, infiltració tissular i dany en òrgans.
L'estudi S'ha fet paral·lelament pels dos gens que la codifiquen ja que, no hi havia informació detallada de l'estructura genòmica de la proteïna de fusió en les bases de dades consultades: NCBI3, Ensembl2 i UCSC1.
De manera que s'ha fet un estudi detallat dels dos gens codificants, basada sobretot en la base de dades de USCS, ja que la informació d'estructura genòmica d'Ensembl era insuficient.
![]() FIP1L1
FIP1L1
El gen FIP1L1 codifica per un factor de processament del mRNA que es troba al nucli. Aquest factor forma part d'un complex anomenat CPSF (cleavage and polyadenylation specificity factor) que està format per CPFS1, CPSF2, CPSF3, CPSF4 i FIP1L1. CPSF té un paper clau en la formació de l'extrem 3' del pre-mRNA, ja que reconeix la senyal AAUAAA i interacciona amb la polimerasa A i altres factors, per tal de poder tallar i afegir la cua poli(A). FIP1L1 contribueix al reconeixement de la senyal AAUAAA i ajuda a l'adicció de la cua de poli(A) unint-se a elements rics en U al voltant del lloc de poliadenització.
![]() PDGFRA
PDGFRA
Aquest gen codifica per un receptor tirosina-quinasa de superfície cel·lular que uneix membres de la família de factors de creixement derivats de plaquetes (PDGFA i PDGFB). Aquests factors de creixement són mitògens per cèl·lules d'origen mesenquimal.
El receptor pot formar homodímers o heterodímers amb PDGFRB.
índex
Després de fer un BLASTP (proteïna-proteïna) amb l'NCBI3 de la nostra seqüència aminoacídica inicial contra tot el genoma humà,
es va poder determinar que la seqüència proteica corresponia a la proteïna de fusió FIP1L1-PDGFRA, ja que era la que presentava un major score, concretament de 1592, i un E-value (probabilitat d'error) de 0 (veure resultat ).
Per tal d'esbrinar quina part de la proteïna de fusió corresponia a cada un dels gens (FIP1L1 o PDGFRA) es va fer un BLAT a nivell del genoma humà, a partir del USCS1, els resultats del qual són al següent enllaç . En el resultat del BLAT podem veure que les dues primeres proteïnes de la llista corresponen a les de la nostra proteïna de fusió. Així doncs, en primer lloc i amb un score de 1518 i una identitat del 100%, trobem la proteïna PDGFRA, i en segon lloc i amb un score de 967 i una identitat del 99.9% trobem la proteïna FIP1L1.
La proteïna de fusió FIP1L1-PDGFRA té un total de 879 aminoàcids: els primers 338 corresponen a la proteïna FIP1L1, i els 511 aminoàcids restants corresponen a la PDGFRA.
La taula següent mostra la seqüència de proteïna dels trànscrits que codifiquen per la proteïna de fusió.
| FIP1L1 : trànscrit 1 (1089aa) | PDGFRA : trànscrit 1 (594aa) |
MSAGEVERLVSELSGGTGGDEEEEWLYGGPWDVHVHSDLAKDLDENEVERPEEENASANP PSGIEDETAENGVPKPKVTETEDDSDSDSDDDEDDVHVTIGDIKTGAPQYGSYGTAPVNL NIKTGGRVYGTTGTKVKGVDLDAPGSINGVPLLEVDLDSFEDKPWRKPGADLSDYFNYGF NEDTWKAYCEKQKRIRMGLEVIPVTSTTNKITAEDCTMEVTPGAEIQDGRFNLFKVQQGR TGNSEKETALPSTKAEFTSPPSLFKTGLPPSRNSTSSQSQTSTASRKANSSVGKWQDRYG RAESPDLRRLPGAIDVIGQTITISRVEGRRRANENSNIQVLSERSATEVDNNFSKPPPFF PPGAPPTHLPPPPFLPPPPTVSTAPPLIPPPGFPPPPGAPPPSLIPTIESGHSSGYDSRS ARAFPYGNVAFPHLPGSAPSWPSLVDTSKQWDYYARREKDRDRERDRDRERDRDRDRERE RTRERERERDHSPTPSVFNSDEERYRYREYAERGYERHRASREKEERHRERRHREKEETR HKSSRSNSRRRHESEEGDSHRRHKHKKSKRSKEGKEAGSEPAPEQESTEATPAE |
MGTSHPAFLVLGCLLTGLSLILCQLSLPSILPNENEKVVQLNSSFSLRCFGESEVSWQYP MSEEESSDVEIRNEENNSGLFVTVLEVSSASAAHTGLYTCYYNHTQTEENELEGRHIYIY VPDPDVAFVPLGMTDYLVIVEDDDSAIIPCRTTDPETPVTLHNSEGVVPASYDSRQGFNG TFTVGPYICEATVKGKKFQTIPFNVYALKATSELDLEMEALKTVYKSGETIVVTCAVFNN EVVDLQWTYPGEVKGKGITMLEEIKVPSIKLVYTLTVPEATVKDSGDYECAARQATREVK EMKKVTISVHEKGFIEIKPTFSQLEAVNLHEVKHFVVEVRAYPPPRISWLKNNLTLIENL TEITTDVEKIQEIRYRSKLKLIRAKEEDSGHYTIVAQNEDAVKSYTFELLTQVPSSILDL VDDHHGSTGGQTVRCTAEGTPLPDIEWMICKDIKKCNNETSWTILANNVSNIITEIHSRD RSTVEGRVTFAKVEETIAVRCLAKNLLGAENRELKLVAPTLRSELTVAAAVLVLLVIVII SLIVLVVIWKQKPRYEIRWRVIESISPDGHEYIYVDPMQLPYDSRWEFPRDGLVLGRVLG SGAFGKVVEGTAYGLSRSQPVMKVAVKMLKPTARSSEKQALMSELKIMTHLGPHLNIVNL LGACTKSGPIYIITEYCFYGDLVNYLHKNRDSFLSHHPEKPKKELDIFGLNPADESTRSY VILSFENNGDYMDMKQADTTQYVPMLERKEVSKYSDIQRSLYDRPASYKKKSMLDSEVKN LLSDDNSEGLTLLDLLSFTYQVARGMEFLASKNCVHRDLAARNVLLAQGKIVKICDFGLA RDIMHDSNYVSKGSTFLPVKWMAPESIFDNLYTTLSDVWSYGILLWEIFSLGGTPYPGMM VDSTFYNKIKSGYRMAKPDHATSEVYEIMVKCWNSEPEKRPSFYHLSEIVENLLPGQYKK SYEKIHLDFLKSDHPAVARMRVDSDNAYIGVTYKNEEDKLKDWEGGLDEQRLSADSGYII PLPDIDPVPEEEDLGKRNRHSSQTSEESAIETGSSSSTFIKREDETIEDIDMMDDIGIDS SDLVEDSFL |
![]() FIP1L1
FIP1L1
<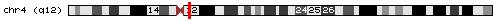 Figura 1. Localització cromosòmica del gen. Figura 1. Localització cromosòmica del gen. |
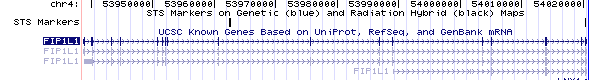 Figura 2. Estructura genòmica dels trànscrits del gen FIP1L1. Figura 2. Estructura genòmica dels trànscrits del gen FIP1L1. |
- Aquest gen es troba en el cromosoma 4 en la posició 53.938.620-54.020.597.
- Trobem 4 transcrits associats a aquest gen que ens donen quatre isoformes possibles.
| trànscrit 1 | trànscrit 2 | trànscrit 3 | trànscrit 4 | |
| posició | chr4:53938620-54020597 | chr4:53938620-54020540 | chr4:53938727-54020540 | chr4:53988990-54021785 |
| cadena | + | + | + | + |
| tamany genòmic | 81.978 | 81.921 | 81.814 | 32.796 |
| tamany mRNA | 2.154 | 1.914 | 3.201 | 2.173 |
| exons | 18 | 15 | 15 | 7 |
| CDS exons | 18 | 15 | 15 | 4 |
| 5' UTR | 143 | 143 | 1328 | 217 |
| 3' UTR | 224 | 167 | 167 | 1333 |
| tamany proteïna | 594 | 117 | 117 | 208 |
L'estudi de l'splicing alternatiu i la pauta de lectura es troba en el següent enllaç
Si comparem les diferents isoformes respecte la isoforma 1 que és el producte del trànscrit que forma part de la proteïna de fusió podem observar:
- Isoforma 2: L'exó 2, 9 i 11 de la isoforma 1 no es troben en aquesta isoforma degut a un fenomen d'splicing alternatiu. A més, podem dir que hi ha canvi de pauta de lectura ja que, el frame no es manté en els exons posteriors.- Isoforma 3: L'exó 1, 2 i 9 de la isoforma 1 no es troben en aquesta isoforma degut a un fenomen d'splicing alternatiu. A més, podem dir que no hi ha canvi de la pauta de lectura perquè el frame es manté en els exons posteriors.
- Isoforma 4: Els exons codificants per aquesta isoforma corresponen als 4 últims exons de la isoforma 1. Degut a splicing alternatiu, s'han incorporat 3 exons no codificants a l'inici del trànscrit que han provocat un canvi de pauta de lectura.
![]() PDGFRA
PDGFRA
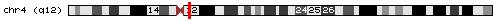 Figura 3. Localització cromosòmica del gen. Figura 3. Localització cromosòmica del gen. |
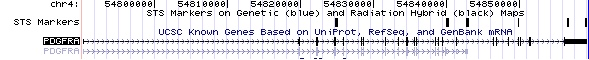 Figura 4. Estructura genòmica dels trànscrits del gen PDGFRA. Figura 4. Estructura genòmica dels trànscrits del gen PDGFRA. |
- Aquest gen es troba en cromosoma 4 en la posició 54.790.204-54.859,168.
- Trobem 2 transcrits associats a aquest gen que ens donen dues isoformes possibles.
| trànscrit 1 | trànscrit 2 | |
| posició | chr4:54790204-54859168 | chr4:54790235-54842902 |
| cadena | + | + |
| tamany genòmic | 68.965 | 52.668 |
| tamany mRNA | 6.405 | 3.032 |
| exons | 23 | 18 |
| CDS exons | 22 | 15 |
| 5' UTR | 148 | 117 |
| 3' UTR | 2.972 | 642 |
| tamany proteïna | 1.089 | 743 |
Taula 3. Estructura genòmica dels 2 transcrits de PDGFRA.
Si comparem la isoforma 2 respecte la isoforma 1 que és el producte del trànscrit que forma part de la proteïna de fusió podem observar:
- L'exó 1, 16 i 18 de la isoforma 2 no es troben en la isoforma 1 degut a un fenomen d'splicing alternatiu, tot i això no afecten a la pauta de lectura perquè són exons no codificants.
![]() FIP1L1
FIP1L1
En la següent taula podem observar la homologia d'aquest gen en altres espècies proporcionada per Ensembl2. Únicament hem tingut en compte un percentatge d'identitat (% ID) superior a 60, ja que creiem que un % ID inferior no és un resultat significatiu i podria no tractar-se d'una proteïna ortòloga.
| Imatge | Espècie | % ID |
 |
Pan troglodytes | 100 |
 |
Macaca mulatta | 99 |
 |
Bos taurus | 98 |
 |
Mus musculus | 95 |
 |
Rattus norvegicus | 95 |
 |
Monodelphis domestica | 90 |
 |
Oryctolagus cuniculus | 86 |
 |
Loxodonta africana | 85 |
 |
Cavia porcellus | 84 |
 |
Ornithorhynchus anatinus | 81 |
 |
Gallus gallus | 79 |
 |
Echinops telfairi | 77 |
 |
Erinaceus europaeus | 74 |
 |
Dasypus novemcinctus | 70 |
 |
Xenopus tropicalis | 69 |
Taula 4. Espècies homòlogues per FIP1L1.
En el següent enllaç hi trobem l'arbre filogenètic de FIP1L1 per les espècies anteriors obtinguts a partir del ClustalW8.
![]() PDGFRA
PDGFRA
En la següent taula podem observar la homologia d'aquest gen en altres espècies que s'ha obtingut fent un BlastP a NCBI. No hem utilitzat les dades d'homologia d'Ensembl degut a que van ser retirades de la web segurament degut algun error.
Cal tenir en compte que els valors d'homologia es troben sobrevalorats perquè s'ha utilitzat la seqüència de la proteïna enlloc de la seqüència genòmica, ja que com sabem el codi genètic és degenerat. Tots els organismes presenten un E-value de 0.
| Imatge | Espècie | % ID |
 |
Macaca fascicularis | 99 |
 |
Canis familiaris | 97 |
 |
Bos taurus | 95 |
 |
Mus musculus | 94 |
 |
Rattus norvegicus | 94 |
 |
Monodelphis domestica | 88 |
 |
Gallus gallus | 85 |
 |
Xenopus laevis | 81 |
 |
Takifugu rubripes | 70 |
 |
Danio rerio | 69 |
Taula 4. Espècies homòlogues per PDGFRA.
En el següent enllaç hi trobem l'arbre filogenètic de PDGFRA per les espècies anteriors obtinguts a partir del ClustalW8.
índex
L'estudi de l'expressió dels gens s'ha realitzat a partir de microarrays obtingudes de la base de dades del USCS1.
- De color vermell observem una alta expressió del gen.- De color verd observem una baixa expressió del gen.
![]() FIP1L1
FIP1L1
GNF Expression Atlas 2 Data from U133A and GNF1H Chips.

Figura 5. Microarray FIP1L1.
En el resultat del microarray s'observa l'expressió de FIP1L1 en molts tipus cel·lulars, sobretot cèl·lules sanguínies i del sistema immunitari, així com en alguns tipus de càncers relacionats amb aquests tipus cel·lulars.
En el següent enllaç hi ha més informació referent a l'expressió en teixits sans i cancerígens.
Cal destacar l'expressió de FIP1L1 en teixits sans com el cervell, l'aparell urogenital i sistema nerviós. Per altra banda, descatem l'expressió en teixits cancerígens com la pell, pròstata, medul·la òssia, múscul i esòfag.
![]() PDGFRA
PDGFRA
GNF Expression Atlas 2 Data from U133A and GNF1H Chips.

Figura 6. Microarray PDGFRA.
En el resultat del microarray s'observa l'expressió de PDGFRA en diferents teixits i òrgans com en el múscul llis, miòcits cardíacs, teixit adipós, ovari i pulmó fetal.
En el següent enllaç hi ha més informació referent a l'expressió en teixits sans i cancerígens.
Cal destacar l'expressió de PDGFRA en teixits i òrgans sans com el teixit adipós, el cartílag, l'úter i els pulmons. Per altra banda, descatem l'expressió en teixits cancerígens com el teixit sinovial, cartílag i fetge.
índex
Per a la caracterització de les regions promotores s'han utilitzat dos mètodes.
- Servidor web del programa PROMO6- Disseny d'un programa en Perl
1. PROMO
Per poder utilitzar el programa PROMO6, prèviament s'han obtingut les seqüències promotores dels factors de transcripció de la seqüència upstream (1000) i downstream (100) d'ambdós gens a partir del USCS.
Els factors de transcripció humans obtinguts amb una dissimilaritat inferior al 10% es mostren a continuació:
Degut a la gran quantitat de factors de transcripció possibles, hem fet una selecció. La selecció ha consistit en agafar només aquells factors amb RE equally inferior a 0.09% (la probabilitat de que es doni per atzar és inferior al 0.09%) i amb una dissimilaritat inferior a 4. Com que la dissimilaritat és molt baixa, podem estar segurs que aquests factors de transcripció són específics per la seqüència. Al augmentar l'especificitat estem rebutjant altres factors de transcripció reals. Els resultats es mostren a continuació:
2. PROGRAMA PERL
S'ha desenvolupat un programa en Perl al qual al donar-li un fitxer amb matrius de factors de transcripció (FTs) i un altre fitxer amb la seqüència de la regió promotora en format FASTA dels dos gens, et dóna una llista dels FTs proporcionats amb un p-value que indica la probabilitat de rebutjar erròniament la hipòtesi inicial de que el factor no s'uneix.
- El programa- El fitxer amb la seqüència promotora FIP1L1 i de PDGFRA
- El fitxer amb les matrius de FTs
Els resultats obtinguts del programa es presenten en la taula següent:
| FTs | p-value (FIP1L1) | p-value (PDGFRA) |
| AP-1 | 0.08 | 1 |
| AR | 0.92 | 1 |
| cMyc | 1 | 1 |
| NF-AT1 | 0.75 | 0.51 |
| NF-KappaB | 1 | 1 |
| SRY | 1 | 1 |
| YY1 | 0.72 | 0.69 |
| RXR-alpha | 0.5 | 0.74 |
| HIF-1 | 1 | 1 |
| AhR | 1 | 1 |
| PU.1 | 0.27 | 0.42 |
| HNF4 | 1 | 0.17 |
| NRSF | 1 | 1 |
Taula 5. Factors de transcripció obtinguts del programa.
Si comparem els dos estudis realitzats per identificar possibles factors de transcripció, podem determinar que el factor de transcripció AP-1 per FIP1L1 amb un p-value de 0.08 és l'únic factor de transcripció obtingut pel programa que podem donar com a significatiu. Aquest s'uneix en les posicions 1016-1024. Si ho comparem amb els resultats del PROMO6, aquest factor l'havíem escollit com a vàlid. Aquest factor reconeix la seqüència AATGAGTCA i s'uneix a les posicions 1014-1022 amb una dissimilaritat de 0.35 i RE equally de 0.05. La no concordànça de les posicions és deguda a que les matrius utilitzades pel programa són de 7 caràcters i les del PROMO6 són de 9 caràcters.
Fent referència als factors de transcripció per PDGFRA, no se n'ha trobat cap de significatiu (factors amb un p-value inferior a 0.1) amb el programa. Cal afegir que els factors no significatius obtinguts amb el programa tampoc ho són en el PROMO6.
Podem dir que el reduït nombre de factors de transcripció utilitzats pel programa no és suficient per a donar-nos factors específics per la nostra seqüència.
índex
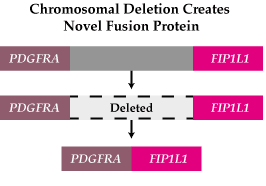
S'ha vist que una delecció intersticial del cromosoma 4q12 provoca la fusió del gen de FIP1L1 amb l'exó 12 del gen PDGFRA. La fusió d'aquests gens és la causa del síndrome hipereosinofílic . S'ha vist que el gen FIP1L1-PDGFRA codifica per una tirosina quinasa activada constitutivament que transforma les cèl·lules hematopoiètiques.
En un estudi8 recent s'ha demostrat que FIP1L1 és completament indispensable per a l'activació de PDGFRA in vitro i in vivo. Per altra banda, el trencament de dos residus conservats de triptòfan en el domini juxtamembrana (JM) és necessari per l'activació i transformació del potencial de FIP1L1-PDGFRA. La presència del domini JM complet en la proteïna de fusió FIP1L1-PDGFRA té una acció inhibitòria de la proteïna. Aquests resultats demostren que el trencament del domini JM evita aquest efecte autoinhibitori i per tant, dóna lloc a un mecanisme que permet l'activació constitucional de l'activitat tirosina quinasa, fet que indueix a la malaltia.
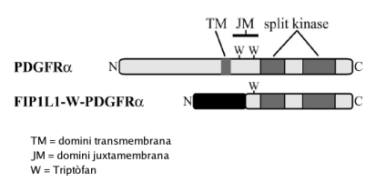
Figura 7. Formació de la proteïna de fusió.
Com ja s'ha dit, FIP1L1-PDGFRA és una proteïna de fusió que s'ha identificat en pacients que pateixen el síndrome hipereosinofílic. FIP1L1 es fusiona amb PDGFRA degut a una delecció intersticial (concretament de 800 Kb) del cromosoma 4q12. El producte d'aquesta fusió dóna lloc a una tirosina quinasa activa constitutivament que permet el creixement independent de factors de creixement en cèl·lules hematopoiètiques i que és sensible a la inhibició per imatinib (fàrmac quimioteràpic). FIP1L1 és una proteïna involucrada en la poliadenilació, però es desconeix perquè proporciona propietats de dimerització en la proteïna de fusió. Per altra banda, s'ha identificat en estudis clínics amb pacients amb síndrome d'hipereosinofília un truncament del domini autoinhibitori de juxtamembrana (JM) de PDGFRA. Tot i que els llocs de truncament de FIP1L1 de la proteïna de fusió tenen una alta variabilitat distribuïda al llarg dels introns 7-13, s'ha vist que en tots els casos el lloc de truncament de PDGFRA està localitzat concretament en l'exó 12, el qual conté el domini JM (7-13). Per tant, podem dir que la predominància del truncament dels dominis JM en FIP1L1-PDGFRA juga un paper significant en l'activació de PDGFRA.
El síndrome d'hipereosinofília és un desordre poc freqüent que es caracteritza per una sobreproducció d'eosinòfils en la mèdul·la òssia (més de 1.500 eosinòfils/mm3), eosinofília, infiltracció tissular i dany als òrgans. El síndrome és més comú en homes que en dones (9:1) i apareix predominantment entre els 20 i els 50 anys.
S'han descrit diferents síndromes segons el compromís orgànic: síndrome de Löffler (afecció pulmonar aïllada), síndrome cardíac (endocarditis i hipereosinofília), malaltia eosinofílica disseminada del col·làgen i leucèmia eosinofílica.
El compromís cutani habitualment consisteix en angioedema i lesions urticàries, eritema i nòduls. El compromís cardíac inclou tres etapes. La primera d'elles, necròtica, seguida per una fase trombòtica i, per últim, l'estadi final, amb fibrosi endomiocàridca i dany de les vàlvules auriculoventriculars que culminen amb una insuficiència cardíaca congestiva. Els sistemes nerviosos central i perifèric poden estar afectats. Existeix una gran heterogeneïtat clínica i el pronòstic és molt variable, des de l'evolució asimptomàtica fins a una malaltia fatal.
- En primer lloc quan vam disposar de la seqüència de la nostra proteïna de fusió, vam realitzar un BlastP (proteïna-proteïna) de la seqüència contra tot el genoma humà, a partir de la base de dades NCBI3, per tal de confirmar que la seqüència corresponia realment a la proteïna de fusió. A continuació es va fer un Blat de la seqüència genòmica amb l'USCS1 per determinar quina part de la proteïna de fusió codificava per cada un dels gens. Finalment, tota la informació de l'estructura genòmica (en quin cromosoma es troba, quants exons són codificants i quants són no-codificants...) dels dos gens es va extreure del genome browser del USCS1.
- Per l'estudi de la conservació entre espècies diferents es van obtenir les dades de cada proteïna de fonts diferents. Per a la proteïna FIP1L1 es van extreure les dades d'homologia a partir de l'Ensembl2, i es va fer una selecció únicament d'aquelles espècies amb un percentatge d'homologia superior a un 60%. Per altra banda, amb la proteïna PDGFRA, al no disposar de les dades al Ensembl2, es va realizar un BlastP a l'NCBI3 de la proteïna contra tots els organismes. Així es va poder obtenir un llistat d'espècies amb alta homologia, agafant com a significatius aquells resultats amb un E-value igual a zero. També es va realitzar per a cada proteïna un arbre filogenètic amb les espècies amb alta homologia a través del ClustalW8, per tal de veure les relacions evolutives.
- Per a la caracterització de l'expressió del gen es van analitzar els microarrays (GNF Expression Atlas 2) que proporcionava el genome browser del USCS1 per a les dues proteïnes per veure en quins teixits, o tipus cel·lulars es trobava una alta expressió del gen. A més a més també es va extreure informació a partir d'un link de l'USCS (Gepis Tissue) on trobàvem les diferències d'expressió entre teixits sans i teixis cancerígens de cada gen.
- Per obtenir en format FASTA la seqüència promotora formada per 1kb upstream del lloc de començament de la transcripció (transcription start site o TSS) i 100bp downstream del TSS es va utlitizar l'USCS1. A partir d'aquesta seqüència es va treballar amb el servidor web pel programa PROMO, per tal d'obtenir un conjunt de factors de transcripció humans amb una dissimilaritat inferior al 10%, que es podien unir al promotor de cada un dels gens. Un cop obtinguts els factors es va fer una selecció basant-los en l'RE equally(inferior a 0,09%) i la dissimilaritat (inferior a 4) per assegurar-nos que aquells seleccionats veritablement es podien unir amb una alta probabilitat a la regió promotora. En paral·lel es va realitzar un programa en Perl amb la mateixa finalitat, es a dir, que a partir d'un fitxer amb matrius de factors de transcripció (FTs) i un altre fitxer amb una seqüència d'una regió promotora en format FASTA, el programa mostra la llista dels FTs proporcionats amb un p-value per cada FT que indica la probabilitat de rebutjar erròniament la hipòtesi inicial de que el factor no s'uneix.
- En l'últim punt del treball es va estudiar la funció del gen per a la proteïna de fusió basant-nos principalment en articles obtinguts a partir del Pubmed (NCBI3) ja que les altres fonts, per exemple la base de dades de Gen Ontology7 no ens proporcionaven suficient informació. A més, també es va obtenir informació de l'USCS1.
índex
- A partir de la proteïna 9 proporcionada s'ha pogut comprovar que es tractava de la proteïna de fusió FIP1L1-PDGFRA.
- Aquesta proteïna es forma a partir d'una delecció en el cromosoma 4 que uneix una part del gen que codifica per FIP1L1 i una altra part del gen PDGFRA que forma la part final.
- Independentment, la funció del gen FIP1L1 està involucrada en el processament del mRNA mentre que PDGFRA codifica per un receptor tirosina quinasa que uneix factors de creixement derivats de plaquetes.
- La fusió dels dos gens dóna com a resultat una malaltia poc freqüent coneguda com a síndrome hipereosinofílic.
- L'estudi s'ha fet dels dos gens per separat ja que la informació de la proteïna de fusió és bàsicament a nivell mèdic i funcional.
- Per tant, s'ha fet un estudi de l'estructura genòmica que ha permès identificar els transcrits de cada gen que formen la proteïna de fusió, la unió entre ambdós i la part codificant.
- Per altra banda, l'estudi de la conservació del gen en altres espècies ha permès esbrinar que ambdós gens es troben molt conservats entre mamífers fet que indica que són d'especial importància.
- Cal dir que també s'ha pogut caracteritzar la seva expressió en teixits i òrgans, i que aquesta ha permès donar una visió més àmplia de la seva involucració a nivell cel·lular.
- En relació a la identificació de possibles factors de transcripció s'ha treballat a partir de dos mètodes molt diferents però que han acabat donant uns resultats molt semblants: per una banda, la realització del programa ha estat una tasca complicada però a la vegada molt útil per permetre fer prediccions de manera molt ràpida i precisa, mentre que el fet de treballar amb un programa com el PROMO, ha permès treballar d'una manera molt més simple i fàcil però amb una gran tasca ha desenvolupar alhora de fer la selecció dels factors de transcripció, degut al gran volum de resultats obtinguts.
- Per acabar podem dir que s'ha fet un estudi bastant complet de la proteïna de fusió i dels dos gens que la formen, a la vegada que s'ha desenvolupat una tasca didàctica i d'adquisió de coneixements de noves eines de recerca com han estat la utilització de les bases de dades (Ensemb, USCS i NCBI).
AGRAÏMENTS
En primer lloc agraïr enormement a les dues persones que han supervisat les pràctiques (Haagen i Charles) que ens han ajudat molt, tan per la seva claredat alhora d'explicar-se com per la gran simpatia que ha fet que no desesperéssim en els moments més difícils. Gràcies per la paciència i les hores dedicades.
En segon lloc voldríem agraïr el gran companyarisme que hi ha hagut entre els companys de classe, l'intercanvi d'idees i les solucions als problemes més obvis. També agraïr la comprensió de tota aquella gent que sempre ens rodeja i que en el transcurs del treball han hagut de suportar el nostre mal humor. Gràcies a la família, amics i companys de pis.
Sobretot, moltes gràcies Mireia, Clàudia, Anna, Cris i a tots els altres per les estones de desconnexió fent el cafè i el vici.
I ja per acabar, gràcies a la paciència que hem tingut les dues, les rialles i els moments tontos que hem passat. Perquè tantes estones juntes sempre serveixen per adonar-te del gran potencial de la persona que tens el costat.
Gràcies a tots!!

Figura 8. Autores del treball.