Maria Segura (maria.segura02@campus.upf.edu) i Gemma Soldevila (gemma.soldevila01@campus.upf.edu)
El gen de fusió AME es forma com a resultat de la translocació t(3;21)(q26;q22). En aquest procés, el gen AML1 es fusiona amb els gens MDS1-EVI1, sense trencar la pauta de lectura (in-frame fusion). El gen AML1 (Acute Myeloid Leukemia 1) és un dels gens amb major freqüència de mutació o translocació en càncers humans. El gen EVI1 (Ecotropic Virus Integration site 1) s'expressa de forma anormal en individus afectats per la Síndrome mielodisplàsica (SMD), Leucèmia mieloide aguda (LMA) o Leucèmia mieloide crònica (LMC). Per últim, del gen MDS1 (Myelodysplasia Syndrome 1) no se'n coneix la funció, tot i que si se sap que la seva localització és immediatament per sobre (upstream) del gen EVI1.
Torna a l'índex
A partir de l'anàlisi computacionalment de les característiques de cada un dels gens que formen la proteïna de fusió final, mitjançant la informació accessible a les bases de dades i de programes específics pel tractament de seqüències, s'ha estudiat l'estructura de cada un dels gens individualment així com la del producte de fusió, amb la finalitat de poder conèixer quines són les regions clau perquè es produeixi la translocació i la unió dels diferents gens. A part, també es descriuen diferents característiques dels gens per tal tenir major informació de les condicions d'expressió de cada un d'ells.
Torna a l'índex
Al costat de la representació de cada proteïna es mostra la seva seqüència, i marcat en color verd, la seqüència codificant del domini Runt.
Torna a l'índex / Torna a l'Estructura genòmica
Cromosoma: 3
Banda citogenètica: 3q.26.2.

Identificació: ENSG00000206115 (Ensembl Gene ID).
Transcrits: existeix un transcrit anomenat MDS1
- Transcrit MDS1:
-
Nom: Myelodysplasia syndrome 1
Identificació: NM_004991 (UCSC Known Genes), NM_004991.1 (RefSeq), ENST00000382851 (Ensembl).
Seqüència consens: -
Posició: chr3:170349964-170864112
Mida genòmica: 514149 nucleòtids Veure seqüència
Cadena: -
Es tracta de l'únic transcrit. Existeixen diferències en el número d'exons en les diferents bases de dades consultades. Segons Ensembl té un únic exó, però segons UCSC en té 3. La informació s'ha extret de UCSC seguint el mètode utilitzat per als altres gens, tot i que també es comenta la procedent de Ensembl. El transcrit està format per 3 exons, on tots són codificants amb una seqüència de mRNA de 901 nucleòtids (BC069498). La seqüència codificant té una llargada de 510 nucleòtids (comptant codó STOP) Veure seqüència. Dóna lloc a una proteïna de 169 aminoàcids segons UCSC (BC069498). Segons la base de dades Ensembl la proteïna obtinguda és de 121 aminoàcids a partir d'un únic exó de 363 aminoàcids.
Cal destacar però, que tot i existir aquesta divergència en exons i proteïna, els aminoàcids d'aquest gen que estan presents en la proteïna de fusió són comuns tant en la proteïna de 169 com en la de 121 aminoàcids.
| Número exó |
Nucleòtid inici exó |
Nucleòtid final exó |
Llargada exó |
Fase |
Nucleòtids codificants |
| 1 |
170863817 |
170864112 |
295 |
- |
37 |
| 2 |
170581668 |
170582006 |
338 |
1 |
338 |
| 3 |
170349963 |
170350237 |
274 |
0 |
132 |
La proteïna resultant de la traducció d'aquest gen no mostra cap domini implicat en cap funció, segons la informació extreta de la base de dades Prosite.
Torna a l'índex / Torna a l'Estructura genòmica
Cromosoma: 3
Banda citogenètica: 3q.26.2.

Identificació: 2122(Entrez GeneID), i ENSG00000085276 (Ensembl Gene ID).
Transcrits: presenta 2 transcrits, anomenats EVI1 i Hypothetical protein DKFZp686J18113 a UCSC que coincideixen amb els transcrits presents a Ensembl, anomenats EVI1_HUMAN i Q9UBK3_HUMAN respectivament.
- Transcrit EVI1:
-
Nom: Ecotropic viral integration site 1
Identificació: NM_005241(UCSC Known Genes), NM_005241.1 (RefSeq), ENST00000264674 (Ensembl).
Seqüència consens: CCDS3205.1 En aquest cas cal destacar que el transcrit es troba en estat provisional a RefSeq.
Posició: chr3:170285244-170346787
Mida genòmica: 61544 nucleòtids Veure seqüència
Cadena: -
Es tracta de la isoforma més llarga. Està formada per 16 exons, 14 dels quals són codificants amb una seqüència de mRNA de 3570 nucleòtids (NM_005241). La seqüència codificant comença a l'exó 3 i acaba al 16 i consta de 3156 nucleòtids (comptant el codó STOP) (CCDS3205.1). Dóna lloc a una proteïna de 1051 aminoàcids segons UCSC (Q03112), RefSeq (NP_005232.1) i Ensembl (ENSP00000264674). Existeix coincidència tant en la seqüència genòmica com en la codificant i també en la proteïna en totes les bases de dades consultades.
| Número exó |
Nucleòtid inici exó |
Nucleòtid final exó |
Llargada exó |
Fase |
Nucleòtids codificants |
| 1 |
170346707 |
170346787 |
80 |
- |
- |
| 2 |
170344179 |
170344314 |
135 |
- |
- |
| 3 |
170331910 |
170332013 |
103 |
- |
49 |
| 4 |
170328325 |
170328542 |
217 |
1 |
217 |
| 5 |
170323061 |
170323209 |
148 |
2 |
148 |
| 6 |
170321537 |
170321691 |
154 |
0 |
154 |
| 7 |
170315864 |
170317221 |
1357 |
1 |
1357 |
| 8 |
170313268 |
170313356 |
88 |
2 |
88 |
| 9 |
170308407 |
170308434 |
27 |
0 |
27 |
| 10 |
170302541 |
170302708 |
167 |
0 |
167 |
| 11 |
170301366 |
170301444 |
78 |
2 |
78 |
| 12 |
170295557 |
170295727 |
170 |
2 |
170 |
| 13 |
170293439 |
170293584 |
145 |
1 |
145 |
| 14 |
170290481 |
170290718 |
237 |
2 |
237 |
| 15 |
170289481 |
170289665 |
184 |
2 |
184 |
| 16 |
170285243 |
170285525 |
282 |
0 |
132 |
5' UTR: 269 nucleòtids // 3' UTR: 147 nucleòtids
- Transcrit Hypothetical protein DKFZp686J18113 (Q9UBK3_HUMAN a Ensembl):
-
Nom: Hypothetical protein DKFZp686J18113
Identificació: ENST00000343372 (Ensembl), no té identificador a RefSeq ja que no hi és present.
Posició: chr3:170283997-170348216
Mida genòmica: 64220 nucleòtids Veure seqüència
Cadena: -
Es tracta de la isoforma més curta del gen. Està formada per 15 exons, 13 dels quals són codificants amb una seqüència de mRNA de 5795 nucleòtids (BX640908). La seqüència codificant comença a l'exó 3 i acaba al 15 i consta de 3129 nucleòtids (comptant el codó STOP) Veure seqüència. Dóna lloc a una proteïna de 1042 aminoàcids segons UCSC (BX640908) i de 166 aminoàcids segons la base de dades d'Ensembl (ENSP00000340389). En aquest cas existeix coincidència en les bades de dades pel que fa a la seqüència genòmica del transcrit però no pel que fa a la proteïna. Tot i això, nosaltres considerem més fiable la proteïna de UCSC de llargada 1042 i que comença amb ATG codificant per metionina, de manera que les següents dades correspondran únicament a aquesta proteïna.
| Número exó |
Nucleòtid inici exó |
Nucleòtid final exó |
Llargada exó |
Fase |
Nucleòtids codificants |
| 1 |
170346707 |
170346787 |
80 |
- |
- |
| 2 |
170344179 |
170344314 |
135 |
- |
- |
| 3 |
170331910 |
170332013 |
103 |
- |
49 |
| 4 |
170328325 |
170328542 |
217 |
1 |
217 |
| 5 |
170323061 |
170323209 |
148 |
2 |
148 |
| 6 |
170321537 |
170321691 |
154 |
0 |
154 |
| 7 |
170315864 |
170317221 |
1357 |
1 |
1357 |
| 8 |
170313268 |
170313356 |
88 |
2 |
88 |
9 |
170302541 |
170302708 |
167 |
0 |
167 |
| 10 |
170301366 |
170301444 |
78 |
2 |
78 |
11 |
170295557 |
170295727 |
170 |
2 |
170 |
| 12 |
170293439 |
170293584 |
145 |
1 |
145 |
13 |
170290481 |
170290718 |
237 |
2 |
237 |
| 14 |
170289481 |
170289665 |
184 |
2 |
184 |
15 |
170285243 |
170285525 |
282 |
0 |
132 |
5' UTR: 1201 nucleòtids // 3' UTR: 1294 nucleòtids
Comparant els dos transcrits es veu que difereixen principalment en la llargada de les seves seqüències UTR, és a dir, tenen diferències en la llargada del primer i últim exons. L'altre fet que s'observa en la comparació és que la segona isoforma proteica està originada per un splicing alternatiu que no conté l'exó 9 del primer transcrit, de manera que en aquest afecta a la seqüència de la proteïna ja que és a la regió codificant. Tot i això, observant les taules veiem que aquest exó està en fase 0, de manera que no hi ha canvi en la pauta de lectura. La diferència entre les dues proteïnes, doncs, recau únicament en la falta de 9 aminoàcids en la segona isoforma que són els que provenen de l'exó 9 del primer transcrit.
Tot i aquestes diferències entre els dos transcrits, no s'altera la presència dels dominis funcionals d'aquestes proteïnes. És a dir, la proteïna resultant del transcrit EVI1 té 10 motius Zinc Finger, i el fet de que no estiguin codificats per l'exó 9, fa que també estiguin presents a la proteïna resultant del transcrit Q9UBK3_HUMAN. Els 10 motius Zinc Finger s'agrupen en dos grans dominis, el primer dels quals agrupa els 7 primers i el segon agrupa els altres tres dominis Zinc Finger.
| Representació |
Seqüència |
 |
NP_005232.1
MKSEDYPHETMAPDIHEERQYRCEDCDQLFESKAELADHQKFPCSTPHSAFSMVEEDFQQKLESEN
DLQEIHTIQECKECDQVFPDLQSLEKHMLSHTEEREYKCDQCPKAFNWKSNLIRHQMSHDSGKHYE
CENCAKVFTDPSNLQRHIRSQHVGARAHACPECGKTFATSSGLKQHKHIHSSVKPFICEVCHKSYT
QFSNLCRHKRMHADCRTQIKCKDCGQMFSTTSSLNKHRRFCEGKNHFAAGGFFGQGISLPGTPAMD
KTSMVNMSHANPGLADYFGANRHPAGLTFPTAPGFSFSVPGLFPSGLYHRPPLIPASSPVKGLSST
EQTNKSQSPLMTHPQILPATQDILKALSKHPSVGDNKPVELQPERSSEERPFEKISDQSESSDLDD
VSTPSGSDLETTSGSDLESDIESDKEKFKENGKMFKDKVSPLQNLASINNKKEYSNHSIFSPSLEE
QTAVSGAVNDSIKAIASIAEKYFGSTGLVGLQDKKVGALPYPSMFPLPFFPAFSQSMYPFPDRDLR
SLPLKMEPQSPGEVKKLQKGSSESPFDLTTKRKDEKPLTPVPSKPPVTPATSQDQPLDLSMGSRSR
ASGTKLTEPRKNHVFGGKKGSNVESRPASDGSLQHARPTPFFMDPIYRVEKRKLTDPLEALKEKYL
RPSPGFLFHPQFQLPDQRTWMSAIENMAEKLESFSALKPEASELLQSVPSMFNFRAPPNALPENLL
RKGKERYTCRYCGKIFPRSANLTRHLRTHTGEQPYRCKYCDRSFSISSNLQRHVRNIHNKEKPFKC
HLCYRCFGQQTNLDRHLKKHENGNMSGTATSSPHSELESTGAILDDKEDAYFTEIRNFIGNSNHGS
QSPRNVEERMNGSHFKEEKALVPSQNSDLLDDEEVEDEVLLDEEDEDYDITGKTGKEPVTSNLHEG
NPEDDYEETSALEMSCKTSPVRYKEEEYKSGLSALDHIRHFTDSLKMRKMEDNQYSEAELSSFSTS
HVPEELKQPLHRKSKSQAYAMMLSLSDKESLHSTSHSSSNVWHSMARAAAESSAIQSISHV |
Al costat de la representació de la proteïna es mostra la seva seqüència, i marcat en color gris i blau, la seqüència codificant de cada domini Zinc Finger altern.
Torna a l'índex / Torna a l'Estructura genòmica
Un cop analitzats per separat els gens que formen part de la proteïna de fusió anem a analitzar quines parts de la proteïna de fusió corresponen a cadascun dels gens.
Aquesta és la proteïna de fusió que obtenim:
MRIPVDASTSRRFTPPSTALSPGKMSEALPLGAPDAGAALAGKLRSGDRSMVEVLADHPGELVRTDSPNF
LCSVLPTHWRCNKTLPIAFKVVALGDVPDGTLVTVMAGNDENYSAELRNATAAMKNQVARFNDLRFVGRS
GRGKSFTLTITVFTNPPQVATYHRAIKITVDGPREPRNNECVYGNYPEIPLEEMPDADGVASTPSLNIQE
PCSPATSSEAFTPKEGSPYKAPIYIPDDIPIPAEFELRESNMPGAGLGIWTKRKIEVGEKFGPYVGEQRS
NLKDPSYGWEILDEFYNVKFCIDASQPDVGSWLKYIRFAGCYDQHNLVACQINDQIFYRVVADIAPGEEL
LLFMKSEDYPHETMAPDIHEERQYRCEDCDQLFESKAELADHQKFPCSTPHSAFSMVEEDFQQKLESEND
LQEIHTIQECKECDQVFPDLQSLEKHMLSHTEEREYKCDQCPKAFNWKSNLIRHQMSHDSGKHYECENCA
KVFTDPSNLQRHIRSQHVGARAHACPECGKTFATSSGLKQHKHIHSSVKPFICEVCHKSYTQFSNLCRHK
RMHADCRTQIKCKDCGQMFSTTSSLNKHRRFCEGKNHFAAGGFFGQGISLPGTPAMDKTSMVNMSHANPG
LADYFGANRHPAGLTFPTAPGFSFSFPGLFPSGLYHRPPLIPASSPVKGLSSTEQTNKSQSPLMTHPQIL
PATQDILKALSKHPSVGDNKPVELQPERSSEERPFEKISDQSESSDLDDVSTPSGSDLETTSGSDLESDI
ESDKEKFKENGKMFKDKVSPLQNLASINNKKEYSNHSIFSPSLEEQTAVSGAVNDSIKAIASIAEKYFGS
TGLVGLQDKKVGALPYPSMFPLPFFPAFSQSMYPFPDRDLRSLPLKMEPQSPGEVKKLQKGSSESPFDLT
TKRKDEKPLTPVPSKPPVTPATSQDQPLDLSMGSRSRASGTKLTEPRKNHVFGGKKGSNVESRPASDGSL
QHARPTPFFMDPIYRVEKRKLTDPLEALKEKYLRPSPGFLFHPQMSAIENMAEKLESFSALKPEASELLQ
SVPSMFNFRAPPNALPENLLRKGKERYTCRYCGKIFPRSANLTRHLRTHTGEQPYRCKYCDRSFSISSNL
QRHVRNIHNKEKPFKCHLCDRCFGQQTNLDRHLKKHENGNMSGTATSSPHSELESTGAILDDKEDAYFTE
IRNFIGNSNHGSQSPRNVEERMNGSHFKDEKALVTSQNSDLLDDEEVEDEVLLDEEDEDNDITGKTGKEP
VTSNLHEGNPEDDYEETSALEMSCKTSPVRYKEEEYKSGLSALDHIRHFTDSLKMRKMEDNQYSEAELSS
FSTSHVPEELKQPLHRKSKSQAYAMMLSLSDKESLHSTSHSSSNVWHSMARAAAESSAIQSISHV
La part de la proteïna de fusió que prové del gen AML1 apareix en verd. Aquesta part prové del transcrit isoforma b. Això ho podem veure realitzant un aliniament entre la proteïna de fusió i les tres proteïnes resultants dels transcrits Veure aliniament. Així es veu com la isoforma b és la que conté la mateixa seqüència aminoacídica que la proteïna de fusió.
La porció de la proteïna de fusió marcada en vermell és aquella que prové del gen MDS1. Respecte a la proteïna que s'obté de forma normal, amb 169 aminoàcids, la proteïna de fusió conté només 113 aminoàcids. La part que es troba a la proteïna de fusió és la marcada en marró.
MRSKGRARKLATNNECVYGNYPEIPLEEMPDADGVASTPSLNIQEPCSPATSSEAFTPKE
GSPYKAPIYIPDDIPIPAEFELRESNMPGAGLGIWTKRKIEVGEKFGPYVGEQRSNLKDP
SYGWEVHLPRSRRVSVHSWLYLGKRSSDVGIAFSQADVYMPGLQCAFLS
En el cas de l'asparagina (N) marcada en taronja cal remarcar que està codifacada a partir d'un nucleòtid (A) provinent de AML1 i de dos nucleòtids (AT) provinents de MDS1.
La part de la proteïna de fusió que prové del gen EVI1 apareix en blau, i conté 1105 aminoàcids. Aquesta porció prové del segon transcrit del gen EVI1 amb una proteïna de 1042 aminoàcids. La diferència de llargada entre les dues proteïnes es deu a que la proteïna del transcrit comença a codificar a l'exó 3 mentre que la porció de la proteïna de fusió comença a codificar a l'inici de l'exó 2, de manera que la part de la proteïna de fusió és més llarga que la proteïna del transcrit. Entre una i altra hi ha una diferència de 63 aminoàcids.
Torna a l'índex / Torna a l'Estructura genòmica
L'anàlisi de la conservació s'analitza per cada un dels gens individualment i posteriorment per a la proteïna de fusió. S'ha buscat homologia amb totes les espècies que disposen de genoma seqüenciat a Ensembl. En el cas de EVI1 s'ha utilitzat HomoloGene de NCBI.
A la taula següent es mostren les espècies que tenen un gen homòleg al gen AML1 humà:
| Espècie |
Tipus |
Gen |
Percentatge d'homologia (%) |
| Aedes aegypti |
many-to-many |
AAEL006167 |
26 |
| Anopheles gambiae |
many-to-many |
ENSANGG00000011627 |
20 |
| Bos taurus |
1-to-1 |
ENSBTAG00000004742 |
98 |
| Caenorhabditis elegans |
1-to-many |
B0414.2 |
18 |
| Canis familiaris |
1-to-1 |
ENSCAFG00000009596 |
92 |
| Cavia porcellus |
1-to-1 |
ENSCPOG00000002684 |
62 |
| Ciona intestinalis |
1-to-many |
ENSCING00000002253 |
24 |
| Ciona savignyi |
1-to-many |
ENSCSAVG00000004072 |
22 |
| Dasypus novemcinctus |
1-to-1 |
ENSDNOG00000015041 |
46 |
| Drosophila melanogaster |
many-to-many |
CG1689 |
30 |
| Echinops telfairi |
1-to-1 |
ENSETEG00000011313 |
86 |
| Felis catus |
1-to-1 |
ENSFCAG00000006761 |
65 |
| Gallus gallus |
1-to-1 (aparent) |
ENSGALG00000016022 |
66 |
| Gasterosteus aculeatus |
1-to-1 |
ENSGACG00000015276 |
49 |
| Loxodonta africana |
1-to-1 |
ENSLAFG00000004290 |
47 |
| Macaca mulatta |
1-to-1 |
ENSMMUG00000001649 |
99 |
| Monodelphis domestica |
1-to-1 (aparent) |
ENSMODG00000021051 |
85 |
| Mus musculus |
1-to-1 (aparent) |
ENSMUSG00000022952 |
89 |
| Ornithorhynchus anatinus |
1-to-1 |
ENSOANG00000011162 |
12 |
| Oryctolagus cuniculus |
1-to-1 |
ENSOCUG00000013731 |
93 |
| Oryzias latipes |
1-to-1 |
ENSORLG00000020699 |
55 |
| Pan troglodytes |
1-to-1 |
ENSPTRG00000013883 |
100 |
| Rattus norvegicus |
1-to-1 (aparent) |
ENSRNOG00000001704 |
88 |
| Takifugu rubripes |
1-to-1 |
SINFRUG00000149207 |
57 |
| Tetraodon nigroviridis |
1-to-1 |
GSTENG00034595001 |
34 |
| Tupaia belangeri |
1-to-1 |
ENSTBEG00000000192 |
74 |
| Xenopus tropicalis |
1-to-1 (aparent) |
ENSXETG00000014140 |
82 |
A la taula tan sols es mostra el gen ortòleg de cada espècie que mostra el percentatge d'homologia major amb el gen AML1 d'humà.
Com es pot observar, les espècies que mostren un percentatge d'homologia més gran són Pan troglodytes (100%), Macaca mulatta (99%) i Bos taurus (98%). Tenint en compte les relacions evolutives de l'espècie humana amb la resta d'espècie, els resultats obtinguts són lògics, ja que aquestes tres espècies són de les més pròximes filogenèticament a l'espècie humana. La relació evolutiva d'aquest gen es pot veure en aquesta imatge. Pel que fa a altres espècies evolutivament pròximes, el fet que el percentatge d'homologia no sigui tan alt pot ser degut a que només comparem un gen i no tota la relació global d'evolució.
Per altra banda, cal destacar el percentatge d'homologia amb Drosophila melanogaster. El domini Runt d'unió a ATP que presenta aquest gen és homòleg al gen pair-rule runt de Drosophila. Per aquest motiu, aquesta espècie presenta un percentatge d'homologia rellevant, encara que no és excessivament elevat degut a que es compara el gen sencer, i no només el domini Runt.
Pel que fa al conjunt d'espècies que presenten homologia, cal destacar que aquest gen està implicat en el desenvolupament, de manera que és lògic que sigui un gen conservat al llarg del temps.
Torna a l'índex / Torna a la Conservació en altres espècies
A la taula següent es mostren les espècies que tenen un gen homòleg al gen MDS1 humà:
| Espècie |
Tipus |
Gen |
Percentatge d'homologia (%) |
| Bos Taurus |
1-to-1 |
ENSBTAG00000020168 |
93 |
| Canis familiaris |
1-to-1 |
ENSCAFG00000024240 |
95 |
| Cavia porcellus |
1-to-1 |
ENSCPOG00000012256 |
91 |
| Dasypus novemcinctus |
1-to-1 |
ENSDNOG00000017489 |
91 |
| Erinaceus europaeus |
1-to-1 (aparent) |
ENSEEUG00000010532 |
89 |
| Felis catus |
1-to-1 |
FENSFCAG00000004548 |
95 |
| Gasterosteus aculeatus |
1-to-1 |
ENSGACG00000007274 |
37 |
| Loxodonta africana |
1-to-1 |
ENSLAFG00000010308 |
93 |
| Macaca mulatta |
1-to-1 |
ENSMMUG00000031567 |
96 |
| Monodelphis domestica |
1-to-1 |
ENSMODG00000024658 |
81 |
| Mus musculus |
1-to-1 (aparent) |
ENSMUSG00000051636 |
84 |
| Ornithorhynchus anatinus |
1-to-1 |
ENSOANG00000007584 |
76 |
| Rattus norvegicus |
1-to-1(aparent) |
ENSRNOG00000011640 |
88 |
| Tetraodon nigroviridis |
1-to-1 |
GSTENG00003821001 |
24 |
| Tupaia belangeri |
1-to-1 |
ENSTBEG00000009672 |
93 |
| Xenopus tropicalis |
1-to-1 |
ENSXETG00000022835 |
57 |
El resultat obtingut a partir de la informació present a Ensembl ens mostra que l'espècie més propera a Homo sapiens és Macaca mulatta, cosa coherent si pensem en la relació evolutiva global entre les dues espècies.Després trobem altres espècies també properes a Homo sapiens, com Canis familiaris, Mus musculus o Tupaia belangeri.
Com a espècies més llunyanes trobem Tetraodon nigroviridis i Gasterosteus aculeatus, cosa que també és coherent ja que són espècies molt llunyanes d'Homo sapiens. La relació evolutiva d'aquest gen es pot veure en aquesta imatge.
Torna a l'índex / Torna a la Conservació en altres espècies
En el cas del gen EVI1 la informació sobre homologia s'ha obtingut a partir de HomoloGene de NCBI, ja que a la base de dades Ensembl no apareixia cap predicció de les homologies.
| Espècie |
Símbol |
Percentatge d'homologia (%) |
| Pan troglodytes |
LOC460837 |
99.4 |
| Canis familiaris |
LOC488148 |
96.5 |
| Mus musculus |
Evi1 |
93 |
| Rattus norvergicus |
Evi1_predicted |
92.2 |
| Gallus gallus |
LOC424997 |
88.7 |
| Drosophila melanogaster |
ham |
42.1 |
| Anopheles gambiae |
ENSANGG00000009594 |
47.5 |
Segons la informació obtinguda de HomoloGene a la pàgina de NCBI, l'espècie més propera a Homo sapiens és Pan troglodytes, cosa coherent si pensem en la relació evolutiva global entre les dues espècies. Després trobem Canis familiaris i Mus musculus. Així doncs, veiem que existeix una gran homologia entre els mamífers. En principi, Mus musculus hauria de tenir una homologia més gran que Canis familiaris. En aquest cas no és així ja que estem comparant únicament un gen i és possible que per aquest cas concret hi hagi més conservació amb el gos que amb el ratolí. Com a espècies més llunyanes trobem Drosophila melanogaster i Anopheles gambiae, cosa que també és coherent ja que són les espècies més llunyanes d'Homo sapiens.
Torna a l'índex / Torna a la Conservació en altres espècies
Per tal de veure si la proteïna de fusió també presenta homòlegs en altres espècies, hem realitzat un BLASTn amb la base de dades NCBI, amb la seqüència de RNAm de la proteïna de fusió.
Tot els resultats que hem obtingut són prediccions, per la qual cosa, tot i que no ho podem assegurar, podem pensar que aquesta malaltia també afecta altres espècies. Les espècies en les quals es preveu homologia es mostren a continuació. Entre parèntesi es mostra el percentatge d'homologia:
- Pan troglodytes (63%). [XM_516864.2]
- Bos taurus (58%). [XM_610719.3]
- Monodelphis domestica (58%). [XR_030269.1]
- Gallus gallus (52%). [XM_422804.2]
El fet de no obtenir resultats concloents en aquest punt pot deure's al fet que aquesta translocació no ha estat estudiada en altres espècies i, per tant, no es coneix de manera segura el percentatge d'homologia que existeix entre humà i altres espècies. Els que presenten una major homologia predita pertanyen al grup dels mamífers, cosa que pot indicar un major estudi de la translocació en aquest grup.
Torna a l'índex / Torna a la Conservació en altres espècies
2.3. Caracterització de l'expressió
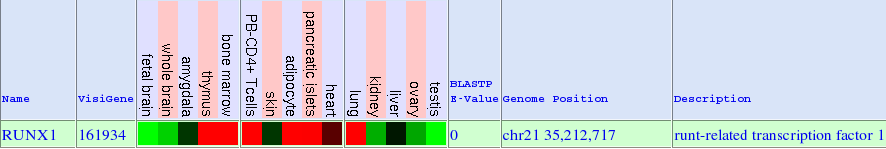
Pel que fa a l'expressió del gen AML1 existeixen diferents fonts d'on s'ha extret informació. En la primera imatge podem veure l'expressió de la proteïna segons el GeneSorter de UCSC. En aquest cas veiem que els majors nivells d'expressió són al tim, medul·la, pàncrees i pulmó. És lògic que la proteïna tingui una major expressió en tim i medul·la a causa de la funció que exerceix.
Una altra font d'informació ha estat l'expressió obtinguda a partir de UCSC en microarrays obtinguts a partir de teixits humans normals. Aquestes dades, però no són coherents amb les obtingudes de les altres fonts d'informació. Això pot ser degut a la utilització de diferents mètodes experimentals per l'estudi d'expressió d'aquesta proteïna.
Dades
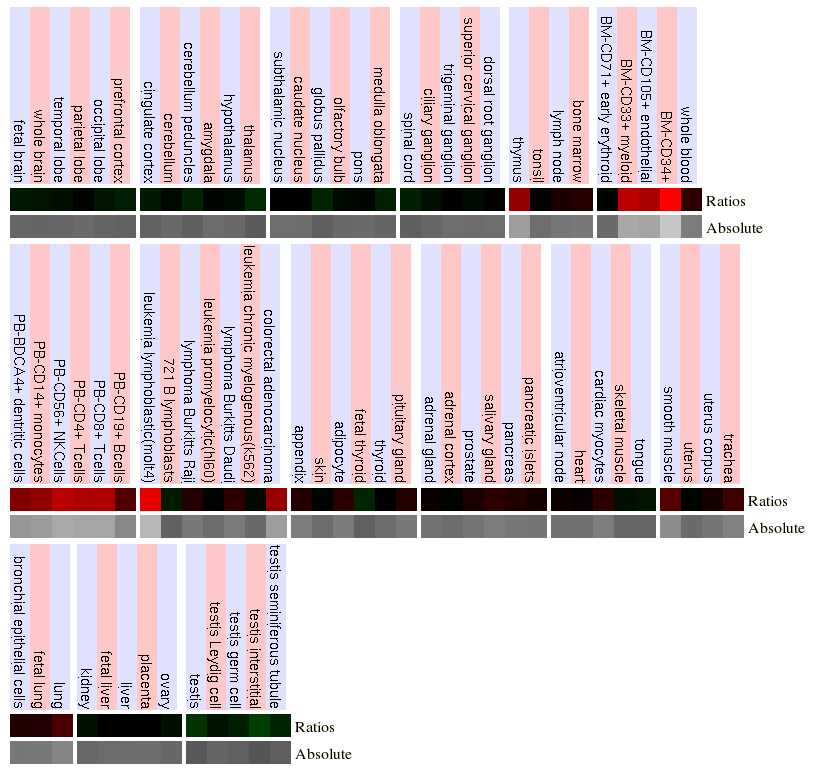
La següent font d'informació consultada procedeix també de UCSC però s'obté de GNF Expression Atlas 2 Data des de U133A i GNF1H Chips. En aquest els resultats són semblants als obtinguts GeneSorter. S'aprecia que els majors nivells d'expressió es troben al tim i també hi ha nivells força elevats en medul·la i cèl.lules implicades en defensa.En aquest últim cas el fet que els limfòcits es desenvolupin al tim explica que tinguin alts nivells de la proteïna. Un últim fet a destacar en aquest punt és que es veu com aquest gen s'expressa a diferents tipus de leucèmies també quan no forma part de la proteïna de fusió.
Per últim s'han consultat dades a de Gene Expression Atlas on es veu que l'expressió més important de la proteïna és a tim. També s'aprecien alts nivells en cèl·lules limfoides que, com s'ha comentat abans, són les que es desenvolupen al tim.
Dades
De la web de UCSC s'ha extret informació sobre l'expressió on es comenta que la proteïna s'expressa a tot arreu tret del cor i el cervell. Aquest és un fet que es relaciona correctament amb els resultats comentats mé amunt. Finalment, i després de fer una comparació entre les diferents fonts d'informació, es pot afirmar que els nivells més grans d'expressió de la proteïna s'obtenen a tim i en cèl·lules limfoides. D'altra banda, amb l'estudi de diferents articles i veient que la proteïna s'expressa en leucèmies, s'extreu que aquesta proteïna també s'expressa en cèl·lules hematopoètiques.
Torna a l'índex / Torna a la Caracterització de l'expressió
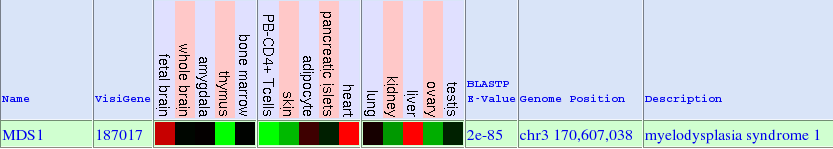
Pel que fa a l'expressió del gen MDS1 existeixen diferents fonts d'on s'ha extret informació. En la primera imatge podem veure l'expressió de la proteïna segons el GeneSorter de UCSC. En aquest cas veiem que els majors nivells d'expressió són al fetge, el cervell del fetus i cor.
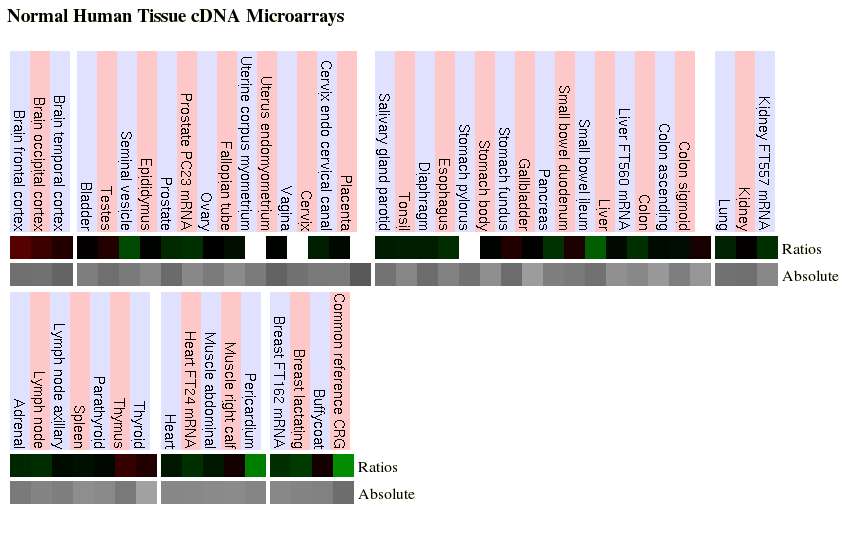
Una altra font d'informació ha estat l'expressió obtinguda a partir de UCSC en microarrays obtinguts a partir de teixits humans normals. A la imatge no s'aprecien grans nivells d'expressió, però els més alts s'obtenen a cervell i fetge. Aquest fet es relacions amb l'obtingut a GeneSorter ja que a ambdós hi ha expressió a fetge i cervell.
La següent font d'informació consultada procedeix també de UCSC però s'obté de GNF Expression Atlas 2 Data from U133A and GNF1H Chips. S'aprecia que els majors nivells d'expressió es troben a cor i fetge, correlacionant amb els resultats obtinguts en les imatges anteriors.
Dades
Per últim s'han consultat dades a Gepis Tissue on es veu que els majors nivells d'expressió s'obtenen a teixit adipós, ovari, cervell i medul.la.
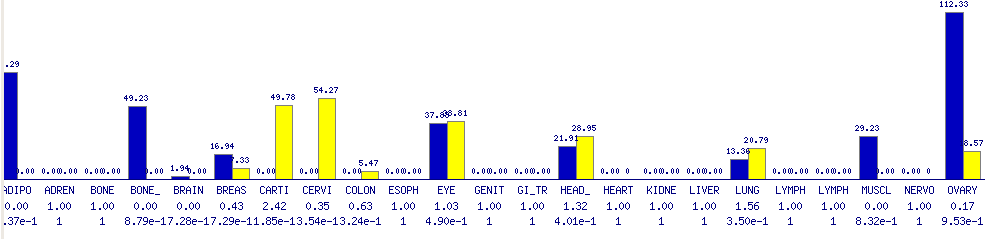
Comparant les diferents fonts d'informació podríem dir que el gen s'expressa principalment a cor, cervell i fetge com a òrgans principals, tot i que també existeix expressió en altres zones. El fet que es coneix molt poc sobre la funció d'aquest gen dificulta l'argumnetació de la localització d'aquesta proteïna als diferents teixits.
Torna a l'índex / Torna a la Caracterització de l'expressió
Per tal d'intentar obtenir informació referent a l'expressió del gen EVI1 s'han fet consultes a difrents fonts. En la primera imatge podem veure l'expressió de la proteïna segons el GeneSorter de UCSC. En aquest cas veiem que els majors nivells d'expressió són a la pell, el pàncrees, pulmons i ronyons.
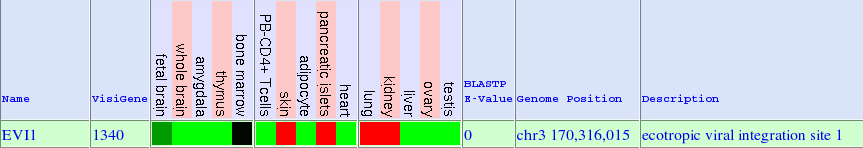
Una altra font d'informació ha estat l'expressió obtinguda a partir de UCSC en microarrays obtinguts a partir de teixits humans normals. A la imatge s'aprecia que els majors nivells d'expressió s'obtenen en general a teixits relacionats amb el sistema reproductor, com cèrvix o pròstata, i també a ronyons i pàncrees, cosa que es relaciona amb l'expressió que s'ha comentat per a la imatge anterior.

La següent font d'informació consultada procedeix també de UCSC però s'obté de GNF Expression Atlas 2 Data des de U133A i GNF1H Chips. S'aprecia que els majors nivells d'expressió es troben a medul·la i, com en els casos anteriors, també a pàncrees, pulmons i sistema reproductor femení.
Dades
Altres dades s'han obtingut de Gene Expression Atlas on es veu que l'expressió més important de la proteïna és a pàncrees, cosa que també s'obté d'altres de les fonts consultades.
Dades
Per últim s'han consultat dades a Gepis Tissue on es veu que els majors nivells d'expressió s'obtenen a òrgans genitals, pulmó, ronyons i pàncrees.
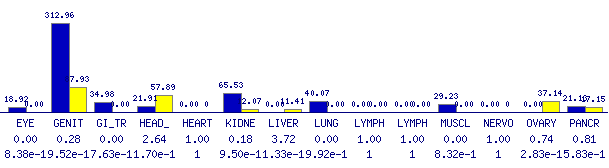
Comparant les diferents fonts d'informació podríem dir que el gen s'expressa principalment a òrgans genitals, pulmó, ronyons i pàncrees. L'afirmació sobre en quins llocs s'obtenen uns nivells d'expressió més elevats es difícil de fer per la divergència existent en les diferents fonts d'informació consultades.
Segons la literatura, l'expressió del gen va ser primer identificada en ratolí on s'expressa en sistema urinari, conducte de Müller, pulmó i cor. Més tard va identificar-se la seva expressió en humà on el gen es troba principalment a ronyó, pulmó pàncrees i ovaris. Aquests resultats d'expressió, són doncs coherents amb els obtinguts de les diferents fonts d'informació. A més cal destacar que aquest gen no s'expressa en cèl·lules hematopoètiques de forma normal, tot i que si que ho fa quan forma part de la proteïna de fusió.
Torna a l'índex / Torna a la Caracterització de l'expressió
Tenint en compte que la translocació que produeix la proteïna de fusió es troba en la fase de crisi blàstica de la leucèmia mieloide crònica i en la síndrome mielodisplàsica, causant transformació anòmala a les cèl·lules hematopoètiques, l'expressió d'aquest gen quimèric es produeix en aquest tipus cel·lular.
Per tal d'obtenir una llista dels factors de transcripció que s'uneixen a les seqüències promotores hem utilitzat, per una banda, el programa PROMO. El procés d'otenció s'ha fet amb uns valors màxims de dissimilaritat del 15% i amb uns valors de RE equally màxims de 0.09. Aquest últim valor és la probabilitat de que la unió es produeixi per atzar en una seqüència de nucleòtids amb una proporció del 25% per cada un. Per tant, considerem que tots els resultats que tenen un valor menor de 0.09 són els que s'uniran a la seqüència promotora. El procediment s'ha repetit per cadascuna de les seqüències promotores, és a dir, un cop per cadascun dels transcrits.
Per tal de complementar el punt de caracterització de les regions promotores, s'ha realitzat un programa en Perl que té per funció trobar els llocs d'unió d'una llista de determinats factors de transcripció a les diferents seqüències promotores obtingudes. A partir d'una matriu de posicions, el programa obté les matrius de pesos per a cadascun dels factors proporcionats, en aquest cas 13, i finalment mitjançant una sèrie de càlculs obtenim els llocs d'unió dels factors a cada seqüència amb un score i p-value determinats.
Analitzant els dos programes, es veu que les matrius de pesos i el número de posicions utilitzades en cadascun dels factors de transcripció són diferents, de manera que poden existir diferències degudes a aquest fet quan es fa la comparació entre els llocs d'unió proporcionats per un o altre programa.
- Isoforma a:
Segons el programa PROMO, en el cas de la isoforma a de AML1 es produeix la unió de 16 factors de transcripció a la seqüència. Alguns d'aquests factors poden unir-se en més d'un lloc diferent al llarg de la seqüència. Es pot observar que els que tenen la possibilitat d'unir-se més a la seqüència són els factors de la família HOXD i ho fan amb diferents valors de RE equally. Veure seqüència promotora.
| Nom del factor |
Posició inici |
Posició final |
Seqüència |
RE Equally |
| NF-AT2 [T01945] |
523 |
532 |
TTATTTTTCC |
0.01 |
| NF-AT2 [T01945] |
435 |
444 |
AATTCTTTCC |
0.02 |
| NF-AT2 [T01945] |
247 |
256 |
TTTGTTTTCC |
0.03 |
| NF-AT2 [T01945] |
167 |
176 |
GGAGCTTTCC |
.05 |
| RBP-Jkappa [T01616] |
171 |
182 |
CTTTCCCAAGTG |
0.01 |
| RBP-Jkappa [T01616] |
445 |
456 |
CTTTCCCAGGCT |
0.01 |
| HNF-3alpha [T02512] |
457 |
464 |
TTAAAATA |
0.02 |
| HNF-3alpha [T02512] |
692 |
699 |
TCAAAATA |
0.05 |
| HOXD9 [T01424] |
514 |
523 |
TCTTTTTATT |
0.02 |
| HOXD9 [T01424] |
380 |
389 |
AATAAAACCA |
0.02 |
| HOXD9 [T01424] |
415 |
424 |
TCCTTTTATT |
0.03 |
| HOXD9 [T01424] |
658 |
667 |
AATTACTATT |
0.03 |
| HOXD9 [T01424] |
68 |
77 |
CCCTTCTATT |
0.03 |
| HOXD9 [T01424] |
185 |
194 |
AATACAAATA |
0.03 |
| HOXD9 [T01424] |
722 |
731 |
AATAACAACA |
0.04 |
| HOXD10 [T01425] |
380 |
389 |
AATAAAACCA |
0.02 |
| HOXD10 [T01425] |
514 |
523 |
TCTTTTTATT |
0.02 |
| HOXD10 [T01425] |
658 |
667 |
AATTACTATT |
0.03 |
| HOXD10 [T01425] |
415 |
424 |
TCCTTTTATT |
0.03 |
| HOXD10 [T01425] |
185 |
194 |
AATACAAATA |
0.03 |
| HOXD10 [T01425] |
68 |
77 |
CCCTTCTATT |
0.03 |
| HOXD10 [T01425] |
722 |
731 |
AATAACAACA |
0.04 |
| PPAR-alpha:RXR-alpha [T05221] |
37 |
47 |
CAGCCCCAGGA |
0.02 |
| SRY [T00997] |
246 |
254 |
CTTTGTTTT |
0.03 |
| SRY [T00997] |
1000 |
1008 |
CTTTGGGCC |
0.07 |
| SRY [T00997] |
751 |
759 |
TCAGCAAAG |
0.08 |
| SRY [T00997] |
217 |
225 |
CTTTGTCCC |
0.08 |
| PEA3 [T00685] |
620 |
628 |
CCACATCCT |
0.04 |
| AP-1 [T00029] |
781 |
789 |
ACTGAGTCA |
0.05 |
| AR [T00040] |
216 |
224 |
GCTTTGTCC |
0.06 |
| PXR-1:RXR-alpha [T05671] |
144 |
151 |
TGAACTAG |
0.07 |
| PXR-1:RXR-alpha [T05671] |
1061 |
1068 |
TGAACCCA |
0.07 |
| TCF-4E [T02878] |
753 |
759 |
AGCAAAG |
0.07 |
| PR B [T00696] |
963 |
969 |
AACAGTT |
0.07 |
| c-Jun [T00133] |
783 |
789 |
TGAGTCA |
0.07 |
| c-Jun [T00133] |
779 |
785 |
TGACTGA |
0.07 |
| PR A [T01661] |
963 |
969 |
AACAGTT |
0.07 |
| GR [T05076] |
711 |
717 |
CAAAAAC |
.08 |
Per altra banda, mitjançant el programa Perl, hem obtingut la següent llista de factors i la seva probabilitat:
| Nom del factor |
Posició inici |
Posició final |
Score |
p value |
| AP-1 [T00029] |
784 |
790 |
3.95 |
0.08 |
| AR [T00040] |
963 |
969 |
2.40 |
0.78 |
| c-Myc [T00140] |
920 |
925 |
-995 |
0.91 |
| NF-AT1 [T00550] |
955 |
961 |
3 |
0.43 |
| NF-kappaB [T00590] |
170 |
178 |
2.78 |
0.16 |
| SRF [T00764] |
534 |
542 |
-995 |
0.57 |
| YY1 [T00915] |
946 |
951 |
2.17 |
0.94 |
| RXR-alpha [T01345] |
1062 |
1067 |
3.14 |
0.17 |
| HIF-1 [T01609] |
477 |
485 |
-995 |
0.32 |
| AhR [T01795] |
274 |
280 |
-996 |
0.89 |
| PU.1 [T02068] |
869 |
875 |
2.85 |
0.34 |
| HNF-4 [T02758] |
1004 |
1011 |
-995.6 |
0.61 |
| NRSF [T06124] |
36 |
44 |
-994 |
0.1 |
Comparant els dos resultats, podem observar que de la llista de factors de transcripció proporcionada, segons els resultats del PROMO, tan sols s'uneixen 2 factors: AP-1 [T00029] i AR [T00040]. Analitzant cada un d'aquests de forma més detadallada, veiem que el primer factor s'uneix a unes posicions similars segons els dos programes. Les diferències però, són degudes a que la llargada d'aquest factor és diferent en cadascun dels programes, la qual cosa modifica els valors i les seqüències obtingudes. Així doncs podem considerar que aquest factor si que s'uneix a la seqüència promotora del transcrit isoforma a.
Pel que fa al factor AR [T00040], tot i que està present als resultats obtinguts amb els dos programes, les posicions on s'uneix el factor no coincideixen. A mé el valor de p-value obtingut amb el programa Perl és molt alt, de forma que no podem considerar que aquest factor s'uneixi en les posicions predites pel programa Perl.
La resta de factors que apareixen a la taula dels resultats del PROMO, si que considerem que s'uneixen a la seqüència promotora del gen, ja que hem seleccionat aquells que tenen un valor de RE equally de màxim 0.09.
Isoforma b:
En el cas de la isoforma b de AML1 es produeix la unió de 12 factors de transcripció a la seqüència. Veure seqüència promotora. Igual que en el cas anterior, alguns d'alquests factors poden unir-se en més d'un lloc diferent al llarg de la seqüència i també amb diferents valors de RE equally. Els factors que tenen més possibilitat d'unió en aquest cas són els de la família NF-AT.
| Nom del factor |
Posició inici |
Posició final |
Seqüència |
RE Equally |
| AhR:Arnt [T05394] |
724 |
733 |
GCACGCGGGC |
0.009 |
| AhR:Arnt [T05394] |
349 |
358 |
GCACGCGCGG |
0.02 |
| AhR:Arnt [T05394] |
696 |
705 |
GCCTGCGTGT |
0.08 |
| AhR:Arnt [T05394] |
1058 |
1067 |
CCACGCTGCC |
0.08 |
| GCF [T00320] |
1070 |
1078 |
GCCCTGCGC |
0.01 |
| GCF [T00320] |
503 |
511 |
GCGCAGGAT |
0.01 |
| GCF [T00320] |
836 |
844 |
GCGCGGGCC |
0.05 |
| GCF [T00320] |
611 |
619 |
GCGCCGGCC |
0.05 |
| GCF [T00320] |
131 |
139 |
TGCCGGCGC |
0.05 |
| E2F-1 [T01542] |
292 |
299 |
GCGGTAAA |
0.02 |
| E2F-1 [T01542] |
1020 |
1027 |
GCGGGACG |
0.08 |
| NF-AT1 [T00550] |
650 |
658 |
TCTTTTTCC |
0.02 |
| NF-AT1 [T00550] |
218 |
226 |
GGAAACTCT |
0.03 |
| NF-AT1 [T00550] |
1088 |
1096 |
CTTCTTTCC |
0.04 |
| NF-AT2 [T01945] |
218 |
227 |
GGAAACTCTT |
0.02 |
NF-AT2 [T01945] |
649 |
658 |
TTCTTTTTCC |
0.04 |
NF-AT2 [T01945] |
202 |
211 |
GGGTCTTTCC |
0.04 |
NF-AT2 [T01945] |
1087 |
1096 |
ACTTCTTTCC |
0.04 |
NF-AT2 [T01945] |
977 |
988 |
GTTCATTTCC |
0.06 |
NF-AT2 [T01945] |
173 |
182 |
TCAACTTTCC |
0.06 |
| c-Ets-2 [T00113] |
947 |
955 |
TTCCTCCGG |
0.04 |
| c-Ets-2 [T00113] |
375 |
383 |
TGCAAGGAA |
0.09 |
| c-Ets-1 [T00112] |
216 |
222 |
TAGGAAA |
0.06 |
| NF-1 [T00539] |
839 |
846 |
CGGGCCAA |
0.07 |
| ENKTF-1 [T00255] |
1076 |
1083 |
CGCCGCCA |
0.07 |
| STAT1beta [T01573] |
885 |
894 |
TACCGGAAAG |
0.08 |
| STAT1beta [T01573] |
981 |
990 |
ATTTCCAGGC |
0.08 |
| USF2 [T00878] |
20 |
29 |
TCCGCACCTG |
0.08 |
| IRF-1 [T00423] |
214 |
222 |
ACTAGGAAA |
0.09 |
Ja que les seqüències promotores dels dos transcrits són diferents, veiem que no hi ha un alt grau de coincidència entre els factos de transcripció que s'uneixen a una o altra pel que fa als resultats del PROMO.
El resultat obtingut amb el promgrama Perl és el següent:
| Nom del factor |
Posició inici |
Posició final |
Score |
p value |
| AP-1 [T00029] |
381 |
387 |
-995 |
0.69 |
| AR [T00040] |
341 |
347 |
-996 |
0.86 |
| c-Myc [T00140] |
25 |
30 |
-995 |
0.78 |
| NF-AT1 [T00550] |
713 |
719 |
2.92 |
0.17 |
| NF-kappaB [T00590] |
944 |
952 |
-995 |
0.25 |
| SRF [T00764] |
997 |
1005 |
-1994.2 |
0.7 |
| YY1 [T00915] |
942 |
947 |
2.68 |
0.67 |
| RXR-alpha [T01345] |
174 |
179 |
-995.8 |
0.66 |
| HIF-1 [T01609] |
971 |
979 |
3.88 |
0.02 |
| AhR [T01795] |
701 |
707 |
2.90 |
0.31 |
| PU.1 [T02068] |
379 |
385 |
3.14 |
0.28 |
| HNF-4 [T02758] |
943 |
950 |
-995.2 |
0.34 |
| NRSF [T06124] |
20 |
28 |
-994 |
0.06 |
En aquest cas només veiem que coincideix el factor NF-AT1 en els resultats dels dos programes. De totes formes però les posicions d'unió són diferents, degut a que altre cop les matrius dels factors no són les mateixes. A part, considerem que aquest factor no s'uneix a les posicions obtingudes amb el Perl, ja que el p-value és elevat (0.17).
El factor HIF-1 [T01609], segons els resultats del Perl, s'uniria a la seqüència ja que el seu score és elevat i el p-value és molt baix. De totes formes, no podem comparar-ho amb els resultats del PROMO, ja que aquest factor no està present en la llista de factors d'aquest programa. Per últim es considera que els factors de transcipció que apareixen a la taula del programa PROMO si que s'uneixen a la seqüència.
Torna a l'índex / Torna a la Caracterització de les regions promotores
En el cas del gen MDS1 el procés només s'ha realitzat un cop ja que el gen té un únic transcrit. En aquest cas es produeix la unió de 12 factors de transcripció. Veure seqüència promotora. Com en el cas de la isoforma b de AML1, els factors que tenen més possibilitat d'unió en aquest cas són els de la família NF-AT.
| Nom del factor |
Posició inici |
Posició final |
Seqüència |
RE Equally |
| RelA [T00594] |
37 |
47 |
GGAATTCCCAT |
0.01 |
| RelA [T00594] |
661 |
671 |
GTGGGAAAAAC |
0.02 |
| RBP-Jkappa [T01616] |
39 |
50 |
AATTCCCATTTC |
0.01 |
| RBP-Jkappa [T01616] |
658 |
669 |
ATGGTGGGAAAA |
0.01 |
| RAR-alpha1 [T00719] |
142 |
154 |
TTCTTTTGACCCC |
0.01 |
| NF-AT1 [T00550] |
344 |
352 |
GGAAAGAAT |
0.01 |
| NF-AT1 [T00550] |
664 |
672 |
GGAAAAACA |
0.02 |
| NF-AT1 [T00550] |
597 |
605 |
TTTTTTTCC |
0.03 |
| NF-AT1 [T00550] |
405 |
413 |
GGAAAAAAC |
0.04 |
| NF-AT1 [T00550] |
86 |
94 |
GGAAATTTC |
0.07 |
| NF-kappaB [T00590] |
35 |
46 |
TAGGAATTCCCA |
0.01 |
| NF-AT2 [T01945] |
344 |
353 |
GGAAAGAATA |
0.02 |
| NF-AT2 [T01945] |
86 |
95 |
TTTTTTTCC |
0.03 |
| NF-AT2 [T01945] |
405 |
414 |
GGAAATTTCA |
0.04 |
| NF-AT2 [T01945] |
119 |
128 |
GTGATTTTCC |
0.05 |
| NF-AT2 [T01945] |
664 |
673 |
GGAAAAACAA |
0.05 |
| NF-AT2 [T01945] |
596 |
605 |
ACTTCTTTCC |
0.04 |
| c-Ets-2 [T00113] |
81 |
89 |
TTAAAGGAA |
0.03 |
| c-Ets-2 [T00113] |
125 |
133 |
TTCCTCCCT |
0.04 |
| IRF-1 [T00423] |
82 |
90 |
TAAAGGAAA |
0.04 |
| IRF-1 [T00423] |
340 |
348 |
GTAGGGAAA |
0.07 |
| IRF-1 [T00423] |
660 |
668 |
GGTGGGAAA |
0.07 |
| IRF-1 [T00423] |
601 |
609 |
TTTCCCCCC |
0.08 |
| STAT1beta [T01573] |
82 |
91 |
TAAAGGAAAT |
0.06 |
| STAT1beta [T01573] |
401 |
410 |
TCGAGGAAAA |
0.08 |
| NF-1 [T00539] |
383 |
390 |
TTGGTATT |
0.07 |
| NF-1 [T00539] |
360 |
367 |
TGTCCCAA |
0.07 |
| Pax-5 [T00070] |
765 |
771 |
GGGCGAG |
0.07 |
| ENKTF-1 [T00255] |
911 |
918 |
TGGCGGCG |
0.07 |
Els resultats amb el programa Perl són:
| Nom del factor |
Posició inici |
Posició final |
Score |
p value |
| AP-1 [T00029] |
5 |
11 |
-995 |
0.86 |
| AR [T00040] |
1064 |
1070 |
-996 |
0.98 |
| c-Myc [T00140] |
948 |
953 |
-996 |
0.98 |
| NF-AT1 [T00550] |
406 |
412 |
3.44 |
0.08 |
| NF-kappaB [T00590] |
38 |
46 |
4.16 |
0.02 |
| SRF [T00764] |
709 |
717 |
-994 |
0.2 |
| YY1 [T00915] |
659 |
664 |
2.33 |
0.87 |
| RXR-alpha [T01345] |
356 |
361 |
1.90 |
0.98 |
| HIF-1 [T01609] |
944 |
952 |
-995 |
0.46 |
| AhR [T01795] |
174 |
180 |
-996 |
0.93 |
| PU.1 [T02068] |
404 |
410 |
-995 |
0.48 |
| HNF-4 [T02758] |
715 |
722 |
-996 |
0.9 |
| NRSF [T06124] |
293 |
301 |
-995 |
0.29 |
Comparant els resultats obtinguts dels dos programes diferents, veiem que només coincideixen els factors de transcripció NF-AT1 [T00550] i NF-kappaB [T00590]. Ambdós factors s'uneixen a unes posicions similars segons els dos resultats, i les diferències de posició es deuen altre cop a la utilització de diferents matrius. Així doncs, considerem que s'uneixen a la regió promotora tots els factors que surten a la taula de resultats del PROMO i els dos que acabem de comentar, ja que el seu valor de probabilitat d'unió a l'atzar (p-value) és baix i el seu score és elevat.
Torna a l'índex / Torna a la Caracterització de les regions promotores
En el gen EVI1 hi ha 2 transcrits, de manera que s'han obtingut dues seqüències promotores que amb el programa PROMO hem comparat amb els factors de transcripció per tal de veure quins s'unien.
- Transcrit EVI1:
En el cas d'aquest trasncrit es produeix la unió de 11 factors de transcripció a la seqüència. Veure seqüència promotora. En aquest cas són també els de la família NF-AT els que més s'uneixen a la seqüència promotora.
| Nom del factor |
Posició inici |
Posició final |
Seqüència |
RE Equally |
| MAZ [T00490] |
882 |
894 |
TCCCCTCCCTCCG |
0.01 |
| MAZ [T00490] |
877 |
889 |
CTCCTTCCCCTCC |
0.01 |
| MAZ [T00490] |
34 |
46 |
TCCCCTCGCCTCC |
0.04 |
| NF-AT1 [T00550] |
758 |
766 |
GCGATTTCC |
0.01 |
| NF-AT1 [T00550] |
607 |
615 |
GGAAAAAAA |
0.03 |
| NF-AT1 [T00550] |
696 |
704 |
CTTCTTTCC |
0.04 |
| NF-AT1 [T00550] |
647 |
655 |
ACAGTTTCC |
0.04 |
| NF-AT1 [T00550] |
395 |
403 |
GGAAATCGA |
0.06 |
| NF-AT1 [T00550] |
855 |
863 |
CTCTTTTCC |
0.08 |
| NF-AT1 [T00550] |
84 |
92 |
TTGTTTTCC |
0.08 |
| T3R-beta1 [T00851] |
191 |
199 |
GGGAGGTGA |
0.02 |
| STAT1beta [T01573] |
761 |
770 |
ATTTCCTTGT |
0.02 |
| NF-AT2 [T01945] |
646 |
655 |
GACAGTTTCC |
0.02 |
| NF-AT2 [T01945] |
607 |
616 |
GGAAAAAAAG |
0.03 |
| NF-AT2 [T01945] |
395 |
404 |
GGAAATCGAG |
0.03 |
| NF-AT2 [T01945] |
241 |
250 |
GGAAAAAGCC |
0.04 |
| NF-AT2 [T01945] |
83 |
92 |
CTTGTTTTCC |
0.05 |
| NF-AT2 [T01945] |
695 |
704 |
CCTTCTTTCC |
0.06 |
| c-Ets-2 [T00113] |
770 |
778 |
TTCCTCCTG |
0.03 |
| c-Ets-2 [T00113] |
701 |
709 |
TTCCTCCTC |
0.03 |
| c-Ets-2 [T00113] |
860 |
868 |
TTCCTTGCG |
0.08 |
| c-Ets-2 [T00113] |
652 |
660 |
TTCCTGCCG |
0.08 |
| c-Ets-2 [T00113] |
763 |
771 |
TTCCTTGTT |
0.09 |
| c-Ets-2 [T00113] |
602 |
610 |
TAAAAGGAA |
0.09 |
| IRF-1 [T00423] |
603 |
611 |
AAAAGGAAA |
0.04 |
| IRF-1 [T00423] |
762 |
770 |
TTTCCTTGT |
0.04 |
| IRF-1 [T00423] |
237 |
245 |
AGGGGGAAA |
0.07 |
| IRF-1 [T00423] |
88 |
96 |
TTTCCCCCA |
0.08 |
| IRF-1 [T00423] |
391 |
399 |
ATTTGGAAA |
0.09 |
| IRF-1 [T00423] |
700 |
708 |
TTTCCTCCT |
0.09 |
| Pax-5 [T00070] |
707 |
713 |
CTCGCCC |
0.07 |
| Pax-5 [T00070] |
756 |
762 |
GGGCGAT |
0.07 |
| Pax-5 [T00070] |
620 |
626 |
GGGCGA |
0.07 |
| c-Myb [T00137] |
990 |
997 |
CCGAGTTC |
0.07 |
| PXR:RXR-alpha [T05671] |
202 |
209 |
TGAACCAG |
0.07 |
| RAR-beta [T00721] |
258 |
267 |
ACCAAACCCG |
0.08 |
Els resultats del programa Perl per aquest transcrit són:
| Nom del factor |
Posició inici |
Posició final |
Score |
p value |
| AP-1 [T00029] |
1051 |
1057 |
2.26 |
0.48 |
| AR [T00040] |
101 |
107 |
2.77 |
0.45 |
| c-Myc [T00140] |
966 |
971 |
-995 |
0.62 |
| NF-AT1 [T00550] |
242 |
248 |
3.8 |
0.05 |
| NF-kappaB [T00590] |
832 |
840 |
-995 |
0.72 |
| SRF [T00764] |
548 |
556 |
-995 |
0.38 |
| YY1 [T00915] |
318 |
323 |
-996 |
1 |
| RXR-alpha [T01345] |
203 |
208 |
3.11 |
0.2 |
| HIF-1 [T01609] |
795 |
803 |
-996 |
0.97 |
| AhR [T01795] |
341 |
347 |
2.35 |
0.65 |
| PU.1 [T02068] |
211 |
217 |
2.34 |
0.35 |
| HNF-4 [T02758] |
1062 |
1069 |
-995 |
0.9 |
| NRSF [T06124] |
486 |
494 |
-1994 |
0.97 |
Per aquest transcrit, considerem que s'uneixen a la regió promotora tots els factors presents a la taula del programa PROMO, i també el factor NF-AT1 [T00550] dels resultats del programa Perl, ja que mostra un valor de score elevat i un p-value molt baix. Aquest factor apareix a les dues taules però les posicions que mostra la taula del Perl no coincideixen amb les del PROMO, la qual cosa també pot ser deguda a les diferents matrius utilitzades.
- Transcrit Hypothetical protein DKFZp686J18113 (Q9UBK3_HUMAN):
En el segon transcrit del gen EVI1 es produeix la unió de 15 factors. Veure seqüència promotora. En aquest cas, veiem certa coincidència entre els factors que s'uneixen a un i altre transcrit segons la comparació de les taules obtingudes al PROMO. Tot i això ho fan a diferents posicions ja que les dues seqüències promotores són diferents.
| Nom del factor |
Posició inici |
Posició final |
Seqüència |
RE Equally |
| HNF-3alpha [T02512] |
598 |
605 |
TTAAAATA |
0.02 |
| NF-AT2 [T01945] |
74 |
83 |
CACACTTTCC |
0.02 |
| NF-AT2 [T01945] |
1064 |
1073 |
GGAAAGAAAT |
0.03 |
| NF-AT2 [T01945] |
734 |
743 |
GGAAATGTAC |
0.04 |
| NF-AT2 [T01945] |
1050 |
1059 |
GGCTATTTCC |
0.04 |
| NF-AT2 [T01945] |
139 |
148 |
TTAACTTTCC |
0.05 |
| HNF-1B [T01950] |
374 |
382 |
AGTTATTAA |
0.03 |
| HNF-1B [T01950] |
60 |
68 |
AGTTTAACC |
0.05 |
| HNF-1B [T01950] |
136 |
144 |
TGCTTAACT |
0.05 |
| SRY [T00997] |
498 |
506 |
GTATCAAAG |
0.03 |
| SRY [T00997] |
510 |
518 |
ATGGCAAAG |
0.07 |
| SRY [T00997] |
251 |
259 |
CTTACAAAG |
0.07 |
| SRY [T00997] |
133 |
141 |
CTTTGCTTA |
0.08 |
| AR [T00040] |
111 |
119 |
CAAGTGTCC |
0.04 |
| NF-AT1 [T01948] |
733 |
742 |
TGGAAATGTA |
0.04 |
| NF-AT1 [T01948] |
75 |
84 |
ACACTTTCCA |
0.04 |
| NF-AT1 [T01948] |
140 |
149 |
TAACTTTCCA |
0.04 |
| HNF-1C [T01951] |
59 |
67 |
AAGTTTAAC |
0.05 |
| HNF-1C [T01951] |
375 |
383 |
GTTATTAAA |
0.05 |
| LEF-1 [T02905] |
499 |
506 |
TATCAAAG |
0.005 |
| Elk-1 [T00250] |
644 |
652 |
CTTCCTCTG |
0.05 |
| Elk-1 [T00250] |
318 |
326 |
CTTCCAATG |
0.07 |
| STAT1beta [T01573] |
1054 |
1063 |
ATTTCCCTGG |
0.07 |
| POU2F1 [T00641] |
574 |
584 |
TATATGCCAAT |
0.06 |
| POU2F1 [T00641] |
1 |
11 |
ATTTGCACTTG |
0.06 |
| TFIID [T00820] |
633 |
639 |
TTTTGCA |
0.07 |
| c-Myb [T00137] |
57 |
64 |
TAAAGTTT |
0.07 |
| TCF-4E [T02878] |
133 |
139 |
CTTTGCT |
0.07 |
| E2F-1 [T01542] |
568 |
575 |
GCGGGTTA |
0.08 |
Per aquest transcrit, els resultats d'executar el programa Perl són:
| Nom del factor |
Posició inici |
Posició final |
Score |
p value |
| AP-1 [T00029] |
529 |
535 |
1.5 |
0.58 |
| AR [T00040] |
810 |
816 |
2.41 |
0.77 |
| c-Myc [T00140] |
112 |
117 |
-995 |
0.89 |
| NF-AT1 [T00550] |
1065 |
1071 |
3.72 |
0.08 |
| NF-kappaB [T00590] |
1087 |
1095 |
-996 |
0.86 |
| SRF [T00764] |
617 |
625 |
-995 |
0.4 |
| YY1 [T00915] |
194 |
199 |
3.06 |
0.22 |
| RXR-alpha [T01345] |
809 |
814 |
2.73 |
0.58 |
| HIF-1 [T01609] |
108 |
116 |
-994 |
0.1 |
| AhR [T01795] |
1083 |
1089 |
-996 |
0.82 |
| PU.1 [T02068] |
667 |
673 |
-996 |
0.98 |
| HNF-4 [T02758] |
168 |
175 |
-995 |
0.57 |
| NRSF [T06124] |
70 |
78 |
-994 |
0.16 |
En aquest transcrit, només coincideix el factor de transcripció AR [T00040] entre les dues taules de resultats. De totes formes, altra vegada no coincideixen les posicions d'unió a la seqüència promotora, també degut a les diferents matrius dels factors. Per altra banda, el p-value d'aquest factor segons els resultats del programa Perl és molt elevat, per la qual cosa considerem que no s'uneix. En canvi, si que creiem que s'uneixen tota la llista de factors obtinguda del PROMO, i el factor NF-AT1 [T00550] que surt a la llista del Perl, ja que els seus valors de probabilitat d'atzar són baixos.
En aquest cas, cal destacar que a les dues taules apareix el factor NF-AT1 però el número que tenen associat és diferent, de manera que considerem que són factors diferents.
Torna a l'índex / Torna a la Caracterització de les regions promotores
En el cas de la proteïna de fusió, els factors que s'uneixen a la seva seqüència promotora considerem que són els mateixos que els de la isoforma b del gen AML1, ja que la regió promotora de la proteïna de fusió és la mateixa que la de la isoforma b del gen AML1, degut a que aquest és el primer gen de la proteïna de fusió.
Torna a l'índex / Torna a la Caracterització de les regions promotores
2.1. Funció dels gens
A partir de la base de dades Gene Ontology, hem obtingut la relació de termes que s'associen al gen AMl1. Es mostren a continuació:
- Nucli (GO:0005634)
- Unió a ATP (GO:0005524)
- Unió a proteïnes (GO:0005515)
- Activitat de factor de transcripció (GO:0003700)
- Activitat d'activador transcripcional (GO:0016563)
- Hematopoesi (GO:0030097)
- Desenvolupament multicel·lular de l'organisme (GO:0007275)
- Regulació positiva de l'angiogènesi (GO:0045766)
- Regulació positiva de la diferenciació granulocítica (GO:0030854)
- Regulació positiva de la transcripció del promotor de l'ARN polimerasa II (GO:0045944)
- Regulació de la transcricpió, dependent d'ADN (GO:0006355)
Segons informació extreta de la literatura, el gen AML1 codifica per un important regulador de l'hematopoesi i de la diferenciació limfoblà:stica. El producte d'aquest gen és una proteïna d'unió a l'ADN (subunitat α) que, juntament amb una subunitat β que no s'uneix a l'ADN, forma un factor de transcripció heterodimèric anomenat Core Binding Factor (CBF). Aquest factor regula la transcripció de diversos gens, mitjançant l'actuació conjunta amb diverses molècules coactivadores o corepressores, de manera que es permet la regulació dels gens necessaris per la correcta diferenciació hematopoètica.
Per la correcta funció d'aquesta proteïna és necessària la integritat del domini Runt. Aquesta regió permet la unió de la proteïna a l'ADN, mitjançant el seu extrem N-terminal, i també la heterodimerització amb la subunitat β, per poder formar correctament el factor CBF.
Torna a l'índex / Torna a la Funció dels gens
Descripció de la funció del gen MDS1:
A la base de dades Gene Ontology tan sols apareix un terme referent al gen MDS1:
- Activitat de factor de transcripció (GO:0003700)
Per intentar obtenir més informació de la funció d'aquest gen hem fet una cerca a la literatura científica. De totes formes però, no hem aconseguit obtenir una definició clara de la seva funció, ja que la informació referent a aquest gen sempre s'inclou associada a la descripció de la funció dels altres dos gens implicats en la mateixa translocació. Això és degut a que realment no es coneix la funció d'aquest gen individualment.
Torna a l'índex / Torna a la Funció dels gens
Descripció de la funció del gen EVI1:
Les funcions associades a EVI1 segons Gene Ontology són:
- Nucli (GO:0005634)
- Funció molecular (GO:0003674)
- Procés molecular (GO:0008150)
Tot i això, quan s'entra en cadascun dels termes no apareix cap explicació concreta sobre la funció que realitza el gen. Així doncs, s'extreu la informació sobre la funcionalitat del gen de diversos articles obtinguts a PubMed.
Segons la literatura, el gen va ser identificat en primer lloc com a un locus comú d'integració per a retrovirus. EVI1 pertany a una família de factors involucrats en leucèmia i és capaç d'interactuar amb diversos activadors i repressors. Es considera que EVI1 és un factor de transcripció complex amb múltiples funcions.
Existeix poca informació sobre quin és el rol normal del gen a la cèl·lula i el que es coneix deriva d'estudis on s'ha sobreexpressat el gen i també a partir d'estudis amb gens reporter.
En alguns tipus cel·lulars el gen incrementa l'expressió de Jun i Fos resultant en una activació de AP-1. D'altra banda, EVI1 també s'ha relacionat amb la protecció de les cèl·lules davant la mort induïda per estrès. Més estudis suggereixen que EVI1 té una funció com a factor repressor d'unió a ADN, tot i que aquesta funció ha estat només demostrada in vitro. S'han realitzat també estudis en ratolins on es creu que el gen juga un paper essencial en el procés d'organogènesi i morfogènesi, com també en proliferació i diferenciació.
En el 70-80% de les cèl·lules la proteïna es troba al nucli i la seva funció es veu molt infuenciada pel nivell d'acetilació.
Estudis sobre la funcionalitat de EVI1 en la medul·la han demostrat que l'expressió inapropiada del gen prohibeix la diferenciació terminal de les cèl·lules progenitores de medul·la a granulòcits i eritròcits, però afavoreix de manera molt important la diferenciació a cèl·lules de la línia dels megacariòcits.
És possible que la seva agressivitat com a oncoproteïna s'associï a l'efecte inhibitori que exerceix en les citoquines que controlen l'expansió cel·lular. Inhibeix també la inducció de gens pro-apoptòtics i reprimeix l'expressió de TGF-beta quan interacciona amb Smad-3.
Torna a l'índex / Torna a la Funció dels gens
Descripció de la funció de la proteïna de fusió:
La proteïna generada per la translocació t(3;21)(q26;q22) és una oncoproteïna multifuncional, relacionada amb la generació de leucèmies. Es tracta d'un factor de transcripció amb una mescla de les funcions dels gens que originen la transloació.
El primer domini Zinc finger corresponent a la proteïna EVI1, i el domini de repressió de la proteïna AML1, bloquegen la senyal del TGFβ. En condicions normals, aquesta molècula té un paper essencial en la regulació del cicle cel·lular. D'aquesta manera, en presència de la proteïna de fusió s'inhibeix la repressió del creixement i s'impedeix la regulació dels processos normals d'apoptosi.Aquest efecte d'anti-apoptosi del primer domini Zinc finger també es veu reforçat per l'acció repressora sobre JNK.
Per altra banda, el segon domini Zinc finger; té un efecte d'estimulació de la proliferació, incrementant l'activitat del factor de transcricpció AP-1 (factor de tipus activador).
També cal destacar que aquesta proteïna actua com a dominant negatiu del gen AML1. El gen AML1 wild-type s'uneix a la subunitat β del CBF, la qual cosa incrementa l'afinitat d'unió a l'ADN. De totes formes però la proteïna de fusió t´major afinitat per la subunitat β, per la qual cosa competeix amb la proteïna normal per aquest unió.D'aquesta manera s'impedeix la funció normal del gen AMLl, i per tant es bloqueja la funció de diferenciació.
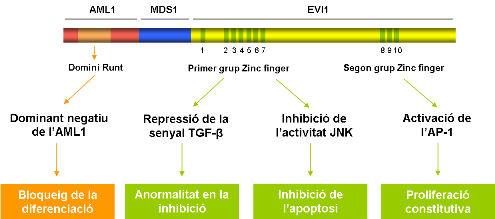
Aquesta translocació s'ha vist que està present en pacients amb la Síndrome mielodisplàsica (SMD), amb Leucèmia mieloide aguda (AML) dependent del tractament, o en la fase blàstica de desordes mieloproliferatius crònics. El producte resultant d'aquesta translocació en cooperació amb altres anormalitats genètiques, és capaç de bloquejar la diferenciació mieloide, produint un excés de proliferació cel·lular i, per tant, l'acumulació de cel·lules hematopoètiques immadures a la medul·la i a la sang perifèrica.
Torna a l'índex / Torna a la Funció dels gens
Inicialment vam fer un BLAST amb l'NCBI amb la seqüència de la proteïna proporcionada per saber a quin gen corresponia. Així, el BLAST ens va permetre fer un aliniament local de la nostra proteïna contra tot el genoma humà. També es va realitzar un BLAT per tal de veure quines regions cromosòmiques presentaven un 100% d'identitat. Posteriorment, a partir de saber el gens, vam buscar informació sobre aquests a diferents Genome Browsers, com són l'Ensembl (Ensembl release 43), el UCSC (Assembly març de 2006) i l'NCBI. Finalment i després de comparar la informació extreta de les diferents bases de dades es va decidir utilitzar com a font bàsica d'informació UCSC perquè en el cas del gen AML1 les seqüències estaven incloses a RefSeq en estat revisat. Per tal d'homogenitzar l'obtenció de les dades, la informació dels altres dos gens també s'ha extret principlament de UCSC. A l'hora de d'intentar extreure informació de les seqüències, també hem utilitzat el programa d'aliniament múltiple de seqüències ClustalW. L'anàlisi del dominis funcionals de les proteïnes s'ha fet mitjançant la base de dades Prosite.
Per obtenir la informació referent a l'homologia, hem utilitzat principalment les bases de dades Ensembl i Biomart (Ensembl 43 homology), i en el cas del gen EVI1, hem extret la informació de la base de dades HomoloGene (Release 53), de NCBI. El fet d'utilitzar Ensembl i Biomart en lloc de UCSC, com s'ha fet en el punt anterior, es deu al fet que la seqüència dels gens és la mateixa a les diferents bases de dades. D'altra banda, les dades d'Ensembl i Biomart són més completes pel que fa a homologia. Pel que fa a l'anàlisi de l'homologia de la proteïna de fusió hem realitzat un BLASTn a NCBI a partir de la seqüència nucleotídica d'aquesta proteïna.
Les dades d'expressió s'han extret de diferents bases de dades, com són Gene Sorter, UCSC, Gepis Tissue (última actualització, 16 de març de 2006), i Gene Expression Atlas (Version 2 dataset). A partir d'aquestes hem realitzat un anàlisi comparatiu, i a la vegada s'ha complementat amb informació extreta de la literatura científica. D'aquesta manera, obtenim una visió global i completa de l'expressió dels diferents gens.
Per realitzar l'apartat de la caracterització de les regions promotores, primer hem obtingut les seqüències promotores de cada transcrit a partir de la base de dades UCSC. Posteriorment hem utilitzat el programa PROMO (versió 3.0), amb la característica de cerca del màxim de dissimilaritat de les seqüències del 15%. Per acabar de completar aquest apartat, també, s'ha realitzat un programa en llenguatge Perl. La implementació Perl d'aquest algorisme es pot trobar aquí. El programa Perl s'ha fet funcionar a partir d'un arxiu amb uns factors de transcripció ja seleccionats. Veure arxiu.
Per a l'últim punt, la informació s'ha extret de la base de dades Gene Ontology (última modificació, 22 de febrer de 2007), i de diversos articles científics extrets de PubMed. A part, també s'ha fet una síntesi de la informació recopilada en tots els punts anteriors.
Torna a l'índex
En el cas del gen AML1 (RUNX1), la informació sobre la seqüència del gen, transcrits, proteïna,... s'ha extret de RefSeq a partir del UCSC. El motiu d'aquesta decisió és que els gens annotats a RefSeq han estat revisats, de manera que podem assegurar que la informació és fiable. En el cas dels dos transcrits d'aquest gen, ambdós estan en estat revisat. Al genome browser de l'Ensembl la infomació referent a aquest gen indica l'existència de 5 transcrits, el més llarg del quals correspon amb la isoforma a extreta de RefSeq. Tot i això, la resta de seqüències no mostraven concordança amb les de RefSeq. Així doncs, segons el nostre estudi podem concloure que en la transcricpió del gen AML1 es poden formar dos transcrits, degut a la presència de dos promotors diferents i que cadascun d'ells origina una proteïna de llargada diferent. Pel que fa a la proteïna que s'origina de la isoforma a segons UCSC pensem que la seva seqüència no és del tot correcta ja que hi ha la pèrdua parcial del domini Runt que és essencial per a la funció de la proteïna.
La seqüència del gen MDS1 l'hem extret del UCSC. En aquest cas, la seqüència de RefSeq es mostra en estat provisional i mostra certes discrepàncies amb la informació del UCSC, com és el cas del número d'exons. Per part de l'Ensembl, la informació existent també és diferent en el cas del exons, i a més, en la regió codificant. Degut a que aquesta seqüència codificant no comença amb ATG, hem decidit descartar-la. De totes formes, la regió de la proteïna de fusió que prové d'aquest gen coincideix en totes les seqüències de les diferents bases de dades. Les nostres conclusions pel que fa a aquest gen són que presenta un sol transcrit de 3 exons.
En el cas del gen EVI1, la informació de l'Ensembl i del UCSC és coincident pel que fa als transcrits. Per altra banda, les seqüències proteiques mostren diferències en el cas del segon transcrit. Degut a que en tots els casos hem decidit utilitzar la informació del UCSC, en aquest cas també ens basem en aquesta base de dades. D'aquesta manera, s'afirma que el gen té dos transcrits, les proteïnes dels quals només es diferencien en 1 exó sense alterar el marc de lecutra.
Finalment, després de l'anàlisi individual dels gens podem concloure que la proteïna de fusió està formada en primer lloc per la isoforma b del gen AML1 seguida d'un fragment del gen MDS1 i finalment per el segon transcrit del gen EVI1 però amb la seqüència codificant des de l'inici de l'exó dos.
En l'aparat d'estudi d'homologia, tan pel AML1 com pel MDS1 hem utilitzat la base de dades Ensembl, però en el cas del gen EVI1 aquest informació no hi era present. Per aquest motiu, hem buscat aquesta informació a la base de dades HomoloGene (NCBI), la qual no ofereix exactament la mateixa estructura d'informació. De totes maneres, en tots els casos hem aconseguit descriure l'homologia de cada un dels gens de la proteïna de fusió. En tots els casos, podem afirmar que els gens mantenen una conservació elevada amb aquelles espècies evolutivament properes. Pel que fa a les espècies més llunyanes també hi ha un percentatge d'homologia considerable, cosa que ens fa pensar que aquests gens tenen una funció important en el desenvolupament dels organismes. Fent referència a la proteïna de fusió, la conclusió a la qual s'arriba després d'analitzar la informació és que probablement no s'han realitzat suficients estudis en altres espècies com per determinar si la translocació també es produeix. Per aquest motiu tots els resultats obtinguts són predits.
L'anàlisi de l'expressió s'ha realitzat després de recollir dades de diferents fonts per tal de tenir una visió global. Els resultats han estat que en el cas del gen AML1 hem pogut concloure de forma detallada els teixits en els quals s'expressa: tim i cèl·lules hematopoètiques. Per als altres dos gens, l'afirmació no pot ser tan clara degut a la manca d'informació sobre la funció i localització d'aquests gens. Tot i això, els nostres resultats só que el gen MDS1 s'expressa a cor, cervell i fetge i el gen EVI1 s'expressa a pulmó, ronyons, pàncrees i òrgans genitals. En el cas de la proteïna de fusió i, tenint en compte que causa una malaltia a les cèl·lules hematopoètiques, creiem que s'expressa precisament en aquest tipus cel·lular.
Per tal d'obtenir conclusions sòlides en la caracterització de les regions promotores, cal considerar que les dues vies utilitzades parteixen de matrius, factors i número de posicions diferents. Per aquest motiu, no ha sigut possible la comparació global dels resultats dels dos programes. Així doncs, mitjançant cadascun d'ells hem obtingut de forma general una llista diferent de factors de transcripció per a cadascuna de les regions promotores. D'altra banda, però, s'ha obtingut coincidències per alguns dels factors a partir dels dos programes. En el cas de la proteïna de fusió considerem que els factors de transcripció que s'uneixen a la seva regió promotora són els mateixos que en cas de la isoforma b de AML1, ja que és el gen que es troba en posició inicial en la proteïna de fusió.
En l'últim punt es realitza una anàlisi de la funció dels diferents gens. Les dades s'han extret de Gene Ontology i de diversos articles científics per tal de poder complementar la informació. Així doncs, podem concloure que la funció de la proteïna de fusió es defineix a partir d'una mescla de les activitats de cadascun dels gens. És a dir, mostra una activitat com a factor de transcripció amb efecte dominant negatiu sobre l'acció normal del gen AML1. D'altra banda, també mostra funcions derivades dels dominis Zinc finger de EVI1. De forma independent, es conclou que la proteïna corresponent al gen AML1 actua com a regulador de la transcirpció de gens implicats en el desenvolupament i la diferenciació cel·lular. En el cas de MDS1 es pot dir que actua com a factor de transcripció peró se'n desconeix la funció concreta. EVI1 &ecute;s també un factor de transcripció important en processos d'organogènesi, morfogènesi, diferenciació i proliferació cel%middot;lular.
La conclusió general de l'estudi és que la translocació que genera l'AME es dóna en pacients que pateixen leucèmia mieloide aguda, síndrome mielodisplàsica dependent de teràpia o leucèmia mieloide crònica amb crisi blástica. La translocació apareix a partir de la fusió de fragments determinats del cromosoma 21 contenint el gen AML1 i del cromosoma 3 a la regió on es troben els gens MDS1 i EVI1. Cal destacar també que degut a que la regió promotora de l'AME és la mateixa que la del gen AML1, la proteïna de fusió s'expressa en els mateixos tipus cel·lulars que AML1. La proteïna de fusió produeix una sèrie d'efectes que porten a l'empitjorament de l'estat dels pacients que pateixen les malaties esmentades anteriorment.
Torna a l'índex
- Buonamici S, Chakraborty S, Senyuk V, Nucifora G. The role of EVI1 in normal and leukemic cells. Blood Cells, Molecules, and Diseases. 2003; 31:206-212.
- Cammenga J, Niebuhr B, Horn S, Bergholz U, Putz G, Buchholz F, Lohler J, and Stocking C.RUNX1 DNA-Binding Mutants, Associated with Minimally Differentiated Acute Myelogenous Leukemia,Disrupt Myeloid Differentiation. Cancer Res. 2007; 67(2):537-45
- Cuenco G, Nucifora G, and Ren R. Human AML1 MDS1 EVI1 fusion protein induces an acute myelogenous leukemia (AML) in mice: A model for human AML. PNAS. 2000; 97:1760-1765
- Cuenco G, Ren R.. Both AML1 and EVI1 oncogenic components are required for the cooperation of AML1/MDS1/EVI1 with BCR/ABL in the induction of acute myelogenous leukemia in mice. Oncogene. 2004; 23:569-579.
- Helbling D, Mueller B, Timchenko N, Hagemeijer A, Jotterand M, Meyer-Monard S, Lister A, Rowley J, Huegli B, Fey M, Pabst T. The leukemic fusion gene AML1-MDS1-EVI1 supresses CEBPA in acute myeloid leukemia by activation of Calreticulin. PNAS. 2004; 101(36):13312-13317.
- Mitani K. Molecular mechanisms of leukemogenesis by AML1/EVI-1. Oncogene. 2004; 23:4263-4269.
- Mitani K, Ogawa S, Tanaka S, Miyoshi H, Kurokawa M, Mano H, Yazaki Y, Ohki M and Hirai H. Generation of the AML 1- EVI- 1 fusion gene in the t(3;21)(q26;q22) causes blastic crisis in chronic myelocytic leukemia. The EMBO Journal. 1994; 13(3):504-510
- Mikhail F, Sinha K.K, Saunthararajah Y,Nucifora G. Normal and Transforming Functions of RUNX1: A Perspective. Journal of Cellular Physiology. 2006; 207:582-593
- Nucifora G, Laricchia-Robbio L, Senyuk V. EVI1 and hematopoietic disorders: History and perspectives. Gene. 2006; 368:1-11
- Tanaka T, Mitani K, Kurokawa M, Ogawa S, Tanaka K, Nishida J, Yazaki Y, Shibata Y, Hirai H. Dual Functions of the AML1/Evi-1 Chimeric Protein in the Mechanism of Leukemogenesis in t(3;21) Leukemias. Molecular and Cellular Biology. 1995; 15:2383-2392.
- Yin C.C, Cortes J, Barkoh B, Hayes K, Kantarjian H, Jones D. t(3;21)(q26;q22) in Myeloid Leukemia An Aggressive Syndrome of Blast Transformation Associated with Hydroxyurea or Antimetabolite Therapy. Cancer. 2006; 106(8):1730-1738
Torna a l'índex

.png)
.png)










