
El nostre treball es basa en l'estudi de la proteïna de fusió PRR5-ARHGAP8,aquesta està formada per dues protïnes consecutives del cromosoma 22. En concret la proteïna estudiada conté a la regió N-terminal trobem 12 aminoàcids provinents del PRR5, seguidament trobem 24 aminoàcids codificats en la regió upstream de l'inici de traducció del ARHGAP8 i finalment la isoforma 2 del ARHGAP8. Per aquest motiu hem realitzat tot el treball analitzant cada una de les proteïnes per separat i intentant relacionar els resultats obtinguts amb la proteïna de fusió.
L'estudi en paral·lel de l'expressió i funció del PRR5 i ARHGAP8 no han permès obtenir uns resultats definitius sobre el paper de la proteïna de fusió a nivell cel·lular i tissular. Tot i així recollint tota la informació obtinguda hem pogut hipotetitzar una possible pauta d'expressió i possible funció d'aquesta. Actualment, s'estan duent a temre diversos estudis per tal de resoldre aquests interrogants sobre la proteïna PPR5-ARHGAP8.
La proteïna PRR5-ARHGAP8 es situa al braç llarg del cromosoma 22 en la posició 13.3 (22q13.3). Observant la figura 1, veiem com la nostra proteïna de fusió (LOC553158) està formada per dues proteïnes consecutives en aquest cromosoma.

Figura 1:Extreta del USCS genome browse
En la figura 1 també es pot veure que la proteïna està formada per 14 exons. La comparació del exons presents en el mRNA i en la proteïn ens permet saber que dels 14 exons, 12 són codificants. En el següent link es mostren la seqüència de la proteïna amb els exons marcats.
seqüènciaMitjançant el Ensembl hem vist que aquesta proteïna té 11 transcrits associats. En la taula inferior es mostra el llistat d'aquests transcrits amb una representació esquemàtica de la seva estructura genòmica i la proteïn que se n'obté al traduir-lo.
| Identificador | Esquema | ||
| 1 | ENST00000336985 |  |
PRR5 isoforma 1 |
| 2 | ENST00000006251 |  |
PRR5 isoforma 2 |
| 3 | ENST00000389774 |  |
ARHGAP8 isoforma 1 |
| 4 | ENST00000389775 |  |
ARHGAP8 isoforma 1 |
| 5 | ENST00000389773 |  |
ARHGAP8 isoforma 2 |
| 6 | ENST00000389772 |  |
ARHGAP8 isoforma 2 |
| 7 | ENST00000336963 |  |
Explicació |
| 8 | ENST00000336995 |  |
Explicació |
| 9 | ENST00000352766 |  |
Explicació |
| 10 | ENST00000356099 |  |
No informativa |
| 11 | ENST00000361473 |  |
No informativa |
Taula 1:Transcrits del Ensembl
El gen PRR5 codifica per una proteïna amb un domini ric en prolines. Existeixen tres isoformes de la proteïna, les quals són generades per splicing alternatiu.
Per tal d'estudiar les diferències; en les tres isoformes de la proteïna, vam realitzar un alineament de les seqüències amb el programa ClustalW.
&bull Alineament. En l'alineament les isoformes es col·loquen en el següent ordre: isoforma 1, 3 i 2. Aquest alineament ens va permetre veure que les tres isoformes només divergien en la part N-terminal.
El mRNA de la isoforma 1 té 8 exons, tots codificants. La traducció s'inicia en el ATG del primer exó marcat en lila. Comparant les seqüències de mRNA de les isoformes 1 i 2 podem observar que en el primer exó de la isoforma 1, 27 nucleòtids més enllà del principi de de traducció d'aquesta, trobem representat l'exó 2 de la isoforma 2. Això ens fa pensar que la transcripció de les dues isoformes s'inicia en punts diferents de la seqüència genòmica.
&bull mRNA isoforma 1 PRR5La isoforma 2 té una llargada de 2,017 bp i tradueix per 379 aminoàcids. Té 9 exóns, dels quals 8 són codificants. El primer exó d'aquesta isoforma no és codificant. La traducció s'inicia en el ATG situat en el segon exó, la qual s'inicia a 11 nucleòtids de l'inici de l'exó dos. Això es pot veure perquè el primer aminoàcid de la proteïna de la isoforma 2 correspon al desè aminoàcid de la isoforma 1.
&bull mRNA isoforma 2 PRR5El mRNA de la isoforma 3 té 8 exons, dels quals 5 són codificants. Excepte l'exó 1, la resta d'exons són iguals als exons de la isoforma 1. Comparant-la amb la isoforma 2 veiem que hi ha concordància de seqüències a partir de l'exó 2 de la isoforma 3 i l'exó 3 de la isoforma 2. La traducció de la proteïna s'incia al ATG marcat en lila, el qual es situa en l'exó 4 del mRNA. La proteïna de la isoforma 3 és la més curta de les tres isoformes. Es diferencia de les altres dues en què el seu inici traducció es situa en la meitat del quart exó codificant de les altres dues isoformes. La resta de la proteïna és exactament igual a les altre dues isoformes.
&bull mRNA isoforma 3 PRR5En el mRNA de la isoforma 1 hi trobem tres ATG que podrien iniciar la traducció de proteïna; un corresponent a aquesta isoforma i els altre dos a les altres dues isoformes respectivament. Això ens fa pensar que les isoformes 2 i 3 es podrien produir per un splicing alternatiu d'aquest mRNA. Tot i que aquest splicing es donaria en exons codificants, no afectaria a la pauta de lectura.
&bull Inici de traducció de les tres isoformesComparant les seqüències proteiques de les 3 isoformes de PRR5 amb la seqüència de la nostre proteïna de fusió veiem que els 12 primers aminoàcids de la PRR5-ARHGAP8 són presents en les tres isoformes. En concret es troben en el quart exó codificant de les isoformes 1 i 2, i el primer de la isoforma 3. Aquesta correspondència es mané en el mRNA.
Existeixen 2 isoformes de la proteina ARHGAP8, les quals es generen per splicing alternatiu.
Per tal d'estudiar les diferències en les dues isoformes de la proteïna, vam realitzar un alineament de les seqüències amb el programa ClustalW.
&bull Alineament. En l'alineament les isoformes es col·loquen en el següent ordre: isoforma 1 i 2. Aquest alineament ens ha permès veure que les dues isoformes es diferencien per un exó, present en la isoforma 1 i absent en la 2.
La isoforma 1 de ARHGAP8 té 12 exons, dels quals tots són codificants.
&bull mRNA isoforma 1 ARHGAP8Per altra banda la isoforma 2 té 11 exons codificants.
&bull mRNA isoforma 2 ARHGAP8Les dues isoformes difereixen en la presencia d'un exó, en concret l'exó 4 de la isoforma 1 és absent en la isoforma 2. Aquest splicing tot i produir-se en la regió codificant, no afecte a la pauta de lectura.
Comparant les seqüències proteiques de les dues isoformes del ARHGAP8 i la de la nostre proteïna de fusió podem afirmar que tota la isoforma 2 hi és present.
Transcrit 7
Aquest transcrit conté 11 exons, els quals tots són codificants. &bull mRNA del transcrit 7La proteïna associada a aquest transcrit conté la isforma 2 del ARHGAP8, però el seu primer exó està format per 29 aminoàcids seguits pel primer exó del ARHGAP8 . A partir de l'observació del mRNA d'aquest transcrit hem comprovat que aquests aminoàcids davanters provenen de la regió upstream no codificant del primer exó de la isoforma 1.
Transcrit 8
Aquest transcrit conté 20 exons, tots codificants. &bull mRNA del transcrit 8Al inici hi ha els 7 primers exons de la isoforma 1 del PRR5 , per tant també hi és present l'inici de la nostre proteïna de fusió. Seguidament observem la regió no codificant de la seqüència upstream del primer exó de ARHGAP8 i la isoforma 1 del ARHGAP8 amb un exó intercalant entre l'exó 4 i el 5 del mRNA.
Transcrit 9
Aquest transcrit conté 17 exons, tots codificants. &bull mRNA del transcrit 9Al inici hi ha els 7 primers exons de la isoforma 1 del PRR5 , per tant també hi és present l'inici de la nostre proteïna de fusió. Seguidament trobem la regió no codificant anterior al primer exó de la proteina ARHGAP8. Finalment trobem la proteïna ARHGAP8, poden haver dues opcions: o es tracta de la isoforma 1 sense l'exó 3 i 4 o bé la isoforma 2 sense l'exó 3.
Gràcies a tota la informació obtinguda amb l'estudi dels transcrits, hem pogut esbrinar com es forma la proteïna de fusió.
&bull proteïna de fusió PRR5-ARHGAP8 En la primera imatge veiem la proteïna dividida en 3 parts. La primera, marcada amb groc, correspon a la regió codificada pel PRR5 (en concret el quart de les isoformes 1 i 2, i el primer de la isoforma 3). La segona regió, marcada amb verd, correspon a la seqüència upstream no codificant del primer exó de la proteïna ARHGAP8. Finalment l'última regió, marcada amb taronja, correspon a la isoforma 2 de la proteïna ARHGAP8.
Seguidament si ens fixem en el mRNA també el veiem dividit en tres parts, coincidents amb les esmentades anteriorment en la proteïna. Tot i així, volem destacar que la primera seqüència de mRNA marcada correspon als exons 2, 3 i 4 de les isoformes 1 i 3 del PRR5; i els exons 3, 4 i 5 de la isoforma 2.
Més endavant en l'apartat de discussió es parlarà sobre les possibles explicacions de la formació de la proteïna de fusió.
Per tal de trobar si la nostra proteïna de fusió es trobava conservada en altres espècies hem accedit a diferents bases de dades, com Ensembl, el UCSC i el NCBI. Com no hem trobat cap ortòleg de la nostre proteïna de fusió hem buscat els ortòlegs de cada una de les dues proteïnes que la formen. En les taules inferiors es mostren els resultats obtinguts, especificant l'espècie on s'ha trobat l'ortòleg, el nom del gen i el tant per cent de conservació que té a nivell de proteïna i de DNA..
Cal dir que els valors de conservació han estat mesurats alineant la seqüència de la isoforma 1 de la proteïna humana PRR5 amb la seva homòloga en l'altre espècie.
| Espècie | Nom del gen | Conservació en proteïna (%) | Conservació en DNA (%) | Link al Blast |
| Mus musculus | Arhgap8 | 89.1 | 84.9 |  |
| Rattus norvegicus | Arhgap8_predicted | 89.9 | 85.8 |  |
| Gallus gallus | PRR5 | 80.5 | 74.0 |  |
Taula 2: Ortòlegs del PRR5
Quan vam realitzar la cerca d'ortòlegs d'aquesta proteïna ens va cridar l'atenció que en el ratolí i la rata els ortòlegs del PRR5 s'anomenessin ARHGAP8, així que vam buscar informació al respecte. Les proteïnes esmentades anteriorment es van identificar l'any 2003 [3] i se'ls hi va otorgar el nom de ARHGAP8, cal destacar que llavors encara no s'havia identificat la proteïna PRR5. Ens estudis posteriors [1], al identificar-se la proteïna PRR5 es van adonar de que en realitat corresponien a ortòlegs d'aquesta última.
Cal dir que els valors de conservació han estat mesurats alineant la seqüència de la isoforma 2 de la proteïna humana ARHGAP8 amb la seva homòloga en l'altre espècie.
Taula 3: Ortòlegs del ARHGAP8
Per tal de mostrar des d'un punt de vista més evolutiu les relacions entre els diferents ortòlegs i la proteïna humana hem construit un arbre filogenètic de cada una de les proteïnes. La figura 2 correspon a l'arbre filogenètic de la proteïna PRR5. Tal i com podem observar l'ortòleg més allunyat és el del Gallus gallus. Per altra banda podem veure que els mamífers comparteixen un nexe i que dins d'aquest grup les proteïnes de la rata i el ratolí són més properes entre elles que amb les dues isoformes humanes. Aquests resultats concorden amb la història evolutiva de les espècies implicades.

Figura 2: Arbre filogenètic dels diferents ortòlegs de la proteïna PRR5
La figura 3 mostra l'arbre filogenètic de la proteïna ARHGAP8. Els resultats són força semblants als anteriors. Amb la diferència que en aquest cas veiem com la proteïna del gos està evolutivament més allunyada del les proteïnes humanes en comparació a les proteïnes dels rossegadors.

Figura 3: Arbre filogenètic dels diferents ortòlegs de la proteïna ARHGAP8
L'objectiu d'aquest apartat és determinar en quins tipus cel·lulars i texits s'expressa la nostre proteïna de fusió. Per estudiar això hem utilitzat dades del UCSC, en concret els microarrays. Hem preferit inicialment estudiar l'expressió en les dues proteïnes per separat i després comparar-les amb les dades d'expressió presents en l'entrada de la nostra proteïna al USCS. Alhora de classificar l'intensitat de l'expressió, hem dividit les dades obtingudes en dos grups: teixits o tipus cel·lulars on hi ha sobreexpressió i teixits o tipus cel·lulars on hi ha una expressió més moderada. Cal tenir en compte que aquestes dades provenen de proves experimentals, per tant són susceptibles a petites discrepàncies amb altres fonts d'informació.
Taula 4: Dades d'expressió de les proteï,nes PRR5 i ARHGAP8
En la taula anterior hi ha inclosa la imatge de l'expressió dels microarrays el qual s'ha extret tota la informació. Podem observar-hi dos colors: el vermell que ens indica molta expressió de la proteïna i el verd poca expressió d'aquesta. Al realitzar la taula només hem agafat els teixits o tipus cel·lulars que contenien els ratios de color vermell. Al observar diferents intensitats del vermell, hem dividit aquest en dos grups, corresponents a vermell brillant (sobreexpressió) i vermell més apagat (expressió moderada).
En alguns articles extrets a partir de la base de dades del OMIM [1] [5], vam observar que descrivien una sobreexpressió de la proteïna PRR5 en ronyó i fetge i que observaven nivells baixos en cervell, melsa, placenta i testicles. Per altra banda, describien una sobreexpressió de ARHGAP8 en ronyó i placenta i un expressió moderada en colon, múscul esquelètic, intestí prim, estómag i testicles, dades obtingudes mitjançant Northern Blot. Aquests resultats observats no són iguals als presentats a la taula anterior, per tant podriem dir que els experiments generats amb microarrays podrien haver patit errors experimentals.
Per tal de caracteritzar la nostra seqüència promotora i identificar els factors de transcripció que s'hi uneixen, hem realitzat dos estudis paral·lels:
Primerament vam extreure la nostra seqüència promotora de la nostra proteïna de fusió PRR5-ARHGAP8 formada per 1000 parells de bases upstream del lloc de començament de la transcripció i 100 parells de bases downstream del TSS.
1- Utilitzant el servidor web del programa PROMO vam predir els factors de transcripció possibles i els llocs d'unió d'aquests en la nostra seqüència promotora, però vam observar que s'obtenien un nombre molt elevat de possibles regions i factors. Per això, vam creure necessari de realitzar un filtratge dels resultats obtinguts per aconseguir reduir falsos positiu.
Aquest filtratge el vam realitzar utilitzant uns valors de dissimilaritat del 10%, que ens indicava el grau de no concordança entre la seqüència i la matriu de pesos utilitzada per predir la regió d'unió al factor de transcripció corresponent, i també agafant únicament els factors de transcripció amb un RE equally de com a màxim 0,08. El RE equally fa referència a la probabilitat de trobar una regió d'unió del factor de transcripció, amb un score superior al obtingut, en una seqüència d'igual llargada que la nostra seqüència promotora però amb una proporció del 25% per cada nucleòtid.
Si tinguessim una seqüència promotora més llarga seria més adequat utilitzar el RE query per realitzar el filtratge del nombre de factors de transcripció que s'hi uneixen. El RE query es basa en la mteixa comparació que el RE equally, però la seqüència que s'utilitza té la mateixa proporció de nucleòtids que la seqüència promotora testada. Com que el nostre promotor és molt curt és indiferent utilitzar el RE equally o RE query.
Aqui podem observar la taula obtinguda després del filtratge dels resultats.
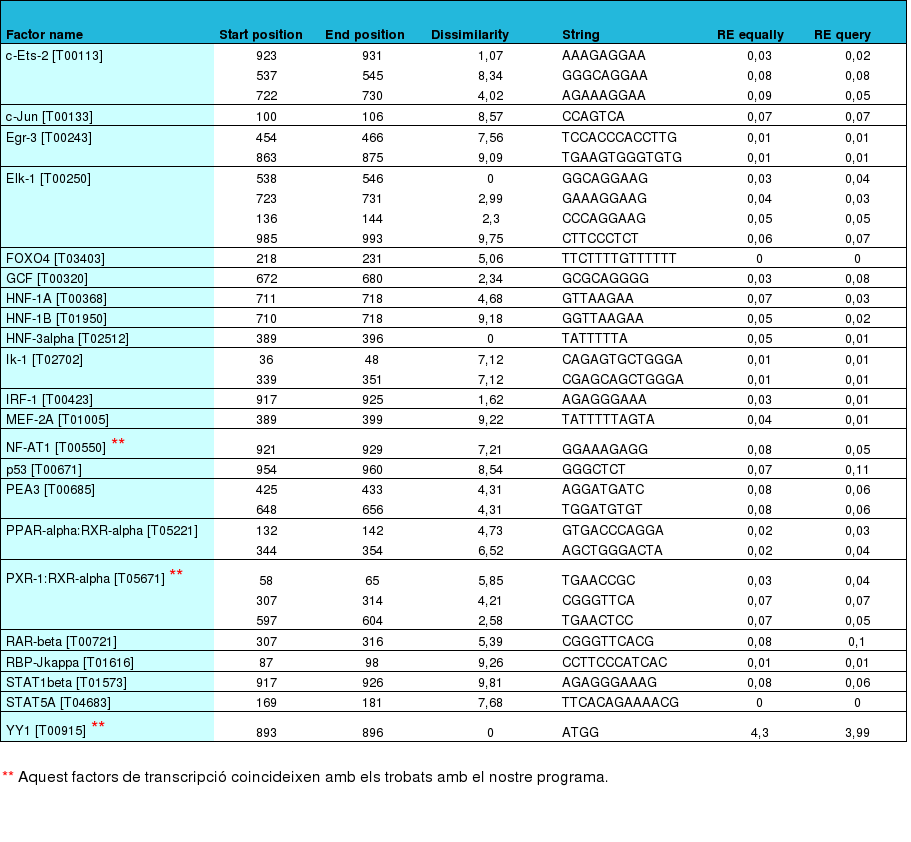
Figura 4: Els millors factors de transcripció que s'uneixen a la nostra seqüència promotora.
En aquesta taula podem observar els 22 factors de transcripció millors que s'uneixen en la nostra seqüència promotora, on tots ells presenten un RE equally igual o inferior a 0,08. També hi podem observar la seqüència concreta a la qual s'unirà el factor de transcripció i l'inici i final d'aquesta.
Per altra banda, observem 3 factors de transcripció amb dos asteriscs, que fan referència als FT que coincideixen amb els trobats mitjançant el nostre programa perl. Cal dir, que l'últim FT de la taula, no presenta un RE equally inferior a 0,08, però hem cregut convenient d'introduir-lo ja que és un d'aquests que coincideix amb l'altre estudi realitzat paral·lelament.
2- Mitjançant un programa perl realitzat per nosaltres mateixes, vam realitzar l'estudi de la cerca dels factors de transcripció que s'uneixen a la nostra seqüència promotora introduïda. En aquest estudi només es testaven 13 factors de transcripció preestablerts pel nostre professorat.
Aqui podem observar els resultats que hem obtingut al avaluar els 13 factors de transcripció donats mitjançant el programa perl que hem dissenyat.
| Factors de transcripció | SCORE | Posició | p-value |
| AP-1 [T0029] | -995.55957008924 | 495 | 0.6 |
| AR [T00040] | 2.26848080557308 | 59 | 0.7 |
| c-Myc [T00140] | -996.00059201113 | 105 | 0.85 |
| NF-AT1 [T00550] | 3.46570122503359 | 921 | 0.09 |
| NF-kappaB [T00590] | -995.8568106973 | 406 | 0.7 |
| SRF [T00764] | -1994.71410727054 | 530 | 0.95 |
| YY1 [T00915] | 2.4876900746637 | 893 | 0.83 |
| RXR-alpha [T01345] | 3.18470724970563 | 58 | 0.16 |
| HIF-1 [T01609] | -996.188442956152 | 101 | 0.98 |
| AhR [T01795] | 3.3337743891868 | 492 | 0.13 |
| PU.1 [T02068] | 3.208893204546 | 138 | 0.22 |
| HNF-4 [T02758] | -996.160202203172 | 59 | 0.95 |
| NRSF [T06124] | -996.28469175131 | 185 | 0.64 |
Taula 5: Resultats dels 13 factors de transcripció donats.
Observant la taula anterior, podem fixar-nos primerament amb els scores obtinguts mitjançant el programa perl. Amb aquests resultats ja podem descartar els factor de transcripció amb un score negatiu, ja que és molt poc provable que aquests FT s'uneixin a la regió promotora de la nostra proteïna de fusió. El valor negatiu ens indica que en la nostra seqüència promotora no trobem la regió específica on s'uneix aquell FT. També podem observar que aquests 7 FT amb valors negatius contenen, la majoria, un p-value molt gran, ratificant doncs la conclusió expressada anteriorment.
Per altra banda observem 6 FT que presenten un score positiu, aquests ja els podem considerar FT que molt provablement s'uniran a la nostra seqüència promotora de la proteïna de fusió. Aquests FT presenten, la majoria, un p-value més petit, això ens indica que la seqüència que uneix aquest FT no s'observa tan freqüentment a l'atzar.
Comparant els dos estudis realitzats podem observar que només hi han 3 factors de transcripció que concordin en quan a la primer posició de la seqüència que s'uneix el FT. Cal dir que dos d'ells el NF-AT1 i el RXR-alpha presenten un RE equally de 0,08 i 0,03, un score de 3,46 i 3,18 i un p-value de 0,09 i 0,16 respectivament. Això ens indica que són FT molt favorables a unir-se a una regió concreta de la seqüència promotora. Per altra banda l'altre FT coincident, el YY1, presenta un RE equally de 4,3, un score de 2,48 i un p-value de 0,83, cosa que ens indica que no seria un FT tan favorable per la unió a la seqüència promotora de la nostra proteïna de fusió.
Finalment volem destacar que la no coincidència total dels dos estudis realitzats paral·lelament és del tot esperable ja que el programa PROMO, el qual és molt més complex, realitza moltes més operacions a l'hora de calcular els FT òptims en la unió a la seqüència promotora. Per tant els resultats obtinguts amb el PROMO són molt més fiables que els obtinguts mitjançant el nostre programa generat.
L'estudi de la funció de les proteïnes que formen part de la proteïna de fusió, s'ha realitzat a partir de la informació obtinguda pel Gene Ontology i d'articles extrets del Pubmed.[1][2][4][5]
En primer lloc parlarem de la informació obtinguda del Gene Ontology, dividint-la en les tres nivells informatius que utilitza aquest servidor: funció molecular, procés biològic i components cel·lulars.
| Funció molecular | Procé biològic | Component cel·lular | |
| PRR5 | --- | GO:0007165 | GO:0005622 |
| ARHGAP8 | GO:0005100 | GO:0007165 | GO:0005622 |
Taula 6: Funcions de les proteïnes trobades al Gen Ontology
&bull Funció molecular
GO:0005100 Rho GTPasa activator activity (GAP)
Incrementa l'activitat GTPasa de les proteïnes GTPasas de la família Rho, fent augmentar el seu ràtio d'hidròlisi de GTP.
&bull Procés biològic
GO:0007165 Transducció de senyals
Mitjançant la interacció de la proteïna amb un receptor específic s'estimula una cascada de senyalització, causant un canvi en els nivells i l'activitat d'un missatger secundari o d'una nova diana downstream de la cascada. Això provoca en últim terme un canvi en la funció cel·lular.
&bull Component cel·lular
GO:0005622 Intracel·lular
A continuació aprofundirem en les funcions de les proteïnes estudiades a partir de la informació extreta dels articles.
Referent el PRR5 podem dir que no té cap domini associat, cosa que ha dificultat assignar-li una funció molecular específica. Tot i així, en l'estructura d'aquesta proteïna es destaca una regió rica en prolines.
Actualment no es coneix la seva funció dins la cèl·lula, però estudis recents la relacionen com a un potencial supressor de tumors, en concret en tumors de pit. Aquesta relació prové de la demostració de que en les cèl·lules d'aquests tumors s'observa menys expressió de PRR5 que en les cèl·lules sanes de pit.
Per altra banda altres estudis han demostrat que aquesta proteïna està sobreexpressada en tumors colorectals. Aquestes controvèrsies fan que el paper cel·lular del PRR5 encara es trobi en estudi.
La funció de la proteïna ARHGAP8 està força més documentada que l'anterior.
ARHGAP8 és un nou membre de la família de proteïnes RHOGAP, per tant és una proteïna activadora de les GTPases de la família Rho (Rho GTPasa activator activity, GAP). Està formada per tres dominis: un primer domini en la regió N-terminal , Sec14p-like (BCH lipid-binding); un segon domini SH3 i finalment el domini RhoGap en la regió C-terminal.
&bull Sec-14p-like és un domini que es troba en proteïnes regulades per lípids, com les RhoGAPS, RhoGEFs i NF1. Aquest domini permet que formi complexes homofílics i heterofílics amb proteïnes que també posseeixen aquest domini.
&bull El domini SH3 és força reduït, està format per la següent seqüència d'aminoàcids : PPRPPLP. En general interacciona amb regions riques en prolina o aminoàcids hidrofòbics. Alguns estudis indiquen que quinases de la familia-SRC poden estar relacionades amb l'activació de RHO GTPases interaccionant amb aquest domini.
&bull El domini RhoGap incrementa l'activitat GTPasa de les proteïnes Rho GTPases, com a conseqüència s'hidrolitza el seu GTP unit fent que s'inactivin. Les proteïnes RhoGTPases formen part de la família Ras. Participen en la reorganització de l'esquelet d'actina i en l'activació de cascades de quinases que regulen l'expressió gènica i controlen la progressió del cicle cel·lular i la proliferació cel·lular. S'ha de dir que esta acceptat que una activació constitutiva d'aquesta GTPasa pot induir la transformació de cèl·lules normals cap a tumorals.
Tenint en compte la funció del domini RhoGap, els resultats respecte l'expressió del ARHGAP8 en cèl·lules tumorals de pit i de colon semblen contradictoris. En aquestes cèl·lules s'ha observat una sobreexpressió de la proteïna, fet que comportaria una inactivació de les Rho GTPases presents, per tant una no transformació de les cèl·lules en tumorals. Aquestes incongruències fan que no es sàpiga del cert la funció del ARHGAP8 en processos tumorals, tot i que s'hipotetitza que la sobreexpressió seria una manera de controlar l'excés d'activitat GTPasa en cèl·lules tumorals.
En resum podem dir que la proteïna ARHGAP8, bàsicament està involucrada en mobilitat cel·lular i en la proliferació cel·lular.
Actualment no es coneix quina és la funció de la proteïna PRR5-ARHGAP8. Tot i així, a partir de la informació funcional de les proteïnes que la formen i tenint en compte que la seqüència majoritària de la proteïna de fusió correspon a la ARHGAP8, podem hipotetitzar que la proteïna fusionada possiblement tingui la mateixa funció que l'ARHGAP8.
A continuació s'explicaràn tots els passos realitzants durant l'elaboració del treball. Aquests els hem dividit pels mateixos punts en què es presenten els nostres resultats.
• Identificació de la proteïna
Primer de tot vam baixar-nos la seqüència corresponent a la nostra proteïna. Per tal d'identificar-la vam accedir al NCBI per realitzat un BLASTp. Gràcies a això vam esbrinar que es tractava de la proteïna de fusió PRR5-ARHGAP8. Posteriorment per caracteritzar la seva estructura genòmica vam utilitzat informació del Ensemble i del USCS.
•Estructura genòmica
A partir del USCS Genome Browser vam poder determinar les característiques més bàsiques de la nostre proteïna, tals com en quin cromosoma es troba i la seva posició específica. Per altra banda gràcies a la representació gràfica de la seva situació dins del cromosoma vam esbrinar que estava formada per dues proteïnes consecutives, les qual presentaven diferents isoformes. Linkant dins del gràfic vam observar que existeixen dues isoformes del ARHGAP8 i tres del PRR5 que podrien participar en la formació de la proteïna de fusió.
Posteriorment realitzàrem un estudi de les isoformes per identificar-ne les diferències, mitjançant alineaments amb el programa clustalW.
Per tal d'escollir els transcrits associats a la nostra proteïna teniem dues possibles fonts: UCSC o Ensembl. Ens vam decantar pels transcrits de la base de dades de l'Ensembl, ja que és una font d'informació verificada, a diferència de la base de dades del UCSC on la informació prové d'experiments, la qual cosa podria aportar informació errònea com a conseqüència d'errors experimentals.
A partir de l'entrada de la nostre proteïna de fusió en l'Ensembl, ENSG00000186654,vam observar que exisiten 11 transcrits diferents. A continuació vam obtenir els mRNA i les proteïnes de cada un d'ells. La comparació d'aquests amb les seqüències de les diferents isoformes de cada proteïna, obtingudes del USCS, ens permetéren esbrinar a que feia referència cada transcrit.
Finalment, la comparació de tots els transcrits analitzats ens ha permès entendre d'on provenia cada part de la proteïna de fusió i donar una explicació de la seva formació.
• Conservació en altres espècies
Per tal de buscar ortòlegs de la proteïna de fusió i de les proteïnes que la formen vam buscar informació en diferents bases de dades però no vam tenir massa èxit.
Inicialment vam buscar ortòlegs a partir de l'Ensembl, però no en vam trobar. Llavors vam decidir buscar ortòlegs pel Biomart. La cerca la realitzàrem a partir de la base de dades Compara homology, utilitzant com a filtre, que nomès comparés els gens humans del cromosoma 22. Es van obtenir 10 resultats, però cap d'ells era ni la proteïna de fusió ni cap de les dues que la formen. Finalment vam buscar ortòlegs a la base de dades del NCBI. Primerament vam accedir al Map Viwer a partir de la pàgina del gen del PRR5. Un cop allà vam accedir a la informació d'homologia d'aquest gen, obtenint els diferents ortòlegs existents. En aquesta pàgina vam trobar informació dels gens de les altres espècies, alineaments i percentatges de conservaciò. Posteriorment aquest procediment el vam repatir per la proteïna ARHGAP8 i per la proteïna de fusió. Els resultats de la cerca apareixen en les taules 2 i 3 dins l'apartat de resultats.
Per la realització dels arbres filogenètics, inicialment vam baixar-nos les seqüències proteiques de les isoformes del PRR5 i dels seus ortòlegs des del NCBI. Seguidament vam realitzar un clustalW per alinear-les. El resultat d'aquest alineament el vam obrir amb el Bioedit i el vam guardar en format fasta. Finalment vam obrir el programa MEGA, vam passar el fitxer amb l'alineament fasta a format mega i vam realitzar l'abre filogenètic per UPGMA. Aquests passos els vam repetir per la proteïna ARHGAP8.
• Caracterització de l'expressió del gen
Per tal d'observar en quins teixits o tipus cel·lulars s'expressa la nostra proteïna de fusió i totes les isoformes de les proteïnes PRR5 i ARHGAP8 vam utilitzar la base de dades del UCSC.
Primerament vam entrar dins la pàgina que ens descriu la nostra proteïna de fusió: LOC553158, vam observar que a la part inferior d'aquesta hi havia un apartat que ens informava de l'expressió amb microarrays de la nostra proteïna de fusió. Al observar diferents microarrays d'expressió, vam decidir utilitzar el GNF Expression Atlas 2 Data from U133A and GNF1H Chips per tal d'unificar la font informativa utilitzada en els tres casos, generant doncs una taula informativa (Taula 4).
Posteriorment vam realitzar el mateix estudi amb les isoformes del PRR5, adonant-nos que tenien la mateixa l'expressió. Finalment ho vam realitzar amb la proteïna del ARHGAP8, obtenint una expressió igual en les dues isoformes.
• Caracterització de la regió promotora del gen
Per tal de realitzar l'estudi de la regió promotora de la nostra proteïna de fusió i obtenir els factors de transcripció que s'hi uneixen, primer de tot vam extreure la seqüència promotora mitjançant la base de dades del UCSC, tot agafant 1000 parells de bases upstream de l'inici de traducció i 100 parells de bases downstream.
Seguidament, utilitzàrem el programa PROMO per tal d'obtenir els factors de transcripció que s'uneixen a la seqüència, restringint primerament la seva cerca amb el % de dissimilaritat. Vam considerar que un 10% de dissimilaritat podria ser favorable alhora de buscar els FT òptims pel nostre promotor.
Al obtenir els resultats vam observar que hi havia un gran nombre de possibles factors de transcripció que s'unirien en regions específiques de la seqüència promotora, per tant, vam decidir realitzar un nou filtratge de la informació per tal de disminuir els possibles falsos positius; vàrem agafar únicament els factors de transcripció amb un RE equally igual o inferior a 0,08, obtenint doncs la taula respresentada anteriorment (Figura 4).
Un segon estudi per la caracterització de la regió promotora va ser la realització d'un programa perl en el qual es testaven 13 matrius de factors de transcripció establerts pel professorat de l'assigantura. Després de molts esforços vam aconseguir tots els resultats necessaris per tal de poder realitzar l'estudi, obtenint doncs la taula representada (Taula 5).
Finalment vam comparar els resultats obtinguts en els dos estudis paral·lels
• Estudi de la funció del gen
Finalment per tal d'estudiar la funció del nostre gen vam utilitzar com a font per obtenir les dades el NCBI, mitjançant el qual hem pogut saber els GO associats a cada proteïna de fusió per separat. Posteriorment vam entrar dins del Gene Ontology per obtenir la informació específica de totes les possibles funcions que podien tenir. Paral·lelament a través del NCBI vam accedir a la informació de les nostres proteïnes present en l'OMIM (Online Mendelian Inheritance in Man). A més a més, vam buscar pel PubMed articles relacionats amb les dues proteïnes per tal d'entendre millor el seu funcionament específic. Cal dir que no hi ha cap article publicat de la nostra proteïna de fusió, ja que actualment no es coneix la seva funció específica, per tant, les nostres hipòtesis les hem basat en els articles trobats de cada proteïna per separat.
A partir de les dades recollides en els resultats i la informació extreta d'articles referents a les proteïnes que formen la proteïna de fusió, PRR5 i ARHGAP8, hem realitzat una sèrie de conclusions i hipòtesis.
La nostra proteïna de fusió està formada per dos proteïnes consecutives situades en el braç llarg del cromosoma 22, concretament a la posició 13.3.
La primera proteïna que la forma és el PRR5, una proteïna que actualment no té funció coneguda ni dominis associats. Presenta tres isoformes diferents, les quals es diferencien en l'inici de la traducció. En la isoforma 1 hi podem trobar els tres inicis de traducció, cosa que ens indica que les altres dues isoformes es poden produir per splicing alternatiu d'aquesta, sense que es produeixi una alteració de la pauta de lectura.
Cal dir, que les tres isoformes presenten una seqüència específica que es troba a l'inici de la proteïna de fusió estudiada (12 aminoàcids), per tant podem dir que totes les isoformes del PRR5 podrien ser vàlides per formar part d'ella.
La segona proteïna que la forma és el ARHGAP8. Aquesta proteïna presenta 2 isoformes característiques, les quals només es diferencien en que l'exó 4 de la isoforma 1 és absent en la isoforma 2. Per tant, la formació de la isoforma 2 també es podria atribuir a un splicing alternatiu del mRNA de la isoforma 1, sense alteració de la pauta de lectura. Aquesta proteïna és la que representa la major part de la nostra proteïna de fusió, en concret hi és representada tota la isoforma 2.
Per altra banda, si ens fixem en la proteïna fusionada , observem que entre les dues seqüències provinents del PRR5 i el ARHGAP8 s'hi troba una seqüència de 24 aminoàcids que no s'observa en les 5 isoformes esmentades. Mitjançant els transcrits vam observar que n'hi havia 3 que contenien aquesta seqüència seguida de la isoforma 2 de la proteïna ARHGAP8, cosa que ens va fer pensar que podria tenir-hi una relació molt estreta. Finalment, vam esbrinar que aquesta seqüència específica formava part de la regió upstream de la proteïna ARHGAP8, que inicialment semblava no codificant perquè no es traduïa quan s'expressava una isoforma sola.
Amb tots aquests resultats podem hipotetitzar dos possibles explicacions sobre la formació de la proteïna de fusió:
Pel que fa a l'expressió en teixits de la proteïna de fusió és força difícil determinar-la.
Teòricament, la seqüència promotora de la proteïna de fusió ha de correspondre a la regió promotora del PRR5, per tant, és lògic pensar que s'expressarà en aquells teixits on s'expressi el PRR5. Per alta banda, al estar formada per la totalitat de la proteïna ARHGAP8 i a més a més al ser factible pensar que faci la mateixa funció, podríem esperar trobar expressió de la proteïna de fusió en els teixits on s'expressa ARHGAP8.
Tot i que PRR5 i ARHGAP8 no tenen el mateix patró d'expressió, coincideixen en alguns teixits.
Nosaltres creiem que la nostra proteïna de fusió s'expressarà, com a mínim, en aquests teixits. Dins d'aquests volem destacar la presència de les dues proteïnes en el limfoma de Burkittis. Val a dir, que no descartaríem trobar la proteïna de fusió expressada en altres teixits tumorals, sobretot aquells on es sobreexpressa el PRR5 o el ARHGAP8 (càncer de colon i de pit).
Mitjançant l'estudi de la seqüència promotora de la proteïna de fusió , ens podem fixar que s'hi poden unir diferents factors de transcripció, on hem pogut observar que alguns d'ells estan involucrats en la regulació del cicle cel·lular, la generació d'apoptosis i la resposta al estrès cel·lular, com per exemple FOXO4, elk-1, c-Jun i p53, cosa que ens remet a que la nostra proteïna de fusió estigui present en teixits cancerigens.
Actualment no es coneix la funció específica de la nostra proteïna, ja que aquesta no ha estat descrita en cap article. Tot i així, a partir de la informació extreta de la funció de cada proteïna que la forma, es pot hipotetitzar la seva possible funció. Tenint en compte que la majoria de la seqüència de la proteïna correspon a la proteïna ARHGAP8 ,podríem atribuir que la funció predominant de la proteïna fusionada sigui la mateixa, és a dir, que formi part de la mobilitat i proliferació cel·lular, gracies a l'acció del domini RhoGap que estimula l'activitat GTPasa de les proteïnes GTPases, hidrolitzant doncs el seu GTP i fent que s'inactivi.
Per concloure, ens agradaria destacar que tant la proteïna de fusió, com les dues proteïnes que la formen, actualment estan sent estudiades per diferents grups d'investigació, per tal de poder aclarir quina és la seva funció específica tant en teixits sans com en processos tumorals.
&bull Articles
&bull Bases de dades
Març 2007