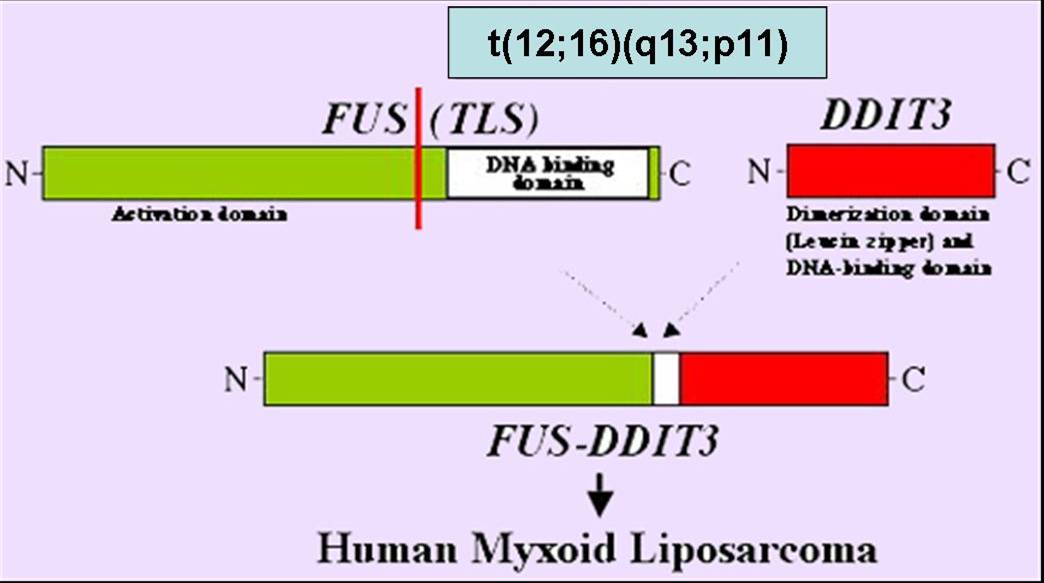
Figura 1: Fusió FUS-CHOP.
Estudi genòmic computacional del gen FUS-CHOP
Patricia Carulla & Alba Macip
patricia.carulla01@campus.upf.es & alba.macip01@campus.upf.es
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
Resum
Aquest treball es basa en l'estudi genòmic computacional de les proteïnes FUS (TLS) i CHOP (DDIT3) que, per translocació cromosòmica t(12;16)(q13;p11), donen lloc a una proteïna de fusió característica del liposarcoma mixoide maligne. Fus és una proteïna d'unió al RNA amb un domini Zinc-finger i Chop actua com a factor de transcripció en resposta a estímuls de dany cel.lular.
Partint de la seqüència aminoacídica de la proteïna quimèrica, analitzarem l'estructura genòmica, funció, expressió i homologia de les dues proteïnes individualment. Aquesta informació ens ajudarà a comprendre la fusió FUS-CHOP.
Per altra banda, l'estudi de la combinació de dominis resultant de la fusió així com la caracterització de la regió promotora ens permetran entendre el paper que juga aquesta translocació en el desenvolupament de la malaltia.

Figura 1: Fusió FUS-CHOP.
Resultats
La identificació de la seqüència proteica ens ha permès saber que aquesta és una proteïna fusionada d'humà.
L'estudi de FUS i CHOP per separat ens serveix per determinar les seves característiques. Els resultats de l'estudi de cadascun dels gens i de les proteïnes per les quals codifiquen els trobem resumits a continuació. Hi ha una informació més detallada seguint els enllaços des de Fus i Chop o des de cadascun dels apartats:
| FUS | CHOP | |
|---|---|---|
| DESCRIPCIÓ | veure descripció proteïna | veure descripció proteïna |
| ESTRUCTURA GENÒMICA |
- cromosoma: 16 - n° transcrits: 3 - n° exons:
|
- cromosoma: 12 - n° transcrits: 1 - n° exons: 4 - n° isoformes: 1 |
| ESTRUCTURA PROTEICA |
- domini d'unió al RNA - zinc-finger [+informació] |
- domini d'unió al DNA - leucine-zipper [+informació] |
| HOMOLOGIA | veure taula | veure taula |
| EXPRESSIÓ | veure taula | veure taula |
| FUNCIÓ | Unió a RNA... [+ informació] |
Factor de transcripció... [+ informació] |
| REGIÓ PROMOTORA | Veure factors de transcripció | Veure factors de transcripció |
Un cop analitzades les proteïnes per separat, passem a estudiar les característiques de la proteïna resultant de la fusió. Aquestes característiques es resumeixen en el quadre següent, on també trobem enllaços cap a informació més detallada.
| FUS-CHOP | |
|---|---|
| DESCRIPCIÓ | Descripció de la fusió |
| FUSIÓ |
-Quins exons de cada proteïna participen en la fusió? -Com es produeix la fusió de les proteïnes? [+ informació] |
| PROTEÏNA RESULTANT |
-Com s'altera l'estructura de la proteïna? -Com s'altera la funció de la proteïna? [+ informació] |
| REGIÓ PROMOTORA | Veure factors de transcripció |
A més hem considerat oportú complementar aquest estudi amb els següents punts:
- Conservació dels dominis de les proteïnes
- Predicció del tipus i punt de fusió entre les proteïnes Fus-Chop
Materials i mètodes
En aquest apartat trobem els enllaços on s'especifiquen ls passos realitzats per obtenir tots els resultats comentats anteriorment.
Discussió
Aquest estudi computacional s'ha centrat en el gen FUS-CHOP humà, un gen fusionat codificant per una proteïna quimèrica causant del liposarcoma mixoide, un dels subtipus més comuns de tumors en teixit tous.
Degut a que aquest és un gen fusionat i no es troba com a tal en la majoria de bases de dades, s'han hagut d'estudiar els dos gens implicats per separat.
Aquest gen quimèric és una fusió de tipus I entre el gen Fus (transcrit 1, exons 1-7) i el gen Chop (exons 2-4). Per una banda, el gen Fus, bastant conservat al llarg de l'evolució, codifica per una proteïna d'unió al DNA i RNA. Per altra banda Chop, també conservat, és un factor de transcripció amb domini leucine zipper que s'indueix en resposta d'estrès cel.lular. Aquestes funcions venen determinades pels dominis de la proteïna. Com que només una porció de cada proteïna participa en la fusió, no tots els dominis es mantenen en la proteïna quimèrica. En conseqüència, la Fus-Chop actua com a un factor de transcripció anormal que altera el control transcripcional de molts gens diana.
Els gens Fus i Chop estan regulats per diversos factors de transcripció amb unió a llocs específics de la seqüència promotora que controlen la seva expressió. S'ha demostrat que aquests gens s'expressen de forma ubiqua, degut a la importància de la seva funció, i que la proteïna fusionada s'expressa sobretot a nivell de teixits tous, on es desenvolupa el liposarcoma.
Referències
Bibliografia:
Panagopoulos I, Mandahl N, Ron D, Hoglund M, Nilbert M, Mertens F, Mitelman F, Aman P. ,Characterization of the CHOP breakpoints and fusion transcripts in myxoid liposarcomas with the 12;16 translocation. .. Cancer Res 15(24):6500-03, 1994
J Peérez-Losada, M Sánchez-Martín, MA Rodríguez-García, PA Pérez-Mancera, B Pintado, T Flores, E Battaner and I Sánchez-García*,Liposarcoma initiated by FUS/TLS-CHOP: the FUS/TLS domain plays a critical role in the pathogenesis of liposarcoma.Oncogene 19:6015±602, 2000
Cristina R. Antonescu,* Abul Elahi,† Marcia Humphrey,* Man Yee Lui,* John H. Healey,‡ Murray F. Brennan,‡ James M. Woodruff,* Suresh C. Jhanwar,† and Marc Ladanyi*†,Specificity of TLS-CHOP Rearrangement for Classic Myxoid/Round Cell Liposarcoma.Journal of Molecular Diagnostics, 2(4), 2000
Cristina R. Antonescu,* Abul Elahi,† Frank Willeke,2 Ruediger Ridder, Gunhild Mechtersheimer, Mathias Schwarzbach, Alexander Duwe, Juergen Weitz, Thomas Lehnert, Christian Herfarth, and Magnus von Knebel Doeberitz,Prognostic Impact of P53 Status, TLS-CHOP Fusion Transcript Structure, and Histological Grade in Myxoid Liposarcoma: A Molecular and Clinicopathologic Study of 82 Cases1.Clinical Cancer Research, 7:3977–3987, 2001
Cristina R. Antonescu,* Abul Elahi,† Martine Pomerance, Daniel Carapau, Françoise Chantoux, Michaël Mockey, Claude Correze, Jacques Francon, and Jean-Paul Blondeau ,C/EBP-homologous protein (CHOP) expression and transcriptional activity are regulated by cyclic AMP in thyroid cells .Mol. Endocrinol, 7 2003
Cristina R. Antonescu,* Abul Elahi,† Fujii R, Takumi T,TLS facilitates transport of mRNA encoding an actin-stabilizing protein to dendritic spines.Cell Sci, 15:5755–65, 2005
Endo M, Mori M, Akira S, Gotoh T. ,C/EBP homologous protein (CHOP) is crucial for the induction of caspase-11 and the pathogenesis of lipopolysaccharide-induced inflammation.J Immunol 15:6245–53, 2006
Shirakawa K, Maeda S, Gotoh T, Hayashi M, Shinomiya K, Ehata S, Nishimura R, Mori M, Onozaki K, Hayashi H, Uematsu S, Akira S, Ogata E, Miyazono K, Imamura T,CCAAT/enhancer-binding protein homologous protein (CHOP) regulates osteoblast differentiation.Mol Cell Biol 26:6105–16, 2006
Bases de dades i software:
Clustalw: http://www.ebi.ac.uk/clustalw/
NCBI: http://www.ncbi.nih.gov/
Ensembl: http://www.ensembl.org/
Biomart: http://www.ensembl.org/biomart/martview/be6035946b855fcf976e76171b44e275/
UCSC: http://genome.ucsc.edu/
Uniprot: http://www.ebi.uniprot.org/index.shtml/
Pfam: http://www.sanger.ac.uk/Software/Pfam/search.shtml/
Atlas of Genetics and Cytogenetics in Oncology and Haematology: http://atlasgeneticsoncology.org/