Un gen està format per: un promotor, el codó ATG d'inici, exons (un o més), introns (cap, algun/s) i un codó stop (TAA, TAG, TGA).
Un exó és una seqüència de DNA que codifica per proteïna; per altra banda un intró és una seqüència de DNA no codificant. Tota la informació necessària per a la síntesi d'una proteïna rep el nom de cistró (conjunt d'exons i introns).
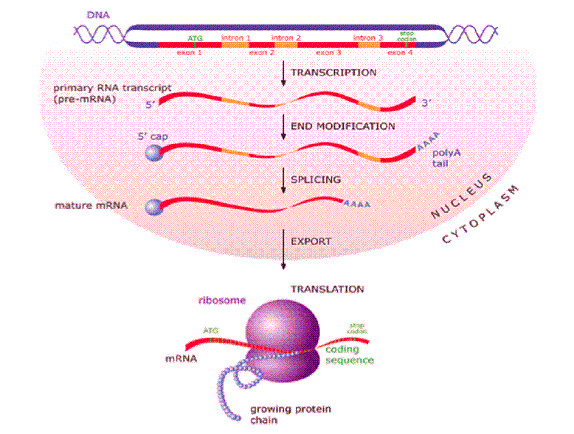
Per tal d'eliminar els introns del mRNA cal que tingui lloc un procés anomenat splicing. Aquest procés requereix unes molècules anomenades RNPnp (RNA+proteïnes) que s'encarreguen d'eliminar els introns del mRNA primari obtenint així un mRNA (madur) format únicament per nucleòtids codificants (exons). Els RNPnp identifiquen seqüències concretes que determinen el processament del mRNA primari. La majoria d'introns comencen amb un seqüència que conté un GT en una posició concreta (donor site) i acaben amb una seqüència que conté un AG en una posició determinada (acceptor site).
Aquestes seqüències senyals, no sempre són reconegudes per els RNPnp de manera que el que obtenim és un splicing alternatiu resultant en una proteïna lleugerament diferent. En el següent esquema es veu en què consisiteix el procés de splicing.

Com que la resta de nucleòtids al voltant del GT i de l'AG no són sempre els mateixos, a partir d'un conjunt de seqüències alineades, es construeix una matriu de pesos, en la que cada nucleòtid en cada posició rep una puntuació (score) concreta. La puntuació global d'un potencial lloc de splicing és la suma de les puntuacions de cada nucleòtid que en forma part.
Els exons que es tradueixen a proteïna mostren un biaix en la utilització de certs codons, és a dir, hi ha una certa exonicitat, en la que determinats codons són utilitzats amb una freqüència superior. A més, els codons sinònims també són utilitzats en freqüències diferents. Aquesta preferència per determinats codons és especïfica de cada grup taxonòmic, és a dir, és espècie-depenent, i existeix una correlació entre la divergència taxonòmica i la similaritat en l'ús de codons. En canvi, aquest biaix és absent en els les seqüències intròniques, on la distribució dels codons és uniforme.
El metòde que nosaltres proposem per predir exons es basa en utilitzar matrius de pesos per localitzar potencials llocs de splicing donadors i acceptors, i en la utilització d'una taula d'ús de codons per determinar el biaix codificant dels exons predits. Un cop predits els dos senyals de splicing, analitzem els codons de la seqüència. Com més elevat sigui el nombre de codons que observem que d'acord amb la taula, apareixen freqüentment en regions codificants, conclourem que més alt és el seu potencial codificant, i en conseqüència, més probable serà que el fragment sigui un exó real.