
| INICI ~ OBJECTIU ~ INTRODUCCIÓ ~ MATERIALS ~ MÈTODES ~ EXPLICACIÓ DETALLADA DEL SCRIPT ~ RESULTATS ~ CONCLUSIÓ ~ REFERÈNCIES ~ AUTORES ~ |
Després de realitzar el pas 1 i 2, utilitzant les comandes sort, uniq...volíem mirar aquelles anotacions que se solapaven. En aquest pas vam tenir un problema, fins ara per mirar si eliminavem o no les anotacions repetides havien de coincidir les posicions cromosòmiques del començament i final dels exons; però fent-ho de la mateixa manera que al pas 1 i 2 ens van donar moltes anotacions:
Vam passar de les 1.670 inicials a 1.646 realitzant el sort i uniq igual que en el pas 1 i 2. Aquest fitxer el vam anomenar juntuniq.txt.
El juntuniq.txt el vam dividir en dos fitxers:
el solapades.txt que contenia 24 anotacions (23 de Ensembl + 1 de RefSeq)
A partir del fitxer nosolapades.txt mitjançant la comanda:el nosolapades.txt que contenia 1622 anotacions (528 de Ensembl + 372 de RefSeq + 561 de genscan + 161 de geneid).
bash$ egrep "NT_" nosolapades.txt per genscan
vam fer un fitxer que només contenia, doncs, les anotacions de genscan i de geneid, anomenat gegenosolapades.txt que contenia 722 anotacions (561 de genscan + 161 de geneid).bash$ egrep "chr[0-9]_" nosolapes.txt per geneid
Com és obvi de pensar era impossible continuar el projecte amb 722 anotacions, per tant, el que vam decidir va ser restringir més. Enlloc de fixar-nos amb les posicions cromosòmiques del començament i final dels exons, el que vam fer va ser veure si les anotacions coincidien o bé estaven entre l'inici i el final de la transcripció. D'aquesta manera vam poder passar de 722 a 279 anotacions, de les quals vam decidir de fer només les de geneid (32 anotacions), ja que com ja està explicat a l'apartat de mètodes és un programa més fiable per predir l'existència de possibles gens novells que no pas genscan.

En aquest apartat explicarem el perquè vam haver de fer servir el UCSC Genome Browser on Human July 2003 Freeze per tal de verificar que les anotacions trobades eren realment resultat de l'eliminació de totes les anotacions que no pertanyien a RefSeq i Ensembl.
El problema, va ser que no vam tenir en compte que alhora d'eliminar les anotacions repetides, no podíem eliminar les de RefSeq i Ensembl, ja que aquestes corresponien a anotacions de gens reals. Per tant, les que hauríem d'haver eliminat eren les anotacions de prediccions, és a dir, les de geneid i genscan. Per mostrar aquesta problemàtica, es mostren els següents equemes:
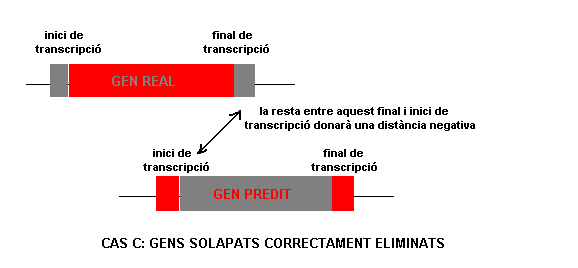
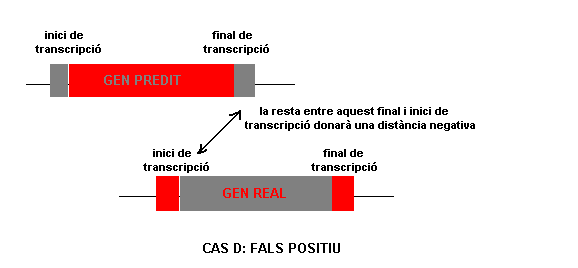
Per tant, el que succeïa era que en els casos on l'anotació de gens reals (la corresponent a RefSeq o Ensembl) precedia a la de gens predits (geneid o genscan) i la diferència donava un número negatiu per als gens predits, elimināvem aquests gens correctament, tal i com es mostra en el dibuix del cas c; ara bé, el problema venia quan teníem el cas contrari, és a dir, que els gens predits precedien els gens reals: en aquest cas, tal i com mostra el bibuix del cas D, si els gens se solapaven, la que adquiria el signe negatiu era l'anotació del gen real, el qual ignoràvem; així doncs, en aquests casos ens quedàvem amb l'anotació de la predicció obtenint un fals positiu.
Com això no ho vam tenir en compte, (no va ser culpa nostra, era un problema de mal plantejament inicial del projecte) vam haver d'anar a la pàgina d'UCSC Genome Browser on Human July 2003 Freeze per comprovar que les 23 anotacions de gens, no coincidien amb cap d'Ensembl i RefSeq. Si aquestes, realment no coincidien, volia dir que probablement ens trobaven davant de possibles canditats a gens novells.
Aquesta imatge,que ens mostra el chr2_2056.1, ens dona un resultat incorrecte ja que observem que aquesta anotació està inclosa a Ensembl, Geneid i Genscan.
Aquesta imatge ,que ens mostra el chr5_1143.1, ens dona un resultat correcte ja que observem que aquesta anotació està inclosa només a Geneid i a Genscan.
