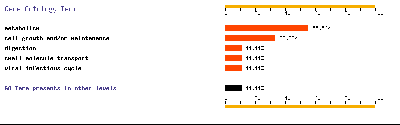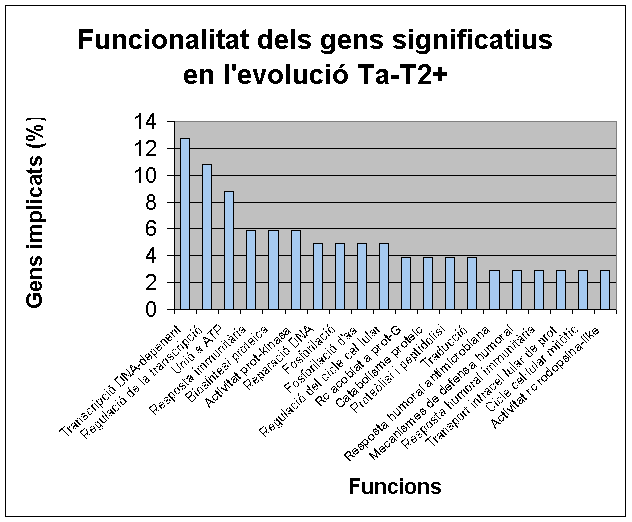
Cluster progressió Ta-T1 |

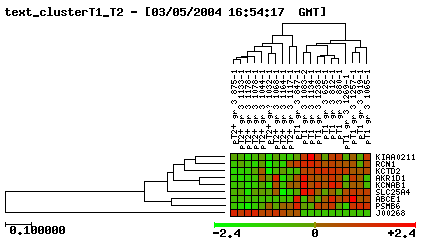
Cluster progressió T1-T2 |
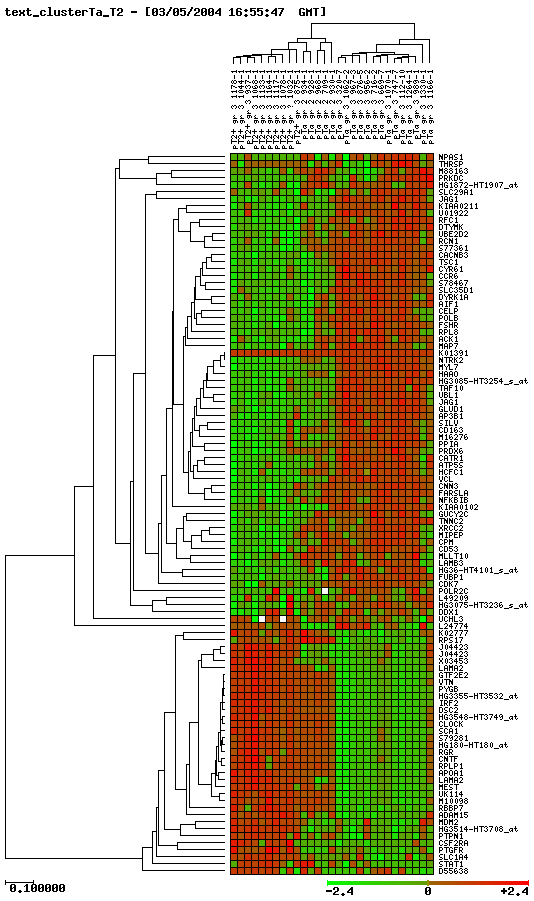
Cluster progressió Ta-T2 |
Tal com hem comentat anteriorment a Materials i Mètodes, mitjançant comandes en Shell vam intentar trobar gens presents tant en l'estudi de Sanchez-Carbayo com en els resultats obtinguts per nosaltres a partir del set de dades de l'altre article comentat.
Malauradament, no hem trobat gens comuns. De totes maneres, estudiarem els més significatius segons els clusters. A continuació indiquem aquests gens, la seva localització cromosòmica i la seva funció.
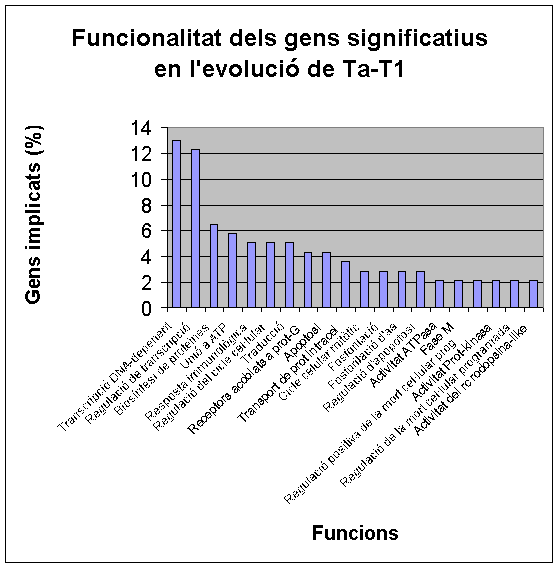
Gens diferencialment expressats en la progressió Ta-T1:
| Sobreexpressats | Gene Symbol | Cromosoma | Funció |
|---|---|---|---|
| ETV6 | 12 | Factor de transcripció de la família ETS. | |
| NPM1 | 5 | Fosfoproteïna nucleolar d'unió a RNA. | |
| DDX3X | X | De la família "DEAD box proteins". Involucrada en: inici de traducció, splicing mitocondrial i nuclear, ensamblatge dels ribosomes. | |
| RORC | 1 | Sistema immune: embriogènesi limfoide i regulació de la timopoiesis. | |
| TNR | 1 | Desenvolupament embriològic. |
| Reprimits | Gene Symbol | Cromosoma | Funció |
|---|---|---|---|
| DSC2 | 18 | Membre de la família de les cadherines. Important en les unions cèl.lula-cèl-lula. | |
| VTN | 17 | Implicada en l'adhesió | |
| NR4A3 | 9 | Activador de la transcripció | |
| MEST | 7 | Juga un paper clau en el desenvolupament |
.gif)
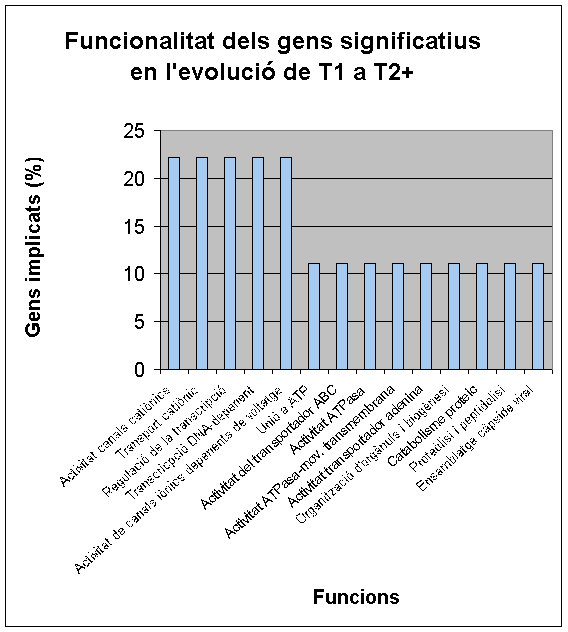
Gens diferencialment expressats en la progressió Ta-T2:
| Sobreexpressats | Gene Symbol | Cromosoma | Funció |
|---|---|---|---|
| LAMA2 | 6 | Proteïna extracel.lular que regula adhesió, migració i organització cel.lular durant el desenvolupament embrionari. | |
| VTN | 17 | Implicada en l'adhesió | |
| MEST | 7 | Juga un paper clau en el desenvolupament | |
| APOA1 | 11 | Metabolisme lipídic. Promou la sortida del colesterol des de diversos teixits cap al fetge. |
| Reprimits | Gene Symbol | Cromosoma | Funció |
|---|---|---|---|
| CCR6 | 6 | Sistema Immune. Receptor de quimiocines de les cèl.lules T i dendrítiques. | |
| AIF1 | 6 | Sistema Immune. Gens induït per citoquines i interferó. Contribueix a la resposta anti-imflamatòria. | |
| TAF10 | 11 | Iniciació de la transcripció | |
| JAG1 | 20 | Desenvolupament. Lligand del receptor notch 1. |
.gif)
Com podem comprovar en les taules anteriors, els gens que nosaltres hem seleccionat com a més significatius tenen una funcionalitat similar als dels dos articles comentats. Així doncs, podem veure gens típicament relacionats amb els processos tumorals com la pèrdua d'adhesió, que estaria involucrada en processos de metàstasi; gens, la repressió dels quals, suposaria una pèrdua en l'eficàcia del sistema immune. També es veu alterat el metabolisme lipídic i, com era d'esperar, observem desregulació en el procés de la transcripció amb la conseqüent afectació del cicle cel.lular.
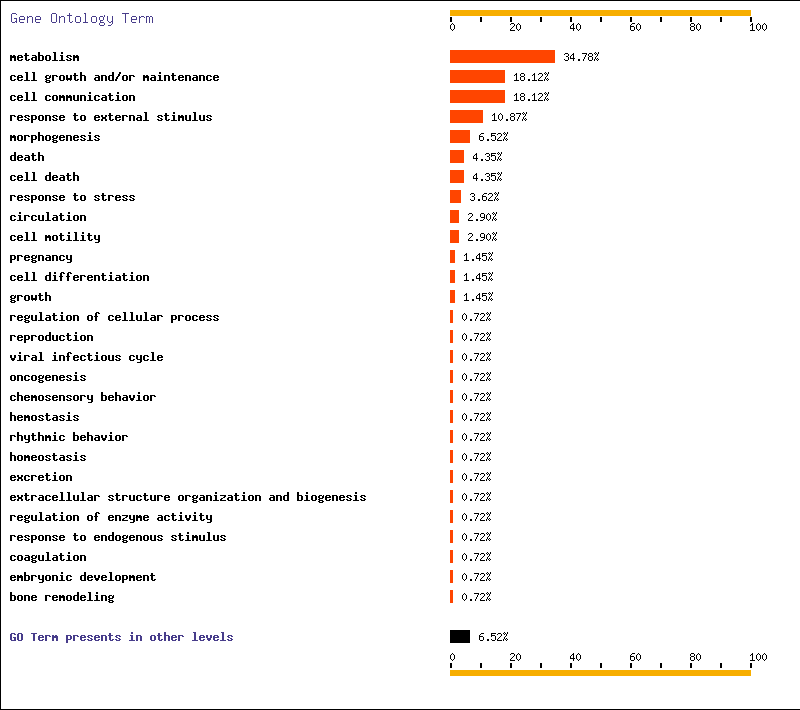
A continuació, mostrem les gràfiques obtingudes amb el FatyGO i GOCharts. A més de les funcions ja comentades anteriorment, cal destacar la importància del procés de mort cel.lular programada i l'oncogènesi. Després de la comparació amb les publicacions en les quals ens hem basat, treiem les mateixes conclusions que observant els clusters i així ratifiquem la funcionalitat dels gens diferencialment expressats en la progressió del càncer de bufeta.