INTRODUCCIÓN
Frecuentemente en las proteínas de eucariotas encontramos repeticiones de aminoácidos en tándem, son las zonas llamadas homopoliméricas. Uno de los mecanismos que puede explicar la abundancia de estas regiones repetitivas es el llamado slippage (1) , sin embargo, también se podrían haber producido por acumulación de mutaciones puntuales (2) .
El proyecto consiste en hacer una compilación de todas las regiones de aminoácidos de longitud 5 o más, en varios genomas, y estudiar varias características. Los objetivos serán determinar qué aminoácidos tienen más tendencia a encontrarse repetidos, analizar qué longitudes tienen los homopolímeros de diferentes aminoácidos y ver si hay preferencias en su localización relativa dentro de las secuencias de proteínas. (1)
Los resultados del programa aparecen en un fichero de salida ( outfile) llamado resultados.txt.
Ejemplo:
En este fichero sólo aparecen las secuencias que tienen repetición (el mismo aminoácido repetido 5 o más veces). En el caso de que una proteína tenga dos repeticiones, saldrá dos veces junto con su identificador (mirar ejemplo casos 2 y 3). Cada secuencia aparece en una línea, y cada columna está separada por un espacio.
1ª columna: identificador de la proteína en ensembl.
2ª columna: identificador del gen que codifica para la proteína en ensembl.
5ª columna: cromosoma en el que se encuentra el gen.
8ª columna: aminoácido que se repite.
9ª columna: longitud de la repetición (número de veces que se repite el aminoácido).
10ª columna: inicio de la repetición.
11ª columna: longitud de la secuencia.
12ª columna: cuartil en el que se encuentra la repetición.
Nosotras analizamos concretamente los proteomas de Homo sapiens, Mus musculus (ratón), Fugu rubripes (un pez) y Drosophila melanogaster .
A partir de los resultados obtenidos, y usando las órdenes de egrep (principalmente cut, paste, uniq, sort y redireccionamiento), obtuvimos datos útiles para hacer luego gráficos en Excel.
Clica aquí para volver al índice
Obtención de los datos para realizar la gráfica (egrep):
$ wc resultados.txt ===> nos da el número de líneas que contiene el fichero resultados.txt, es decir, el número de repeticiones que tiene el proteoma analizado.
El número de proteínas analizadas lo obtenemos a partir de un contador del programa que realizamos.
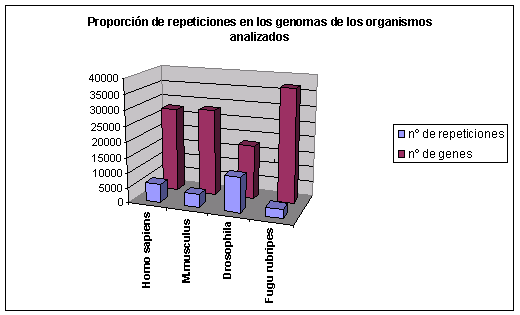
Fig.1
Observaciones:
Como vemos en la gráfica no existe relación entre el número de proteínas de una especie y el número de repeticiones que hay en el proteoma. Así, por ejemplo, tenemos como casos extremos y opuestos el de Drosophila melanogaster y Fugu rubripes: Drosophila melanogaster es la especie con el menor nº de proteínas de las especies analizadas y el mayor nº de repeticiones (en valor absoluto), y Fugu rubripes es la especie con el mayor nº de genes y el menor nº de repeticiones. Entre Homo sapiens y Mus musculus, dos especies evolutivamente cercanas, observamos cierta diferencia. Resulta curioso ver que en Homo sapiens aún y teniendo un número de proteínas comparable al de Mus musculus (Mus musuclus tiene únicamente 369 proteínas más que el hombre) tiene un mayor número de repeticiones (1592 más).
El hecho de que las repeticiones aminoacídicas sean más abundantes en la mosca podría ser debido a diferencias innatas en: procesos de replicación del DNA, mecanismos de reparación, sistemas de modificación del DNA y sesgos mutacionales(4). Esto además, para que fuera sometido a una selección positiva tendría que tener alguna función biológica. De hecho, se sabe que en Drosophila melanogaster, más del 80% de las proteínas que contienen repeticiones son esenciales en su desarrollo, como reguladores de la transcripción.(4) Muchas de estas proteínas tienen homólogos en humano y en ratón.(3)
Clica aquí para volver al índice
Una vez obtenidos los resultados de la gráfica anterior, se nos planteó la duda de si el aumento de las repeticiones de un genoma respecto de los otros, se debía a que había más proteínas con repetición o más repeticiones por proteína. Por eso, calculamos el número de proteínas que tenían una o más repeticiones y lo comparamos con el número de repeticiones totales (respecto al número de proteínas del proteoma).Es decir, por un lado hicimos nº de proteínas con repetición entre nº de proteínas totales y, por otro lado, nº de repeticiones entre nº de proteínas totales.
Obtención de los datos para realizar la gráfica (egrep):
$ cut -d ' ' -f 1 resultados.txt > identificador.txt ===> obtenemos la columna que tiene el identificador de la proteína.
$ uniq -c identificador.txt > identificadoruniq.txt ===> juntamos las líneas iguales (proteínas que tienen más de una repetición). En este caso no hacía falta hacer un sort antes de hacer el uniq, dado que las proteínas que tenían más de una repetición (con el mismo identificador), ya saldrían juntas.
$ wc identificadoruniq.txt ===> contamos el número de líneas que tiene el nuevo archivo (será el número de proteínas que tienen al menos una repetición).
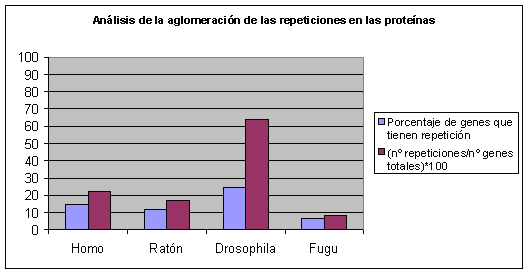
Fig.2
Observaciones:
Cuanta más diferencia exista entre las dos barras, más proteínas habrá con más de una repetición. En Drosophila melanogaster, por ejemplo, vemos una gran diferencia, por tanto podemos afirmar que en su proteoma, las repeticiones tienden a aglomerarse, mientras que en Fugu rubripes la mayoría de repeticiones son únicas en la proteína. La diferencia entre humano y ratón no es significativa, sólo podemos volver a decir que en humano hay más repeticiones que en ratón.
Clica aquí para volver al índice
Aquí analizamos cuáles eran los aminoácidos que más se repetían por especie.
Obtención de los datos para realizar la gráfica (egrep):
$ cut -d ' ' -f 8 resultados.txt > aa.txt ===> tendremos un archivo con únicamente la columna con el aminoácido que se repite.
$ sort aa.txt | uniq -c > aauniq.txt ===> ordenamos alfabéticamente los aminoácidos y juntamos los que son iguales, así nos da el número de veces que aparece cada aminoácido.
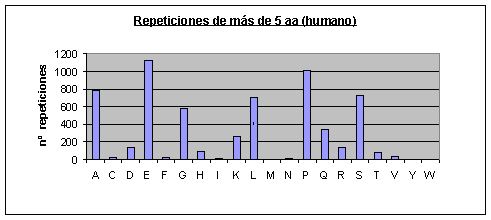
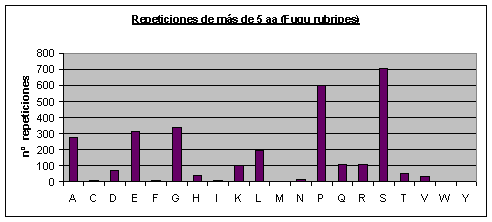
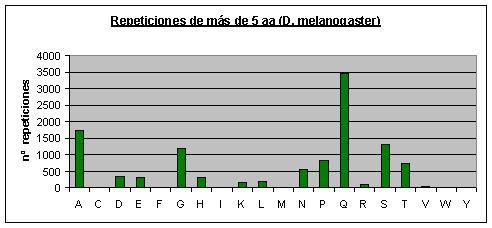
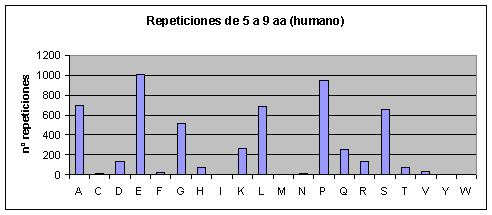
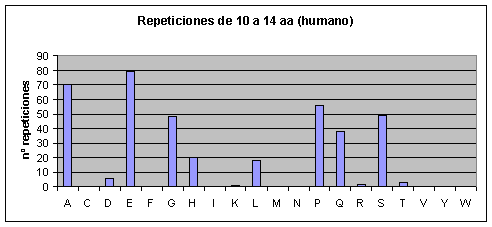
Fig.3
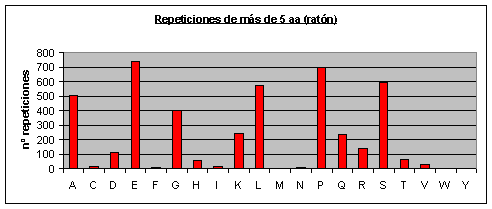
Fig.4
Fig.5
Fig.6
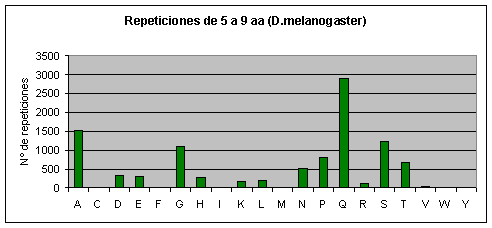
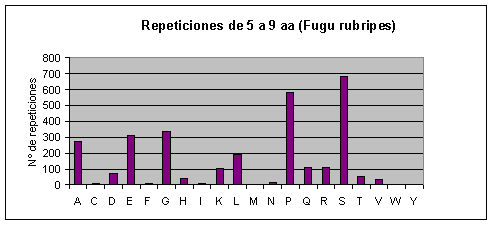
Fugu rubripes: Los aminoácidos que más se repiten son serina, prolina y glicina. Casi no se repiten o no se repiten la cisteína, la fenilalanina, la isoleucina, la metionina, la asparagina, el triptófano y la tirosina. Fig.5
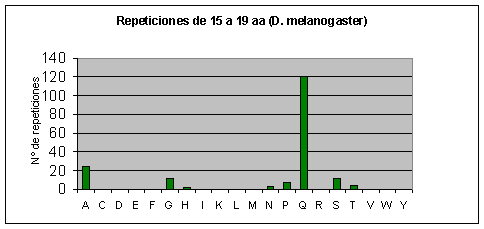
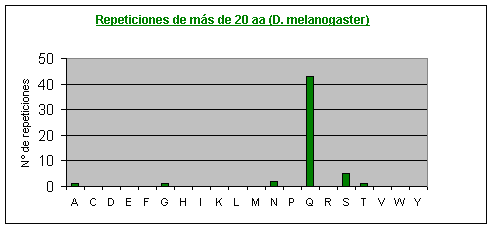
Drosophila melanogaster: El aminoácido que más se repite con diferencia es la glutamina (el 30% de la repeticiones son de poliglutaminas). También se repiten mucho la alanina y la serina. Casi no se repiten o no se repiten la cisteína, la fenilalanina, la isoleucina, la metionina, el triptófano y la tirosina.Fig.6
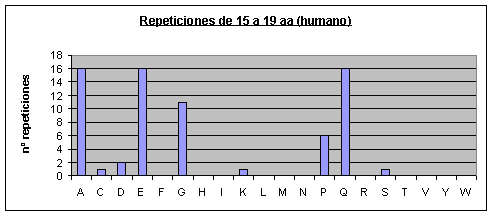
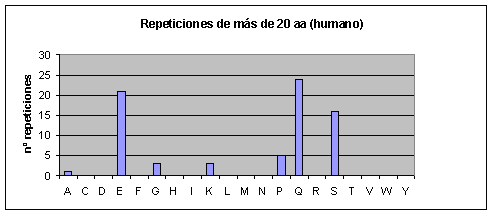
Humano: Los que más se repiten son ácido glutámico, prolina, alanina, serina, leucina y glicina. Los que menos se repiten son triptófano, tirosina, asparagina, metionina, isoleucina, fenilalanina y cisteína.Fig.3
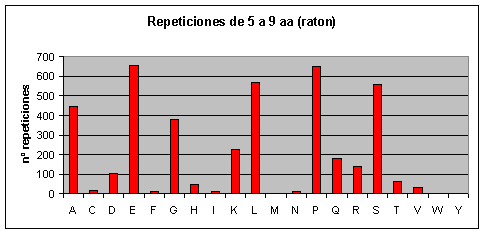
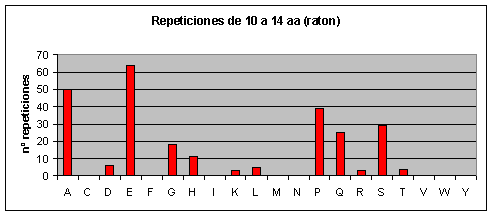
Ratón: Los que más se repiten son ácido glutámico, prolina, alanina, serina, leucina y glicina. Los que menos se repiten son triptófano, tirosina, asparagina, metionina, isoleucina, fenilalanina y cisteína.Fig.4
Observaciones:
Los aminoácidos que menos se repiten coinciden en las cuatro especies, excepto la asparagina que aparece bastantes veces repetida en Drosophila melanogasterFig.6. Estos resultados contradicen en algunos aspectos un artículo escrito en 1994 (3) en el cual se dice que no aparecen repeticiones de Lisina, y además no incluye como repeticiones raras las de fenilalanina y asparagina, pero debemos considerar que en 1994 aún no se habían secuenciado muchas proteínas y los autores realizaron el estudio a partir de proteínas de diferentes especies.
Muchos artículos afirman que los aminoácidos más repetidos son los hidrofílicos(3). Sin embargo, vemos que hay aminoácidos hidrófobos, como la prolina, que también se repiten abundantemente. De todas formas, los resultados de los aminoácidos más repetidos concuerdan con los obtenidos en algunos artículos más recientes (5). También estamos de acuerdo con el mismo artículo (en el cual no se analizaron los mismos proteomas que los nuestros) en que las diferencias más significativas entre los proteomas están en las repeticiones de glutamina, asparagina y leucina, y en nuestro caso estas diferencias son sobretodo entre los proteomas de los mamíferos (humano y ratón) y el de Drosophila melanogaster.
Observando los gráficos de humano y ratón Fig.3 y Fig.4 resulta impactante ver que parece que se trate del mismo gráfico. Sin embargo, vemos que la escala del eje de las y es diferente (como ya hemos dicho el proteoma humano tiene más repeticiones). Por lo tanto, en el humano el aumento de repeticiones, con respecto al proteoma de ratón, parece haberse distribuido de forma homogénea por todos los aminoácidos.
Clica aquí para volver al índice
Si desglosamos los gráficos anteriores en diferentes longitudes de las repeticiones, obtenemos los gráficos siguientes.
Humano
Fig.7
Fig.8
Fig.9
Fig.10
Ratón
Fig.11
Fig.12
Fig.13
Fig.14
Drosophila melanogaster
Fig.15
Fig.16
Fig.17
Fig.18
Fugu rubripes
Fig.19
Fig.20
Fig.21
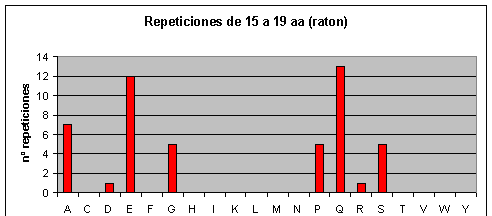
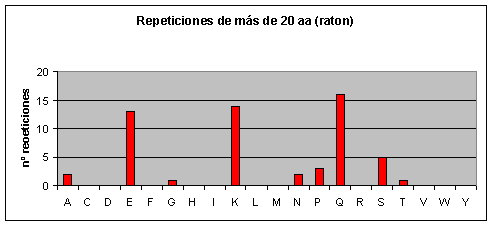
A medida que las repeticiones se hacen más largas también se hacen más escasas en todos los proteomas. Hay ciertas repeticiones, sin embargo, que tienden a ser más largas que otras. Así por ejemplo, en el caso de humano y de ratón, conforme vamos aumentando la longitud de la repetición también va aumentando la abundancia relativa (relativa a los otros aminoácidos) de las poliglutaminas. Las repeticiones largas en el caso de humano y ratón, pues, están formadas en su mayoría por glutamatos y glutaminas. Cabría destacar que en el ratón también aparecen muchas repeticiones largas de lisinas, mientras que en humano no hay ninguna.
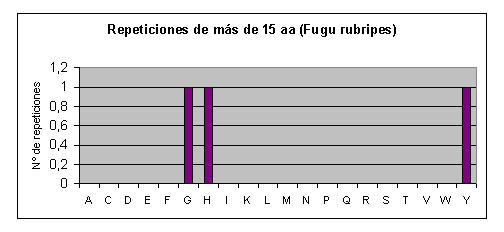
Por otra parte, en Fugu rubripes no existen repeticiones largas. En Drosophila melanogaster, también aumenta la abundancia relativa de las poliglutaminas a medida que aumenta la longitud de la repetición y además son prácticamente las únicas que aparecen.
Clica aquí para volver al índice
En este caso, se trataba de ver si las repeticiones se distribuían aleatoriamente en la secuencia o bien tendían a producirse en alguna región concreta de la proteína.
Obtención de los datos para realizar la gráfica (egrep):
$ cut -d ' ' -f 12 resultados.txt > cuartil.txt ===> tendremos un archivo con únicamente la columna en la que aparece el cuartil en el que se encuentra la repetición.
$ sort cuartil.txt | uniq -c > cuartiluniq.txt ===> ordenamos los cuartiles y juntamos los que son iguales, así nos da el número de repeticiones por cuartil.
Fig.22
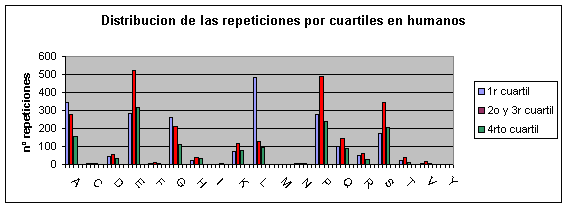
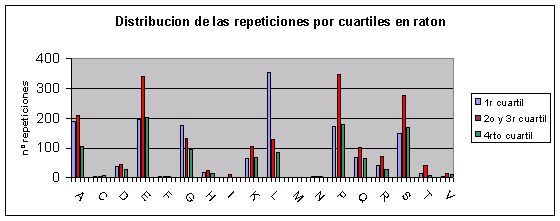
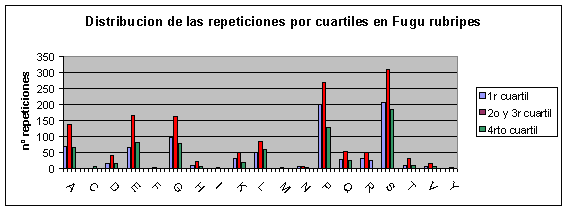
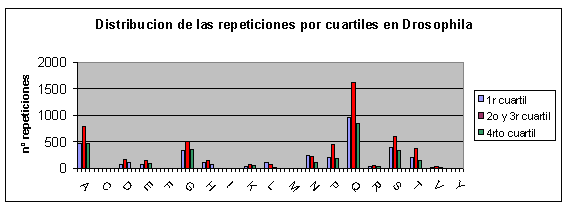
Observaciones:
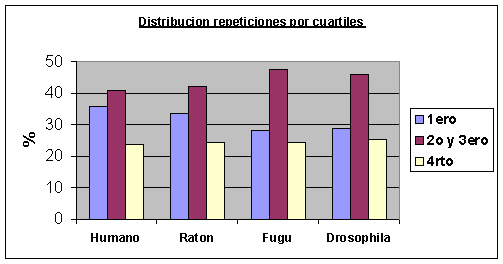
Para analizar estos gráficos hay que tener en cuenta que la barra del medio de cada especie corresponde a la cantidad de repeticiones en dos cuartiles (2º y 3º) mientras que las otras dos barras sólo indican el número de repeticiones de un cuartil. Por lo tanto, si la distribución fuera al azar aparecerían las barras del medio con el doble de altura que las otras 2, que deberían tener una altura semejante.
En Fugu rubripes y Drosophila melanogaster aunque aparecen más repeticiones en el primer cuartil la diferencia con los otros no parece significativa. Sin embargo, en humano y ratón sí que podríamos decir que no existe una distribución aleatoria de las repeticiones a lo largo de la proteína sino que se suelen concentrar en el primer cuartil. En la bibliografía leída no aparece ninguna alusión a este hecho, de modo que desconocemos el porqué del mismo.
Clica aquí para volver al índice
Si esta distribución por cuartiles la analizamos en cada aminoácido podremos saber si hay ciertos aminoácidos que se repiten preferentemente en alguna región.
Obtención de los datos para realizar la gráfica (egrep):
$ cut -d ' ' -f 12 resultados.txt > cuartil.txt ===> tendremos un archivo con únicamente la columna en la que se encuentra la posición relativa de la repetición.
$ paste aa.txt cuartil .txt | sort | uniq -c > aacuartiluniq.txt ===> obtendremos un archivo en el cual la primera columna corresponderá al aminoácido y la segunda al cuartil. Con el sort ordenamos alfabéticamente por aminoácidos de manera que aminoácidos iguales saldrán juntos. Con el uniq, por último, juntamos las líneas iguales consecutivas, es decir, los aminoácidos iguales que además aparecen en el mismo cuartil.
Fig.23
Fig.24
Fig.25
Fig.26
Observaciones:
Tanto en Fugu rubripes (Fig.25) como en Drosophila melanogaster(Fig.26) no destaca ningún aminoácido por tener una tendencia a acumularse en alguna región concreta de la proteína. Sólo existirían un par de casos en Drosophila melanogaster en el que el aminoácido aparece más veces en el primer cuartil pero no podemos decir que esto sea significativo ya que el número de repeticiones es pequeño, por lo tanto, lo más seguro es que sea efecto del azar.
En cambio, en humano (Fig.23) y ratón (Fig.24) existen algunos aminoácidos con tendencia a agrupar las repeticiones en el primer cuartil. Estos aminoácidos son: la alanina, la leucina y la glicina. Concretamente en la leucina, la diferencia parece muy significativa, así que es muy posible que las polileucinas en el extremo N-terminal tengan alguna función biológica importante.
Clica aquí para volver al índice
Con estos gráficos la intención era estudiar si había algún cromosoma que tuviera más repeticiones que el resto. Para poder comparar los diferentes cromosomas, ya que son de diferente tamaño, dividimos el número de proteínas que tenían repetición en cada cromosoma entre el número de proteínas totales de dicho cromosoma.
Sólo analizamos los cromosomas de ratón y de humano ya que son de los únicos que conocemos algo.
Obtención de los datos para realizar la gráfica (egrep):
$ cut -d ' ' -f 5 resultados.txt > chr.txt ===> tendremos un archivo con únicamente la columna en la que se encuentra el cromosoma
$ sort chr.txt | uniq -c > chruniq.txt> ===> obtendremos un archivo en el cual la columna con el número de cromosoma la ordenamos numéricamente y juntamos las líneas iguales, obteniendo así el número de proteínas con una o más repeticiones que aparecen en cada cromosoma.
Fig.27
Fig.28
Observaciones:
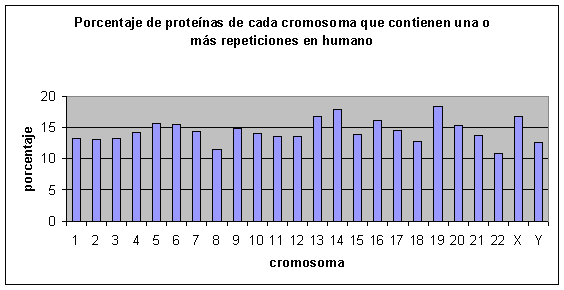
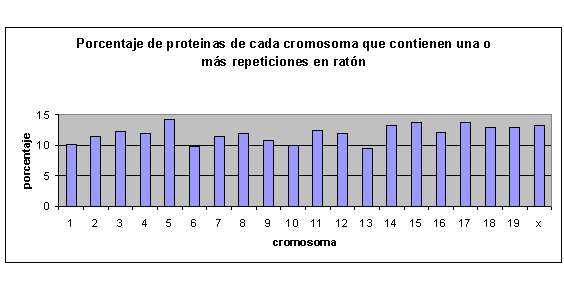
Tanto en humano como en ratón no aparece ningún cromosoma que destaque por tener un porcentaje muy elevado de proteínas con repeticiones ni tampoco por tener un porcentaje bajo.
Vuelve al índice
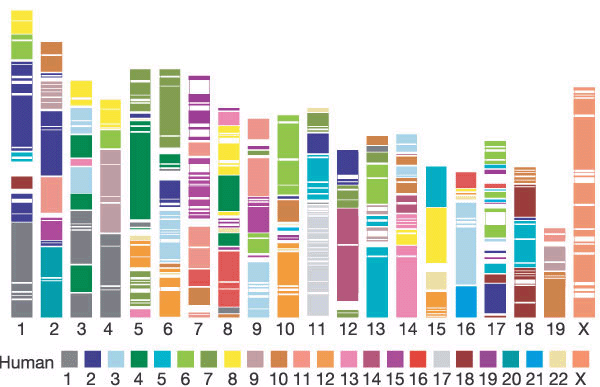
Sólo es posible comparar el cromosoma X entre ratón y humano, ya que es el único que se mantiene conservado entre estas dos especies. No nos sería relevante compararlos también con el cromosoma X de Drosophila melanogaster, ya que no está constituido por los mismos genes.
Fig.29. Mapa de sintenia entre los genomas de humano y ratón
Obtención de los datos para realizar la gráfica (egrep):
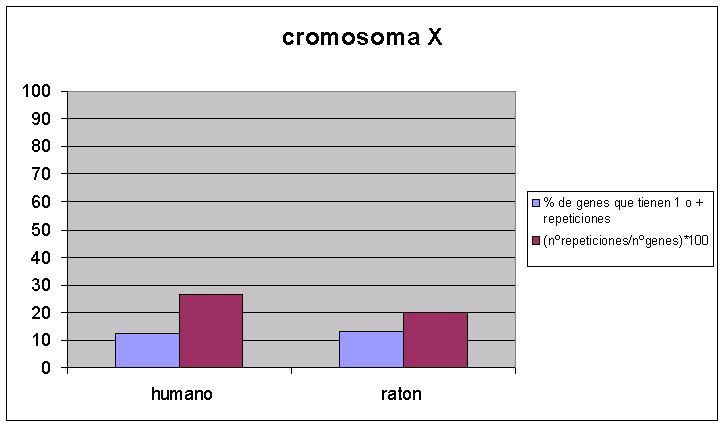
Para comparar estos cromosomas hicimos la gráfica del mismo modo que la de análisis de aglomeración de todo el genoma, pero únicamente en el cromosoma X. Es decir, por un lado hicimos nº de proteínas con repetición en el cromosoma X entre nº de proteínas totales del mismo y, por otro lado, nº de repeticiones en el X entre nº de proteínas totales del X.
Fig.30
Observaciones:
Resulta interesante ver que el número de proteínas que tienen repetición en los cromosomas X de las dos especies, es prácticamente el mismo. Sin embargo, existen más repeticiones en el cromosoma X de humano, lo cual indica que las proteínas de humano, al menos en el cromosoma X (aunque también hemos visto que pasa en el proteoma), tienden a acumular más de una repetición.
Clica aquí para volver al índice
Como hemos visto, las poliglutaminas son las repeticiones más largas, por eso decidimos consultar la bibliografía. La mayoría de artículos hacen referencia a que la expansión de las poliglutaminas causan diversos desórdenes neurológicos en humano (4) , varios cánceres, categorías de leucemias y abundancia de canales de calcio y potasio(5). Se conoce que las poliglutaminas se unen a otras proteínas posiblemente interfirienedo en su función, y esta podría ser la causa de estas patologías (4).
Como ejemplos conocidos tenemos (6)(7):
Todas estas enfermedades son autosómicas dominantes y los síntomas aparecen en edades avanzadas.
Viendo que existían tantas enfermedades relacionadas con la expansión de poliglutaminas, seleccionamos algunas proteínas que tenían una larga región de poliglutaminas y buscamos información sobre ellas para ver si también estaban asociadas a enfermedad.
Primero, ordenamos las proteínas que tenían repetición por aminoácido, seleccionamos aquellas en las que se repetía la glutamina y ordenamos por longitud de la repetición para escoger aquellas proteínas que tenían la repetición más larga. Con el identificador del gen, buscamos la proteína, su función y su posible asociación a enfermedad en swissprot y ensembl.(7)
Después de obtener estos datos concluímos que las proteínas con regiones largas de poliglutaminas son susceptibles a producir enfermedad, ya sea cáncer o enfermedad neurodegenerativa, por expansión o contracción de las mismas. Se sabe, que las regiones de poliglutaminas tienden a expanderse con las sucesivas generaciones. El principal mecanismo que puede explicar este hecho es el slippage: durante la replicación se darían errores en la hibridación de zonas que contienen repeticiones de trinucleótidos (codones), de manera que en la cadena sintetizada de nuevo se generarían copias extras del codón ( o también podría ser que perdiésemos copias).
Vuelve al índice
Tras ver en el gráfico de la distribución de los aminoácidos por cuartiles (fig.23), que la distribución de las polileucinas no estaba regida por el azar sino que la mayoría se acumulaban en el primer cuartil, intentamos dilucidar el porqué de este hecho.
Usando prosite(6) buscamos todas las proteínas que tenían 7 o más leucinas. Para nuestra sorpresa todas ellas eran precursores (quizás las repeticiones de leucinas sean reconocidas por alguna proteasa) y tenían alguna función relacionada con la interacción entre proteínas; entre ellas había cadherinas, receptores, semaforinas, receptores delta-notch, enzimas.... Para ver los resultados de la búsqueda clica aquí. Nos llamó la atención también el hecho de que muchas de estas proteínas actuaban durante el desarrollo embrionario (notch-delta, smothened, semaforinas, efrinas, precursores placentarios, coriogonadotropinas....), quizás debido a que en el desarrollo se producen muchas interacciones proteína-proteína para transducir señales. También había muchas relacionadas con el sistema inmunitario (interleukinas, proteínas del complemento, citokinas, proteínas estimuladoras del crecimiento de los macrófagos...) probablemente debido a los mismos motivos.
La mayoría de estas proteínas tenían un dominio de unión en el extremo N-terminal cosa que coincide con la localización de la mayoría de polileucinas. Por tanto, es muy posible que tengan algo que ver con la interacción entre proteínas.
Clica aquí para volver al índice
Muchos hechos interesantes se desprenden de las regiones homopoliméricas, aunque a decir verdad, no se ha profundizado en el tema. Dado que estas repeticiones se relacionan con muchas enfermedades, desarrollo embrionario e incluso respuesta inmunitaria resultaría conveniente que se llevasen a cabo más investigaciones; tanto a nivel descriptivo como sobretodo a nivel funcional, para llegar a comprender y asimilar el porqué y la importancia biológica de las mismas.
Clica aquí para volver al índice
MATERIAL Y MÉTODOS
Antes de nada hicimos un programa en perl para extraer todos los datos que necesitábamos. Dado un fichero con todo el proteoma de una especie en formato FASTA bajado desde Ensembl, el programa:
>ENSP00000000412 Gene:ENSG00000003056 Clone:AC006581 Contig:AC006581.16.1.172931 Chr:12 Basepair:8801021 Status:known L 6 11 277 1
>ENSP00000001178 Gene:ENSG00000004487 Clone:AL031428 Contig:AL031428.9.1.100951 Chr:1 Basepair:22916064 Status:known A 12 40 950 1
>ENSP00000001178 Gene:ENSG00000004487 Clone:AL031428 Contig:AL031428.9.1.100951 Chr:1 Basepair:22916064 Status:known P 5 185 950 1
>ENSP00000002165 Gene:ENSG00000001036 Clone:AL031320 Contig:AL031320.6.1.133574 Chr:6 Basepair:143439781 Status:known L 9 12 467 1
>ENSP00000004980 Gene:ENSG00000079387 Clone:AC004801 Contig:AC004801.1.1.193561 Chr:12 Basepair:48451793 Status:known S 5 102 643 1
>ENSP00000005011 Gene:ENSG00000004948 Clone:AC003078 Contig:AC003078.1.1.182433 Chr:7 Basepair:91589227 Status:known A 6 427 474 4
>ENSP00000005286 Gene:ENSG00000006118 Clone:AC004126 Contig:AC004126.1.1.136328 Chr:11 Basepair:63084863 Status:known E 9 577 774 2 y 3
>ENSP00000005340 Gene:ENSG00000004975 Clone:AC026954 Contig:AC026954.11.19017.39923 Chr:17 Basepair:7526566 Status:known P 6 685 736 4
RESULTADOS Y DISCUSIÓN
Proporción de repeticiones en los proteomas de los organismos estudiados
Análisis de la aglomeración de las repeticiones en las proteínas.
Número de repeticiones por aminoácido


Distribución de las repeticiones por cuartiles
Distribución de las repeticiones de cada aminoácido por cuartiles
Porcentaje de proteínas en cada cromosoma que contienen una o más repeticiones
Comparación cromosoma X entre humano y ratón
Poliglutaminas y enfermedad
Estudio de las polileucinas
CONCLUSIONES
REFERENCIAS