- Construcció del programa
- Anàlisi de resultats
- Referències i links
5. Ús de BLASTP amb tots els ORFs obtinguts per la identificació de noves proteïnes.
6. Anàlisi dels resultats obtinguts en l'anterior pas.
![]()
![]()
![]()
![]()
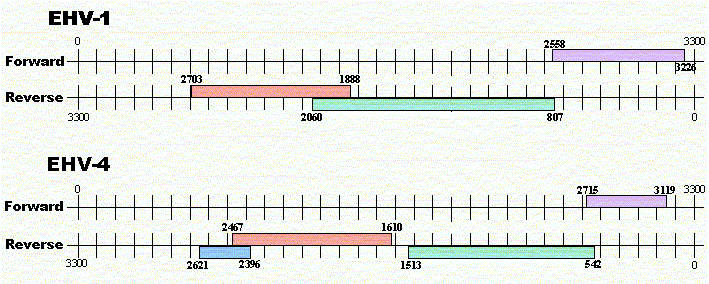
1. Introducció de les seqüències proteiques obtingudes en format FASTA a la base de dades VIDA per trobar homologia amb alguna família de proteïnes.
Els resultats obtinguts en aquest apartat mostren homologia de tres seqüències proteiques de les dues espècies d'herpesvirus (que molt probablement correspondran a gens ortòlegs) amb tres famílies de proteïnes. Les correspondències es mostren a continuació:
Homologia amb la família HPF40 de les següents seqüències:
1) EHV-1:
>Pauta de lectura: 1; Direccio de la cadena: forward; Inici de l'ORF: posicio 2558; Final de l'ORF: posicio 3226 MENKQCDHLTDWFSTTSDASESMDTTPPLPPPTPSVDPSYSGAAADEDLYSDISEGDLEYSDCD SASESDEDDDDCLIPSKEKAREVAASFGYTVIKTLTPGSEGRVMVATKDGQPEPVVLKIGQKGTT LIEAMMLRNVNHPSVIQMKDTLVSGAITCMVLPHYSSDLYTFLTKESRRIPIDQALIIEKQILEGLRYL HAQRIIHRDVKTENIFINSVDQVCI
2) EHV-4:
>Pauta de lectura: 2; Direccio de la cadena: forward; Inici de l'ORF: posicio 2715; Final de l'ORF: posicio 3119 MENKQYDHLLSDWLSGNISEASESMDTTPPLQLSVHPQNPSCGGAAANEDLYSDISDGDLECSD CDSASESDEDDDDGLMPPKEKAKEVAASFGFKVIKTLTPGSEGRVMVATKEGQPDQVVLKIGQK GTTLIEA
A l'hora de fer la cerca en la base de dades VIDA, l'ORF identificat com a membre de HPF40 a EHV-1 també presenta homologia amb la família HPF29. El fet que l'E-value sigui superior per HPF29 i que l'ORF equivalent en EHV-4 tan sols mostri homologia amb HPF40, fa pensar que aquesta última és la família a la qual pertanyen aquestes dues proteïnes obtingudes.
Segons la informació proporcionada per la base de dades VIDA, la família proteica HPF40 correspon a un grup de proteïna quinases (molt probablement serina-treonina quinases).
Homologia amb la família HPF139 de les següents seqüències:
3) EHV-1:
>Pauta de lectura: 0; Direccio de la cadena: reverse; Inici de l'ORF: posicio 1888; Final de l'ORF: posicio 2703 MNSDMMTAATAGTEVFRCALARRRNANPPHLVLAPTFAAAAAGGAANSSGEEAPRGERKHLFN PFGCMLGRSYFRRCREEMNEGYFAKVPTGYFPVAPSEVPCRVPVEGVVAGEVLSYSALPLPKIE KRFYKQLNDGTFVRLPFLYPEVYYEGEEEPADERYYIRADAADASSADPSTLPEEAFAKVPPAIA EGITNWQGPKRIPIPSERYVMKLGFEYQLHVTEDAFQEVNTSFMRLDLQSSPDPHPRGARQPRS RAHVSAENPEDTPVAV
4) EHV-4:
>Pauta de lectura: 1; Direccio de la cadena: reverse; Inici de l'ORF: posicio 1610; Final de l'ORF: posicio 2467 MSSDMLTAATAGTEVFRCNLARRRNANPPHLVLAPTFSSATVSESNAKDGASQEPRKYLFNPYK YMLGRPYFRRCREEMNEGYFAKVPEGYFPVGPSEMPGRVPVESKVDGEVLSFKALPPPKFEKR FYKQLDDGTFVRLPFLYPEEYYEGENQPSETLYYIRADTKDAYSVDPSDLLEEAFAEVPKWLEEE MSNWTGPKKLPIPSRRYVLKHGWEFQSNVTEDAFQEINTTFLRLDLQGEPKQHTRNAQQSSIGD QVLDAHPRDAQQPKNGNSVDKNSDNLVVAV
La funció d'aquesta família de proteïnes resta desconeguda segons la base de dades VIDA.
Homologia amb la família HPF66 de les següents seqüències:
5) EHV-1:
>Pauta de lectura: 2; Direccio de la cadena: reverse; Inici de l'ORF: posicio 807; Final de l'ORF: posicio 2060 MGVVLITVVTVVDRHKALPNSSIDVDGHLWEFLSRQCFVLASEPLGIPIVVRSADLYRFSSSLLTLP KACRPIVRTRGATAIALDRNGVVYHEDRMGVSIEWLSVLSGYNHLNSSLIINQPYHLWVLGAADLC KPVFDLIPGPKRMVYAEIADEFHKSWQPPFVCGKLFETIPWTTVEHNHPLKLRAAGGEDTVVGEC GFSKHSSNSLVRPPTVKRVIYAVVDPARLREIPAPGRPLPRRRPSEGGDARPEAALARSRARSV HGRRRDAAPPRGPAGARRPPGGRRDVDGTPTLGSVRPDIHTLKGRGLSPVPHLALWVEQWFL ALQKPRLYTHHRLALHNIPSILKGEKREDTFEDNRRDELRHDDSRHRRHRGLPLRARSPPQCQP APPRLGPHLRRSRGRGGRQLQRRGGAERRA
6) EHV-4:
>Pauta de lectura: 1; Direccio de la cadena: reverse; Inici de l'ORF: posicio 542; Final de l'ORF: posicio 1513 MGVVLITVVMVVDRHKALPDSSIDVDGKLWEFLGRLCFVLASEPLGIPIVVRSADLYKFSSSLLALP KACRPIVRTRGATAIALERNGVIYQEDRIGISIEWLSVLSGYNYLNSSIIINRPYHLWVLGAADLCRP VFNLIPGPKRIVYVEIEDEFNKSWQPSFVCGKLFETIPLTTVDYKHLLKQKVLPGQDHPESARSLLQ HKSSFVSPPPNFKRLIYAVVDPMRLQENLCPQITNRTKTKRRSKKTYNGLFCQESTASLNDKMCF TPQPSKGKNLQRVSTSMQANSTIPPSTLSPRAAARKPTEMTWKSRLLGGVFDRTARR
En aquest cas, segons la base de dades VIDA, la família HP66 també té una funció desconeguda.
![]()
![]()
![]()
![]()
2. Selecció de les seqüències amb homologia significativa segons la cerca anterior, i utilització del programa BLASTP per determinar els ortòlegs entre ambdues espècies d'herpesvirus.
Posterior a l'anàlisi amb la base de dades VIDA, escollim les sis seqüències que presenten homologia significativa amb alguna família proteica, numerades de l'1) al 6) en l'apartat anterior. Utilitzem el programa BLAST per tal de buscar els ortòlegs entre ambdós tipus d'herpesvirus.
Anàlisi de la família proteica HPF40:
- Resultats del BLAST realitzat amb la seqüència proteica 1):
L'homologia inicial amb dues possibles famílies proteiques (HPF40 i HPF29) ve donada pel fet que ambdós grups són proteïna quinases, i per tant tenen dominis molt semblants, com es pot observar en el següent gràfic obtingut en fer el BLAST:
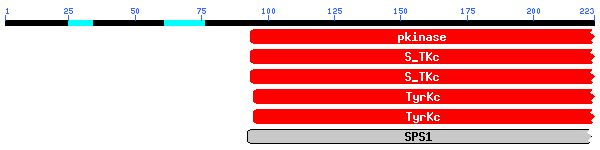
En el següent gràfic es pot veure que tots els aliniaments es donen com a mínim en la regió corresponent al domini conservat de les proteïnes quinases.
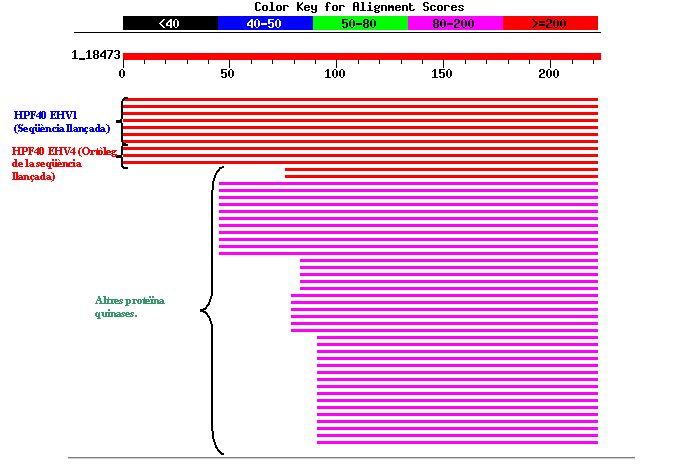
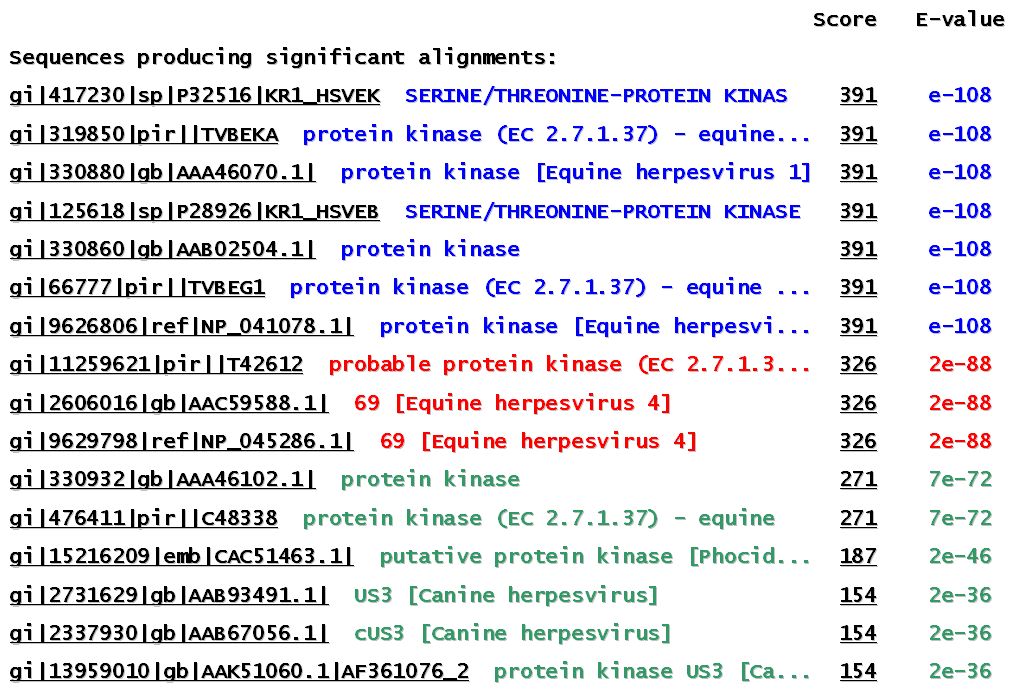
- Resultats del BLAST realitzat amb la seqüència proteica 2):
Els resultats són molt semblants als obtinguts en llançar la seqüència ortòloga 1).
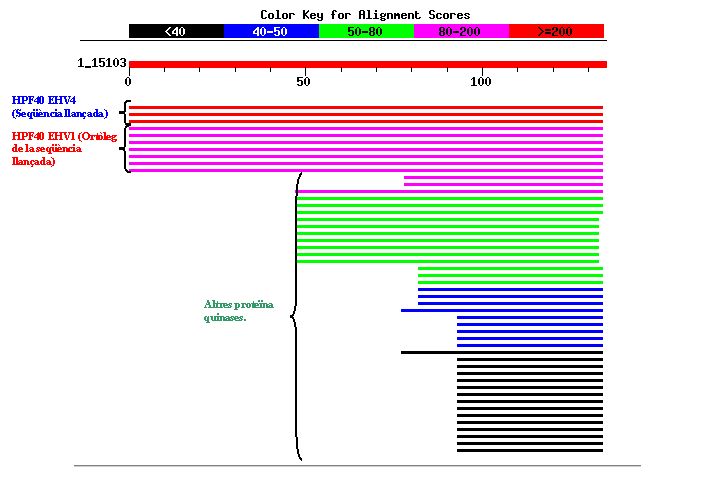
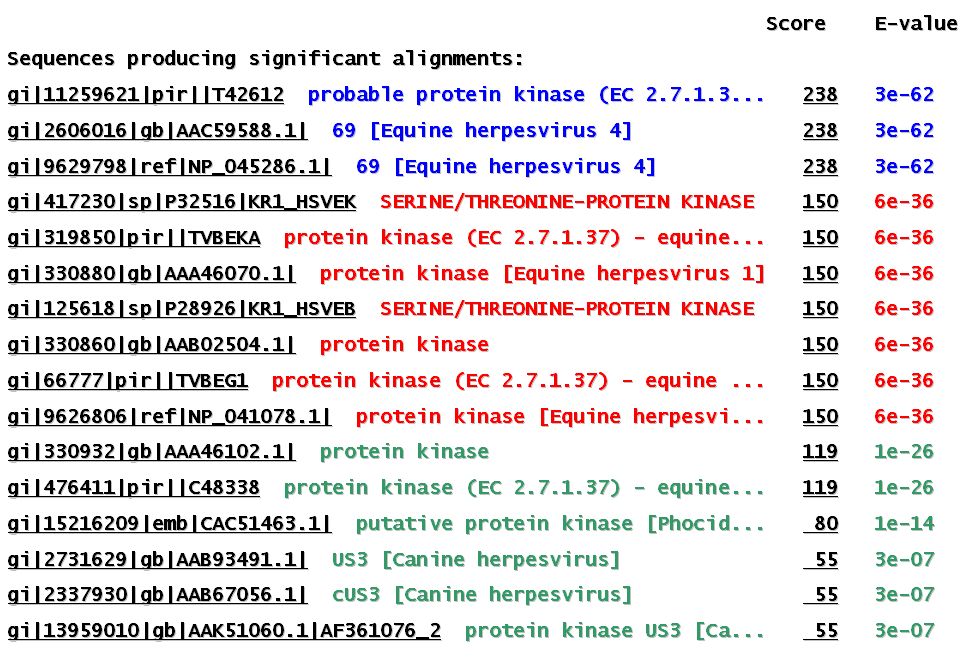
Com es pot observar en aquesta imatge, la màxima homologia es troba amb la pròpia seqüència llançada i amb la què és ortòloga en l'altre tipus d'herpesvirus. Amb la resta de proteïnes s'observa homologia sols en la regió corresponent al domini conservat de les proteïna quinases. També podem veure que l'Score per la família proteica HPF40 és superior en EHV-1 que en EHV-4. Això es deu a que la seqüència aminoacídica corresponent a l'ORF de EHV-1 és més llarga i en conseqüència l'Score es veu augmentat i l'E-value disminuït.
Anàlisi de la família proteica HPF139:
- Resultats del BLAST realitzat amb la seqüència proteica 3):
En fer córrer el programa BLAST no s'han trobat dominis conservats per aquesta família proteica.
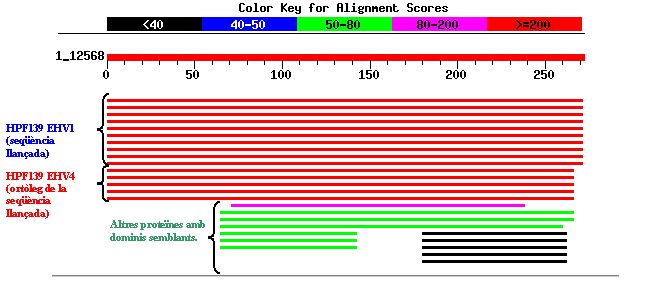

En aquesta imatge es pot observar com la màxima homologia es mostra amb les seqüències proteiques ortòlogues en l'altre grup d'herpesvirus. També es pot veure com altres proteïnes d'herpesvirus d'altres espècies presenten dominis homòlegs amb la proteïna analitzada. Cal destacar que en aquest cas existeixen algunes proteïnes G humanes (representades en color negre) que mostren certa homologia amb una regió de la proteïna 3), tot i que els E-values corresponents no són gaire significatius.
Tot i que la base de dades VIDA indica que la funció d'aquesta família de proteïnes és desconeguda, si entrem a Interpro , la proteïna és classificada com una possible quinasa de carbohidrats ("It has been shown that the following carbohidrates and purine kinases are evolutionary related and can be grouped into a single family, which is known as the pfkB family") . Com veurem a continuació, aquesta classificació no està ben definida, ja que l'ortòleg en EHV-4 no es troba inclòs dins la família pfkB.
- Resultats del BLAST realitzat amb la seqüència proteica 4):
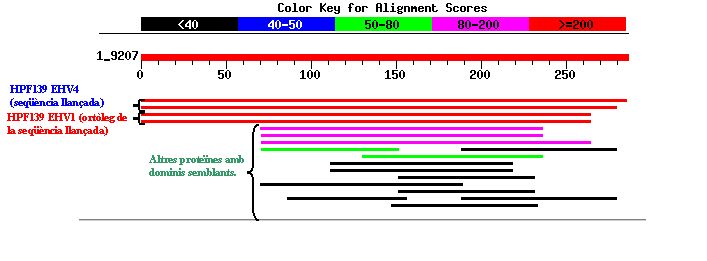

Igual que en el cas anterior, s'observa la màxima homologia amb la pròpia proteïna analitzada i amb la seqüència ortòloga en EHV-1. Podem veure també que altres proteïnes d'herpesvirus d'altres espècies tenen dominis homòlegs, però no existeix la semblança amb les proteïnes G humanes que apareixia en llançar la seqüència corresponent a HPF139 en EHV-1.
Com ja hem citat anteriorment, la base de dades VIDA no dóna funció coneguda per aquesta família proteica. Si fem la cerca a Interpro, tampoc ens integra la proteïna dins de cap grup. Això es contradiu amb la informació que aquesta base de dades ens havia donat en el cas d'EHV-1. Aquest fet pot ser degut a una possible taxa mutacional superior en EHV-4, que hauria provocat una major divergència amb la família pfkB.
Anàlisi de la família proteica HPF66:
- Resultats del BLAST realitzat amb la seqüència proteica 5):
L'estudi dels dominis proteics comuns en el programa BLAST per aquesta família, mostra l'existència d'un domini conservat (el mateix en EHV-1 i EHV-4): US2.
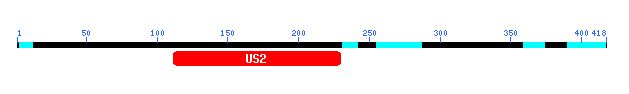
Com es pot observar en el següent gràfic, tots els aliniaments es donen com a mínim en la regió corresponent al domini conservat US2.
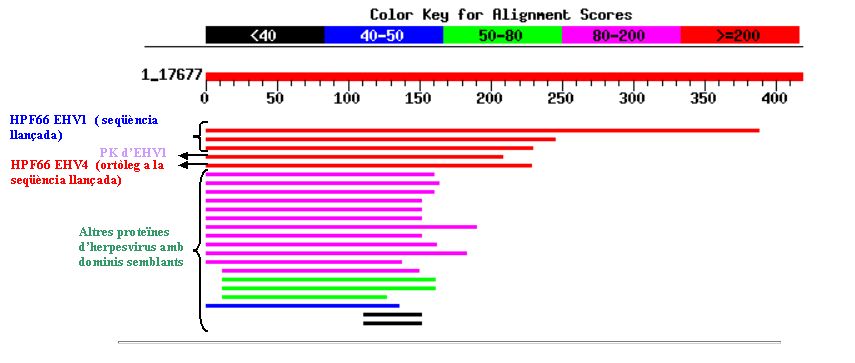
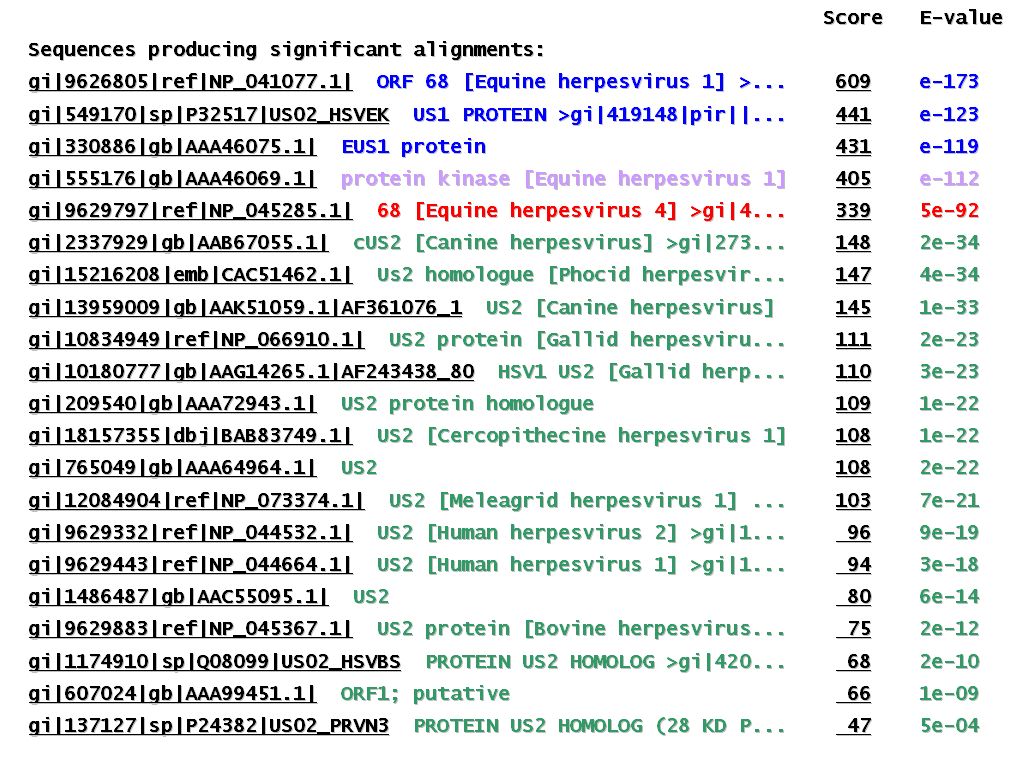
Com ens mostra la imatge, l'Score més gran l'obtenim amb la pròpia seqüència aminoacídica llançada. El que més sorprèn és que l'ortòleg en EHV-4 no apareix entre els millors aliniaments. El fet que l'homologia entre les seqüències ortòlogues no s'apropi al 100% es deu possiblement a la diferent taxa mutacional entre ambdós genomes. En el següent apartat veurem que les prote´nes ortòlogues dels dos tipus d'herpesvirus sí es troben en la zona de màxima homologia.
En l'anterior imatge també es pot observar una certa homologia amb un domini d'una proteïna quinasa d'EHV-1 i amb altres dominis de diferents proteïnes d'herpesvirus de diverses espècies.
- Resultats del BLAST realitzat amb la seqüència proteica 6):
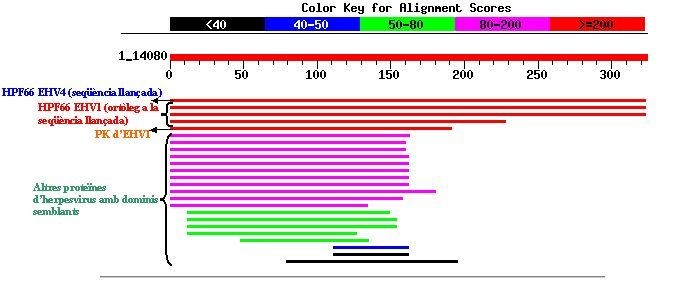

En aquest cas, en la zona de màxima homologia trobem tant la pròpia seqüència aminoacídica llançada com la proteïna ortòloga en EHV-1. El fet que ara aparegui la seqüència ortòloga entre els millors aliniaments, a diferència del cas anterior, pot venir donat perquè la prote´na d'EHV-1 és més llarga, i això influeix en l'Score obtingut.
També en aquest cas apareix homologia amb la mateixa proteïna quinasa d'EHV-1 citada en l'apartat anterior, així com amb altres dominis de diferents proteïnes d'herpesvirus d'altres espècies.
![]()
![]()
![]()
![]()
3. Repetició dels passos 1 i 2 amb els resultats obtinguts sense limitar l'inici de l'ORF a metionina.
En repetir el pas 1 amb les proteïnes obtingudes sense limitar l'inici de l'ORF a metionina, només trobem dues noves seqüències que presentin homologia amb una altra família proteica: HPF281 . Totes dues pertanyen a EHV-4, i es mostren a continuació:
>Pauta de lectura: 0; Direccio de la cadena: forward; Inici de l'ORF: posicio 172; Final de l'ORF: posicio 423 TKAFKYLPTSPPPHLPTSPPPHLPTSPPPHLPTSPPPHLPTSPPPHLPTSPPPHLPTSDRGWETSY PGHRTNAQRLGFSTMRFY
>Pauta de lectura: 1; Direccio de la cadena: forward; Inici de l'ORF: posicio 146; Final de l'ORF: posicio 412 TFKLSILHKPKRSSTSPPPHLPTSPPPHLPTSPPPHLPTSPPPHLPTSPPPHLPTSPPPHLPTSPPP IGGGKQATRAIEQTRRGWGSLL
La informació que la base de dades VIDA proporciona per la família proteica HPF281 tan sols inclou una espècie de virus: Pseudorabies virus . Si s'intenta buscar a les bases de dades de NCBI alguna proteïna de Pseudorabies virus pertanyent a la família de proteïnes citada, es pot observar que la seva seqüència aminoacídica és molt llarga, al contrari que la trobada a herpesvirus. És probable que en aquest cas l'homologia s'hagi trobat per la gran presència de repeticions de l'aminoàcid P (prolina) tant en les seqüències d'herpesvirus analitzades com en les de certes proteïnes d'HPF281.
Pel que fa al pas 2, la utilització del programa BLAST per les seqüències aminoacídiques trobades sense limitar l'inici de l'ORF a metionina, proporciona uns resultats molt semblants als obtinguts quan l'inici de l'ORF sí és limitat. Les principals diferències es troben en la zona de màxima homologia; en el cas que l'inici de l'ORF no estigui limitat a metionina, s'obté un menor nombre de seqüències amb un Score superior a 200. Això pot ser degut al fet que si l'inici de l'ORF no està limitat, les seqüències que s'obtenen presenten una major llargada, de manera que és més complicat trobar proteïnes amb gran similaritat. Cal dir, però, que les proteïnes homòlogues en ambdós tipus d'herpesvirus es distribueixen de la mateixa forma en fer córrer el programa BLAST, tant si es limita l'inici de l'ORF a metionina com si no.
![]()
![]()
![]()
![]()
4. Aplicació del programa BLAST PAIRWISE per tal de cercar regions conservades entre els fragments EHV-1 i EHV-4.
Aquest pas de l'anàlisi de dades consisteix en la cerca de regions conservades entre els fragments d'EHV-1 i EHV-4 lliures de gens (que van aproximadament des del nucleòtid 0 fins el 800 en la cadena forward, i des del nucleòtid 2500 fins el 3300 en la cadena reverse). Aquesta finalitat requereix l'ús del programa BLAST PAIRWISE (BLAST per dues seqüències), per tal d'anar comparant totes les possibles combinacions de dues proteïnes (una de cada tipus d'herpesvirus) de la zona citada. Els resultats mostren una certa homologia entre les següents zones:
- Regió que va de la posició 2784 a la 3227 de la cadena reverse de l'EHV-1.
- Regió que va de la posició 146 a la 412 de la cadena forward de l'EHV-4.
Cal dir, però, que l'anàlisi d'aquestes àrees mostra que l'homologia trobada existeix bàsicament perquè totes dues seqüències contenen moltes repeticions curtes dels motius SPP i LP . Probablement aquest aliniament (del qual mostrem un fragment en el gràfic a continuació) no sigui gens significatiu.

![]()
![]()
![]()
![]()
5. Ús de BLASTP amb tots els ORFs obtinguts per la identificació de noves proteïnes.
Per tal d'identificar noves proteïnes, aquest apartat requereix la utilització novament del programa BLASTP , que s'ha de fer córrer amb tots els ORFs obtinguts amb el programa CERCADORFS quan es limita l'inici a metionina i la llargada a 60 aminoàcids (escollim aquesta opció ja que al llarg del treball hem pogut observar que la majoria de proteïnes comencen per metionina). Per la cerca de noves proteïnes és interessant desactivar l'opció del filtre de baixa complexitat , ja que per defecte aquest filtre és actiu per tal de disminuir el nombre de falsos positius. És necessari doncs, desactivar el citat filtre si no es vol ometre qualsevol possible resultat en la cerca de noves proteïnes.
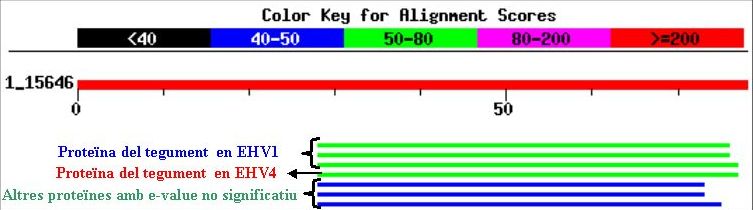
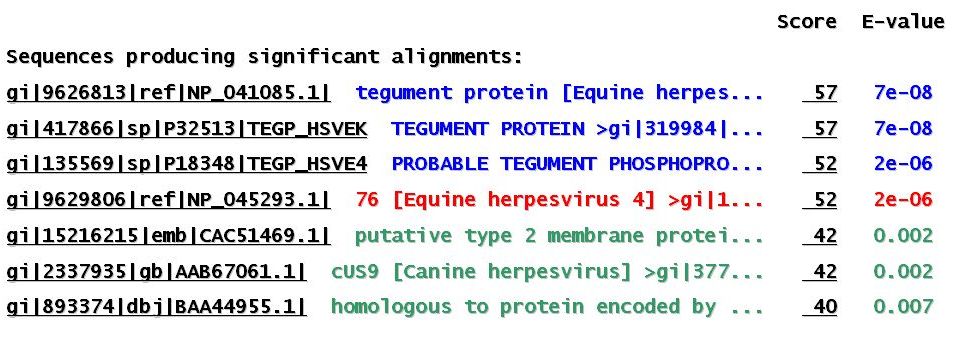
Quan es fa córrer el programa BLASTP amb les opcions citades per les seqüències proteiques d'EHV-1, no es troba cap resultat significatiu d'homologia (els E-values que surten són molt dolents). En canvi, si les seqüències que es llancen són les corresponents a EHV-4 s'obté un hit del fragment que va des del nucleòtid 2388 fins el 2621 en la cadena reverse amb una proteïna del tegument (seqüència a l'entrada AAB02511 ). Considerem l'homologia trobada com a significativa, ja que l'E-value és força baix.
La seqüència proteica per la qual s'ha trobat l'homologia citada és la següent:
>Pauta de lectura: 2; Direccio de la cadena: reverse; Inici de l'ORF: posicio 2388; Final de l'ORF: posicio 2621 MHIPEMPNSQKMEIRLIKTQTTWSLLFNASLFIQRVGRRQARRHRRRRVALTVAGVVLVAVLCAIS GIVGAFLARVFQ
Els resultats de córrer el programa BLASTP amb aquesta seqüència es mostren a continuació:
![]()
![]()
![]()
![]()
6. Anàlisi dels resultats obtinguts en l'anterior pas.
L'estudi de la nova proteïna trobada a la base de dades VIDA indica que correspon a una proteïna del tegument de funció desconeguda, i es troba classificada dins la família proteica HPF119 . El motiu que en fer córrer el programa BLASTP amb les seqüències proteiques d'EHV-1 no doni homologia amb aquesta nova proteïna fa pensar que probablement el gen que codifica per ella no es troba en el fragment genòmic que hem utilitzat per trobar i analitzar els ORFs. Per aquest motiu es pot fer un estudi de la situació del citat gen en el genoma complet d'EHV-1 . Per tal d'aconseguir aquesta finalitat cal anar a la base de dades GenBank (NCBI) i, mitjançant l'identificador de la proteïna del tegument, es pot observar que la regió que codifica per aquesta es troba entre les posicions 136782 i 137441 del genoma complet. Aquesta àrea queda fora del fragment que hem estudiat, que va des del nucleòtid 123961 fins el 127080.
Resum dels gens trobats en els fragments genòmics analitzats: